版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
血液肿瘤是一类具有高度异质性的疾病,其诊疗需要结合形态学、免疫学、遗传学和分子生物学进行综合分析。二代测序(Next-generation sequencing, NGS)作为新的分子生物学技术,具有通量高、灵敏度高、成本低等优势,是探索血液肿瘤的分子发病机制并指导临床诊疗的重要手段。为推动NGS在血液肿瘤诊疗中的应用,提高诊疗水平,中国抗癌协会血液肿瘤专业委员会、中华医学会血液学分会、中华医学会病理学分会组织国内相关的血液、病理和检验专家,结合国外权威资料和已积累的大样本数据,制订了NGS在血液肿瘤中应用的中国专家共识。
血液肿瘤中常见的分子生物学异常主要包括基因突变、融合基因及基因异常表达。目前NGS在基因突变的检测方面应用最为广泛和成熟,本共识仅涉及基因突变的检测。
基因突变的检测在急性髓系白血病(AML)伴重现性遗传学异常、遗传易感性髓系肿瘤、骨髓增殖性肿瘤(MPN)、骨髓增生异常综合征伴环形铁粒幼红细胞(MDS-RS)、毛细胞白血病(HCL)和淋巴浆细胞淋巴瘤/华氏巨球蛋白血症(LPL/WM)的诊断中具有关键性的作用,对于其他血液肿瘤则起到辅助诊断的作用[1,2,3,4,5,6,7,8]。
基因突变是各类血液肿瘤预后判断的重要依据,目前NCCN指南已提出了基于基因突变的AML预后分层体系[1]。此外,MDS、MPN、MDS/MPN、急性淋巴细胞白血病(ALL)、慢性淋巴细胞白血病/小淋巴细胞淋巴瘤(CLL/SLL)、LPL/WM、大颗粒淋巴细胞白血病(LGLL)中,已经证实了一些具有明确预后意义的突变基因[2,3,4,5,6,7,8]。其他血液肿瘤中突变基因的预后意义尚有待于进一步研究。
一方面,基因突变检测可提供分子治疗靶点,对应靶向药物进行治疗,目前已有基于突变基因的靶向药物应用于临床或处于临床试验阶段,其中FLT3、IDH1/2、BRAF及JAK-STAT信号通路相关的突变基因已有靶向药物上市[1,4,5];另一方面,基因突变可以导致对某些药物的敏感或者耐受,及时检测有助于治疗方案的调整。例如,TP53突变的CLL/SLL患者对常规化疗反应差,MYD88和CXCR4基因突变可影响伊布替尼在LPL/WM中的治疗效果,ABL1激酶区突变是慢性髓性白血病(CML)和Ph+ ALL酪氨酸激酶抑制剂(TKI)耐受的主要机制之一[4,7,8]。
基因突变是MRD监测的分子标志物之一[9]。虽然实时定量PCR方法和流式细胞学是目前主流的MRD监测方法,但是由于NGS灵敏度随测序深度加深可进一步提高,在MRD监测方面具有更大的优势。
血液肿瘤在发展过程中会伴随动态的克隆演变,或是基因突变负荷的改变,或是新的突变基因的出现,及时监测基因改变,有助于了解疾病进展并调整治疗方案[10]。
血液肿瘤相关检测基因由临床血液肿瘤专家和实验室专家共同制定,主要依据中国抗癌协会、中华医学会、WHO、NCCN等机构发布的血液系统疾病诊疗指南或者专家共识。对于其他权威文献报道的重要基因,可在验证之后纳入基因列表。血液肿瘤易感的胚系突变基因,根据检测需求可以纳入,例如CEBPA、DDX41、RUNX1、ETV6、GATA2等基因除了可发生体细胞突变外,发生胚系突变可导致髓系肿瘤的易感[2,4]。检测基因的数量应在满足临床需求的基础上,综合疾病类型、实验技术、样本数量以及检测成本达到最优化的设计[11,12]。随着基础及临床研究的不断深入,检测的基因种类亦需随之更新。
被检测的基因所覆盖区域可参考临床诊疗指南、权威数据库、文献及实验室自建数据库等。首先应纳入基因热点突变区域或重要结构域的编码区域,例如NPM1基因c.860_863为热点突变区域,NOTCH1基因突变最多见于HD结构域及ΔPEST结构域[13];无明确热点突变区域或突变位点分布比较分散的基因,应检测全部外显子,例如TP53、RUNX1等;剪接位点突变有可能导致外显子跳跃,影响蛋白功能,建议根据数据库记录和文献报道将剪接位点±5 bp的区域纳入检测范围;对于存在非翻译区(UTR)突变的基因,例如BCL6,则应纳入相应区域检测[14]。需要注意的是,对于不能覆盖的热点突变类型或重要区域可通过其他检测方法进行补充,并在检测报告中明确说明。
疑诊为血液肿瘤的患者,初诊时应留存新鲜骨髓、外周血或组织样本,以备进行NGS检测,未留存者可使用初诊时的骨髓涂片进行检测。骨髓采集量以1~3 ml为宜,外周血采集量3~5 ml为宜,若白细胞计数偏高或偏低,应适当调整采集量,使单个核细胞总数达到1×107以上。抗凝剂首选EDTA抗凝剂或枸橼酸钠抗凝剂,禁止使用肝素抗凝,推荐24 h内4 ℃冷藏运送。福尔马林固定后的组织标本需选择肿瘤成分,切片厚度8~10 µm,5~8片为宜,常温运输。已染色标本不推荐进行NGS检测。条件允许的病例,可从同一患者采取正常组织作为胚系突变对照。
DNA质量是NGS检测成功的关键因素,同一患者的不同病灶组织、不同组织切片应分别单独进行DNA提取,并对DNA质量进行评估,包括浓度、纯度和完整性分析。由于FFPE组织标本以及骨髓涂片所提取的DNA易片段化,且因保存环境及保存时间不同,导致DNA片段化的程度参差不齐,因此各实验室应具有相应的方法检测DNA的完整性或降解程度,并设立明确的合格样本标准[11]。
文库制备用于目标区域的富集,主要有杂交捕获法和扩增子建库法,无论采用何种方法制备文库,均需使用已验证过的建库试剂[15]。多标本同时测序应有相应的分子标签用于区分不同的测序标本,明确不同的检测标本和分子标签的一一对应关系;必要时,可对加标签的方法及试剂进行说明[16]。文库浓度过高会导致多克隆数据的产生,降低有效测序数据量;过低会导致整体测序数据的减少,因此在测序之前推荐使用实时定量PCR或其他方法对文库进行定量,将文库浓度调整至合适的水平。每个检测项目应设定其文库质量的要求,并设立明确的合格文库标准。
目前测序主要有电位检测和光学检测两种基本原理,检测DNA合成过程中释放的氢离子或荧光信号。为保证测序质量、检测区域覆盖深度及报告时效性,需要根据样本数量和质量选取适当的测序平台和芯片。
由测序平台产生的原始数据通常会存在碱基召回错误、插入删除错误、低质量读段(reads)及接头污染等问题,会在一定程度上影响下游的分析过程。因此,在对测序结果进行具体的生物信息学分析之前,要先对下机数据进行质量评估,制定有效的质量控制标准,包括碱基的质量Q20、目标区域平均测序深度、均一性等评价参数。血液肿瘤的基因突变检测,测序深度应≥500×,平均测序覆盖度、均一性以及在靶率均≥90%。
低质量读段会影响下游的序列处理和分析,因此需要对测序数据进行过滤处理。数据过滤所针对的读段主要有四种:测序质量低的读段(low quality reads),重复读段(duplicate reads),含有核苷酸的插入、删除和替换等错误的读段(insertion/deletion/mismatch)和带有人工污染的读段(adaptor等)。常用的数据过滤软件有Trimmomatic、Fastx_toolkit、NGS QC Toolkit等。
当读段经过质控与过滤等步骤达到一定的质量标准之后,需要将其比对到参考基因组上。目前普遍参考的人类基因组参考序列为Human hg19。不同的比对方法根据不同的比对策略设计了不同的算法,常用的比对程序软件有MAQ、BWA、Bowtie/Bowtie2、SOAP/SOAP2、mrFAST和Stampy等,实验室应当选取合适的比对软件,建立完善的实验室比对流程。
突变位点识别常借助于Samtools、GATK等识别工具。对突变位点的注释内容包括功能信息、频率信息、软件预测结果信息以及疾病数据库信息,常用的包括Ensembl、RefSeq、GENCODE、dbSNP、1000genomes、ESP、ExAC、COSMIC、HGMD、Clinvar、polyphen和SIFT等。
筛选与鉴别疾病相关突变位点需要经过严格的筛选流程,至少需要排除低质量变异、未有明确意义的非编码区变异、同义单核苷酸变异(SNV)及已知健康人群中的基因多态性位点。
报告中除患者信息、标本信息、送检日期、报告署名及日期等一般资料外,还应包括:检测基因列表、检测区域及突变类型;使用的检测平台、目标区域的富集方法、平均测序深度、数据分析软件名称及版本号;对相关专业术语进行解释说明;本实验室报告突变位点的规则、检测方法的局限性、检测灵敏度和准确度等;注明是否通过其他方法补充了NGS未覆盖或覆盖不佳的检测区域等[11,16]。
突变位点信息应该包括基因名称、突变的物理坐标、cDNA的转录本号、外显子位置、符合人类基因组变异协会(HGVS)书写规范的突变类型、氨基酸突变类型、变异等位基因频率以及该突变位点的测序深度等[17]。参考序列推荐使用美国国立生物技术信息中心(National Center of Biotechnology Information)收录的参考序列。当使用各基因编码DNA参考序列描述突变时,推荐最常使用的经典转录本(canonical sequence)[18]。
不推荐将良性或可能良性突变在报告中报道。建议突变位点根据临床意义的明确性进行分级报告:
①一级变异:具有明确临床意义的突变,包括:国家食品药品监督管理总局(CFDA)、美国食品药品管理局(FDA)等机构批准的用药治疗靶点;血液肿瘤诊疗指南或专家共识中有明确诊断、治疗、预后意义的突变;尚未进入诊疗指南或专家共识,但有权威文献或大规模报道在血液肿瘤中具有诊疗意义的突变。
②二级变异:具有潜在临床意义的突变,包括:基于多个小规模研究报道在血液肿瘤中具有诊断、治疗或预后意义,但尚未达成共识的突变;新发现的疾病相关基因重要结构域的体细胞突变。
③三级变异:临床意义未明的突变,对于尚有争议的突变位点,实验室必须制定相关规则,可以发现突变即报告,并附上说明和意义;也可以不报告或只报告小部分突变结果,并附上说明、参考文献及数据库。
突变位点的临床意义解读要平实客观、清晰易懂。推荐写明突变位点的人群突变频率、蛋白功能危害性预测和疾病数据库的记录,根据疾病诊疗指南、专家共识和既往研究说明突变位点在临床诊断、治疗和预后评估中的意义,注明其相关药物及正在开展的状态信息,并附上数据库和参考文献来源。对于胚系突变,除了疾病诊疗指南及重要参考文献外,推荐参考OMIM、HGMD和ACMG等数据库或学术机构中遗传疾病变异分类指导注释临床意义,并附上数据库和参考文献来源。阴性结果并不能完全排除患者不存在基因突变,可能是由于检出灵敏度或检测区域局限性所致,报告中应当以医师可以理解的方式对此予以说明[19]。
NGS检测实验室的总体设计与要求应参考《分子病理诊断实验室建设指南(试行)》、《医疗机构临床基因扩增检验实验室工作导则》等行业文献和要求。检测人员、生物信息分析人员、报告分析人员和提供咨询人员应具备相应专业背景且经过相应培训取得上岗资质[20]。实验室区域设置和环境需要满足实验环节和仪器要求。NGS检测试剂及测序平台应首选CFDA认可的产品。涉及实验室自配试剂、探针等,应该有严格的试剂制备标准操作规程(SOP),并经过临床实验室自建项目(LDT)验证合格后方可使用[11,18,21,22]。
NGS实验室应建立质量管理体系,对于标本采集和处理、实验操作、生物信息分析和报告分析各个环节均需要建立SOP文件,实验操作程序和生物信息分析须经过性能验证或确认[15]。
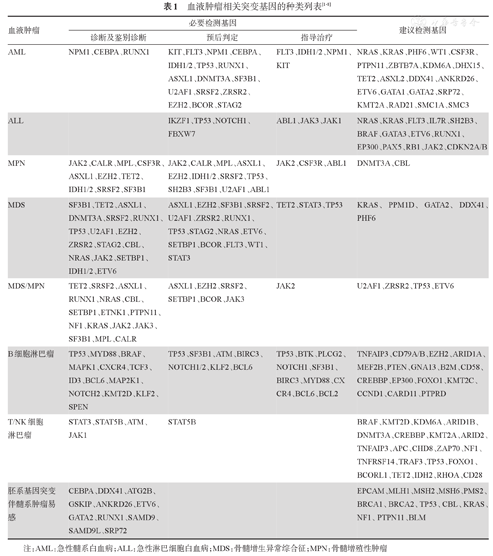
| 血液肿瘤 | 必要检测基因 | 建议检测基因 | ||
|---|---|---|---|---|
| 诊断及鉴别诊断 | 预后判定 | 指导治疗 | ||
| AML | NPM1、CEBPA、RUNX1 | KIT、FLT3、NPM1、CEBPA、IDH1/2、TP53、RUNX1、ASXL1、DNMT3A、SF3B1、U2AF1、SRSF2、ZRSR2、EZH2、BCOR、STAG2 | FLT3、IDH1/2、NPM1、KIT | NRAS、KRAS、PHF6、WT1、CSF3R、PTPN11、ZBTB7A、KDM6A、DHX15、TET2、ASXL2、DDX41、ANKRD26、ETV6、GATA1、GATA2、SRP72、KMT2A、RAD21、SMC1A、SMC3 |
| ALL | IKZF1、TP53、NOTCH1、FBXW7 | ABL1、JAK3、JAK1 | NRAS、KRAS、FLT3、IL7R、SH2B3、BRAF、GATA3、ETV6、RUNX1、EP300、PAX5、RB1、JAK2、CDKN2A/B | |
| MPN | JAK2、CALR、MPL、CSF3R、ASXL1、EZH2、TET2、IDH1/2、SRSF2、SF3B1 | JAK2、CALR、MPL、ASXL1、EZH2、IDH1/2、SRSF2、TP53、SH2B3、SF3B1、U2AF1、ABL1 | JAK2、CSF3R、ABL1 | DNMT3A、CBL |
| MDS | SF3B1、TET2、ASXL1、DNMT3A、SRSF2、RUNX1、TP53、U2AF1、EZH2、ZRSR2、STAG2、CBL、NRAS、JAK2、SETBP1、IDH1/2、ETV6 | ASXL1、EZH2、SF3B1、SRSF2、U2AF1、ZRSR2、RUNX1、TP53、STAG2、NRAS、ETV6、SETBP1、BCOR、FLT3、WT1、STAT3 | TET2、STAT3、TP53 | KRAS、PPM1D、GATA2、DDX41、PHF6 |
| MDS/MPN | TET2、SRSF2、ASXL1、RUNX1、NRAS、CBL、SETBP1、ETNK1、PTPN11、NF1、KRAS、JAK2、JAK3、SF3B1、MPL、CALR | ASXL1、EZH2、SRSF2、SETBP1、BCOR、JAK3 | JAK2 | U2AF1、ZRSR2、TP53、ETV6 |
| B细胞淋巴瘤 | TP53、MYD88、BRAF、MAPK1、CXCR4、TCF3、ID3、BCL6、MAP2K1、NOTCH2、KMT2D、KLF2、SPEN | TP53、SF3B1、ATM、BIRC3、NOTCH1/2、KLF2、BCL6 | TP53、BTK、PLCG2、NOTCH1、SF3B1、BIRC3、MYD88、CXCR4、BCL6、BCL2 | TNFAIP3、CD79A/B、EZH2、ARID1A、MEF2B、PTEN、GNA13、B2M、CD58、CREBBP、EP300、FOXO1、KMT2C、CCND1、CARD11、PTPRD |
| T/NK细胞淋巴瘤 | STAT3、STAT5B、ATM、JAK1 | STAT5B | BRAF、KMT2D、KDM6A、ARID1B、DNMT3A、CREBBP、KMT2A、ARID2、TNFAIP3、APC、CHD8、ZAP70、NF1、TNFRSF14、TRAF3、TP53、FOXO1、BCORL1、TET2、IDH2、RHOA、CD28 | |
| 胚系基因突变伴髓系肿瘤易感 | CEBPA、DDX41、ATG2B、GSKIP、ANKRD26、ETV6、GATA2、RUNX1、SAMD9、SAMD9L、SRP72 | EPCAM、MLH1、MSH2、MSH6、PMS2、BRCA1、BRCA2、TP53、CBL、KRAS、NF1、PTPN11、BLM | ||
注:AML:急性髓系白血病;ALL:急性淋巴细胞白血病;MDS:骨髓增生异常综合征;MPN:骨髓增殖性肿瘤
实验环节需要设置阴/阳性对照[11,18]。阳性对照一般采用包含已知突变信息的混合样本作为质控材料,并且其中应当包含最低检测限的突变位点,实验时同时进行检测,以确保其检出能力。阴性对照一般采用明确无突变或无核酸的样本作为质控材料,实验时同时进行检测,以确保实验过程中无污染及无非特异性。
实验室应定期参加相应检测项目的室间质量评价或能力验证;若无法实现,应通过与其他实验室(如使用相同检测系统的实验室)比对的方式,判断检验结果的可接受性。
样本质量问题、检测过程的不确定因素以及数据分析可能存在的漏洞,都可能导致检测结果中存在假阳性或者假阴性。实验室需要确定适宜的cut off值,在cut off值以上的结果可以采用自动化结果,但若出现比较疑似单碱基插入或缺失的未知突变,建议进行人工核对,并采用Sanger测序、ARMS-PCR、数字PCR等方法进行验证。而对于cut off值以下的有意义位点必须进行人工核对,同时采用ARMS-PCR、数字PCR或二次建库/测序方法进行验证[18]。很多实验室的NGS测序平台都可能会出现一定数量的"实验室特异性"的突变,此类突变是该检测系统特有的假阳性突变,应在报告时予以剔除。
随着分子生物学在血液肿瘤发病机制和临床诊疗研究的不断深入,NGS在血液肿瘤中的应用将会越来越广泛和高效。本共识阐述了NGS在血液肿瘤中应用的基本原则和总体流程,并对血液肿瘤的临床诊疗提供有效帮助。NGS在未来的技术、分析内容和结果解读方面会不断进步和完善,在血液肿瘤的实践应用过程中也会带来各种挑战。
参与共识讨论的专家:哈尔滨血液病肿瘤研究所(马军、邱林);吉林大学第一医院(白鸥);中国医科大学附属盛京医院(刘卓刚、张继红);北京大学第一医院(任汉云);解放军总医院(高春记);北京协和医院(陈杰、梁智勇、周道斌);中国医学科学院阜外医院(周洲);中国食品药品检定研究院(黄颖);中国合格评定国家认可委员会(周亚莉);卫生部临床检验中心(彭明婷);北京大学肿瘤医院(朱军);北京大学人民医院(秦亚溱);北京大学第三医院(克晓燕);山西省肿瘤医院(苏丽萍);山西医科大学第二医院(杨林花、王晨);河北燕达陆道培医院(刘红星);河北医科大学第二医院(罗建民);空军军医大学西京医院(高广勋、王哲);山东省立医院(王欣);山东大学齐鲁医院(侯明、纪春岩);上海交通大学医学院附属瑞金医院(赵维莅);海军军医大学附属长海医院(杨建民、何妙侠);上海交通大学医学院附属仁济医院(侯健);复旦大学附属肿瘤医院(杜祥);复旦大学附属中山医院(刘澎、侯英勇);江苏省人民医院(乔纯、李建勇);苏州大学附属第一医院(吴德沛、陈苏宁);江苏省临床检验中心(许斌);南京大学医学院附属鼓楼医院(樊祥山);徐州医科大学附属医院(李振宇、桑威);安徽省立医院(孙自敏);安徽医科大学第一附属医院(曾庆曙);福建医科大学附属协和医院(胡建达);厦门大学附属第一医院(徐兵);河南省肿瘤医院(宋永平);郑州大学第一附属医院(姜中兴);河南省人民医院(孙恺);华中科技大学附属协和医院(胡豫);华中科技大学附属同济医院(周剑峰);武汉大学中南医院(左学兰);中南大学湘雅医院(赵谢兰);暨南大学医学院血液病研究所(李扬秋);南方医科大学南方医院(刘启发、梁莉);广东省人民医院(杜欣、翁建宇);深圳市第二人民医院(杜新);南方医科大学珠江医院(李玉华);重庆医科大学(丁克越);陆军军医大学新桥医院(张曦、郭乔楠);四川大学华西医院(牛挺、刘卫平);陆军军医大学西南医院(陈洁平);四川省人民医院(黄晓兵);重庆医科大学第一附属医院(刘林);宁夏医科大学总医院(郑波);青海省人民医院(李文倩);云南省人民医院(杨同华);昆明医科大学第二附属医院(周泽平);新疆维吾尔自治区人民医院(王晓敏);海南省人民医院(姚红霞);天津市临床检验中心(杨彬);天津市肿瘤医院(张会来、李晓玲);中国医学科学院血液病医院(蔺亚妮、张冬雷、朱平、魏辉、邱录贵、肖志坚、王建祥、汝昆)
(执笔:蔺亚妮、张冬雷、朱平、刘红星、丁克越、乔纯、彭明婷、汝昆)