
金黄色葡萄球菌是儿童呼吸道感染的常见病原之一,由于其易于产生耐药,导致临床治疗面临许多问题。临床医师要掌握细菌耐药规律及其机制,并合理使用才能降低其快速产生耐药的速度。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
金黄色葡萄球菌(Staphylococcus aureus,简称金葡菌)是医院和社区获得性感染的常见致病菌之一,可引起一系列化脓性感染,包括皮肤软组织感染、肺炎、伪膜性肠炎、心包炎等,甚至引起菌血症、中毒性休克综合征等严重危及生命的疾病。自1961年在英国首次发现耐甲氧西林金黄色葡萄球菌(methicillin-resistant staphylococcusaureus,MRSA)以来,国内外MRSA的感染率不断攀升[1]。2005年美国Deleo和Chambers等[2]报道美国因MRSA感染死亡的住院患者总数已超过因肝炎和艾滋病死亡的人数总和,带来了极大的社会负担及经济负担。因此,MRSA感染已成为现代抗感染治疗中最关注的感染性难题之一。随着抗生素种类的不断增加及广泛应用,MRSA对多种抗生素耐药,耐药性日趋严重,所以MRSA引起的感染一旦发生,常难以控制。现将对MRSA的流行病学、耐药现状及耐药机制等进行综述。
1961年Jevons首先分离到MRSA,1975年美国在临床分离出的金葡菌中MRSA仅占2.4%,而1991年则迅速增至29.0%。近年来各个地区和医院的MRSA检出率均有逐年增加的趋势。现在美国的某些医院在临床分离出的金葡菌中MRSA可以占到30%~50%。同样在欧洲的葡萄牙和意大利,在临床分离出的金葡菌中MRSA占50%;土耳其和希腊>30%[3]。国内上海地区1977年至1979年MRSA占5%,1985年至1986年占24%,1990年综合性大医院增至50%,而1993年则上升至60%。重庆地区1983年至1985年为8.3%,1989年至1992年上升至18.7%;1995年北京5家教学医院MRSA分离率平均为47.0%[4]。2014年中国细菌耐药性监测(CHINET)数据显示金葡菌中MRSA的平均检出率为44.6%[5]。《儿童社区获得性肺炎管理指南》中指出,金葡菌是儿童社区获得性肺炎的重要细菌病原之一,近年陆续有社区相关性耐甲氧西林金黄色葡萄球菌(community-associated methicillin-resistance Staphylococcus aureus, CA-MRSA)感染社区获得性肺炎(CAP)的报道,多发生在年幼儿,应引起重视[6]。
MRSA除对甲氧西林耐药外,对其他与甲氧西林结构相同或相似的β内酰胺类抗菌药物皆可产生耐药,MRSA还能通过产生修饰酶、改变抗菌药物的作用靶点、降低膜通透性等不同机制,可对氨基糖苷类、氟喹喏酮类、大环内酯类、磺胺类、四环素类、利福平产生耐药。治疗MRSA引起的感染临床首选万古霉素等糖肽类抗菌药物,但在1997年日本报道了第1例中介度耐万古霉素的金葡菌(vancomycin-intermediate Staphylococcus aureus,VISA)[7],2002年美国疾病预防控制中心(CDC)报道了耐万古霉素的金葡菌(vancomycin -resistance Staphylococcus aureus,VRSA)[8]。随着VRSA的出现以及万古霉素明显的不良反应,限制了其临床应用。近年相继研发了一些新型抗MRSA的药物:利奈唑胺、达托霉素、替加环素、奎奴普丁/达福普汀等。目前已发现对这些新型抗生素耐药的MRSA。临床可用于治疗MRSA感染的抗菌药物越来越少,使MRSA成为临床最关注的一类耐药菌。
MRSA的耐药机制复杂,主要包括由染色体介导的固有耐药、通过质粒转移耐药基因获得的耐药(如VanA介导的万古霉素耐药)、基因表达调控有关的耐药和主动外排系统等。现就MRSA对临床常用药物的耐药机制进行介绍。
mecA基因决定了MRSA对β内酰胺类的耐药性,其是一个外源基因,来自凝固酶阴性葡萄球菌或肠球菌属,通过转座子或R质粒转到原本敏感的金葡菌中,并整合在染色体第10节段上。此基因负责编码一种新型的青霉素结合蛋白2a(PBP2a),该蛋白是一种酶,与β内酰胺类抗菌药物的亲和力很低,可代替正常的青霉素结合蛋白发挥作用,保证细菌肽聚糖和细胞壁合成,使细菌得以正常生长、繁殖,从而干扰了β内酰胺类抗菌药物的抗菌作用[9]。mecA基因位于葡萄球菌染色体mec盒(Staphy1ocossal cassette chromosome mec,SCCmec)上。SCCmec包括mec复合体(mec gene complex)、ccr复合体(cassette chromosome recombinases gene complex)以及J区。
mec基因复合体由结构基因mecA和位于其上游的调节基因mec R1和抑制基因mecI组成,三者控制MRSA耐药性的表达程度,其中mecA编码PBP2a,mecI编码的抑制因子(MEC I蛋白)结合在mecA基因的启动子部位,使mecA基因不能被转录;mecR1在诱导剂(如β内酰胺类抗菌药物)的作用下编码产生诱导因子(MECR1蛋白),能够去除MECI蛋白对mecA的阻遏作用,使mecA转录产生PBP2a[10]。mecA基因的表达还受BlaR1和BlaI基因的调节,二者与mecR1和mecI基因有高度的同源性[11]。目前已发现5种mec复合物类型(A到E)[12](表1)。

5种mec复合物类型
The five types of mec complex
5种mec复合物类型
The five types of mec complex
| 类型 | 结构 | SCCmec类型 |
|---|---|---|
| A | mecI-mecR1-mecA-IS431 | Ⅱ,Ⅲ,Ⅷ |
| B | IS1272-△mecR1-mecA-IS431 | Ⅰ,Ⅳ,Ⅵ |
| C | IS431-△mecR1-mecA-IS431 | Ⅴ,Ⅶ |
| D | △mecR1-mecA-IS431 | — |
| E | △mecR1-mecA-IS431 | — |
注:SCCmec:葡萄球菌染色体mec盒 SCCmec:Staphy1ocossal cassette chromosome mec
ccr复合体由ccr基因与其相邻的orfs组成,属于转化酶/解离酶类,存在于所有的SCCmec中。其功能是将SCCmec从供体菌的特定位点——SCCmec attachment site(attBscc)删除,整合进受体菌染色体中,在SCCmec的水平转移中发挥重要作用。ccr复合体可分为7类:ccrA1和ccrB1(SCCmec Ⅰ型),ccrA2和ccrB2 (SCCmec Ⅱ和Ⅳ型),ccrA3和ccrB3(SCCmecⅢ型),ccrA4和ccrB4 (SCCmecⅥ和Ⅷ型),ccrC1(SCCmecⅤ和Ⅶ型),ccrA1和ccrB6(SCCmecⅩ)以及ccrA1和ccrB3(SCCmecⅪ)[13,14,15,16,17,18]。
SCCmec盒上除了mec与ccr复合体外,还有J区,是SCCmec的非必需成分,也是SCCmec划分亚型的依据。每个SCCmec元件具有3个J区,具有如下的组成结构:J3-mec-J2-ccr-J1。J区包含各种假基因及转座、插入序列,一些从质粒或转座子获得的对非β内酰胺类抗生素和重金属耐药的基因也存在于J区(如介导红霉素、链霉素耐药基因的Tn554,带有博来霉素、妥布霉素耐药基因的PUB110,携带介导氨基糖苷类抗菌药物耐药基因aacA-aphD的Tn4001,与四环素耐药密切相关的PT181等)。因此,SCCmec的存在使MRSA表现出对多种抗生素的耐药能力[10]。现在认为J区是细菌在进化过程中为适应环境,从外界获得对其生存有益的基因。
SCCmec依靠mec复合体和ccr复合体的不同来分型,目前已鉴定出11种不同的SCCmec亚型(表2)[19]。
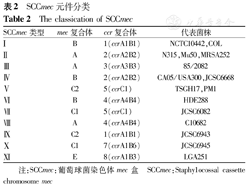
SCCmec元件分类
The classication of SCCmec
SCCmec元件分类
The classication of SCCmec
| SCCmec类型 | mec复合体 | ccr复合体 | 代表菌株 |
|---|---|---|---|
| Ⅰ | B | 1(ccrA1B1) | NCTC10442,COL |
| Ⅱ | A | 2(ccrA2B2) | N315,Mu50,MRSA252 |
| Ⅲ | A | 3(ccrA3B3) | 85/2082 |
| Ⅳ | B | 2(ccrA2B2) | CA05/USA300,JCSC6668 |
| Ⅴ | C2 | 5(ccrC1) | TSGH17,PM1 |
| Ⅵ | B | 4(ccrA4B4) | HDE288 |
| Ⅶ | C1 | 5(ccrC1) | JCSC6082 |
| Ⅷ | A | 4(ccrA4B4) | C10682 |
| Ⅸ | C2 | 1(ccrA1B1) | JCSC6943 |
| Ⅹ | C1 | 7(ccrA1B6) | JCSC6945 |
| Ⅺ | E | 8(ccrA1B3) | LGA251 |
注:SCCmec:葡萄球菌染色体mec盒 SCCmec:Staphy1ocossal cassette chromosome mec
研究表明PBP2a产量与MRSA耐药水平的高低及最小抑菌浓度(MIC)值并无相关性,PBP2a产量相同的MRSA的MIC值可相差很大,提示除了mecA基因外还有其他耐药基因。femA、femB、femC、femd、femE和femF是金葡菌染色体上的固有基因,与甲氧西林的耐药表达有关,这些辅助基因与mecA基因的协调作用产生对β内酰胺类抗生素的高度耐药性,这是由染色体介导的固有耐药。hmrC、hmeD、chr基因是染色体突变基因,引起金葡菌对甲氧西林高度耐药,其机制尚未明确。mecA基因的表达还受环境因素,如pH、湿度及生长温度的影响[20]。
大多数MRSA菌株的一个显著特点为对β-内酰胺类抗菌药物的耐药性表现为异型性。即来自于一个菌落的接种培养细菌,大多数的培养细胞表现为低水平耐药, 0.01%~0.10%的培养细胞表现为高水平耐药,菌株表现出统一高水平耐药是很少见的。这就是所谓的异型性耐药[9]。对实验室菌株进行基因组测序,比较高水平耐药亚群与它们的亲本菌群,发现在27个基因及3个间区域中至少有44个不同的基因突变参与了异型性耐药[21]。
万古霉素是糖肽类抗生素的代表药物。由于MRSA的广泛流行,万古霉素成为治疗MRSA感染的首选药物。令人担心的是出现了对万古霉素不再敏感的菌株,首先是表现为对万古霉素中介敏感,之后出现对万古霉素完全耐药的菌株。首先,除了具有异质耐药性的菌株外,VISA和VRSA基本上均来自MRSA;其次,耐药性不是逐步发展的,VRSA不是来源于VISA,即VISA和VRSA具有完全不同的耐药机制。日本早在1996年就发现了VISA,相继多个国家均有报道。回顾性分析发现早在1987年的库存菌株中即存在VISA[22]。根据万古霉素敏感性断点,VISA菌株可以被中间浓度范围的万古霉素所抑制(万古霉素MIC为8~16 mg/L)[23]。一般而言,替考拉宁对VISA的MIC明显高于万古霉素。一些菌株在常规药敏试验中是对万古霉素敏感的,因此,只能通过复杂的方法(种群分析)发现对万古霉素部分或者完全耐药的亚群。这些菌株被称为异质性(H)-VISA,而且可能较报告更常见。由于万古霉素治疗严重感染的失败往往是经历了VISA,H-VISA以及对万古霉素临界MIC处于敏感和中介之间的菌株。VISA的耐药性是在万古霉素存在的情况下,金葡菌对多种因素适应的结果,如肽聚糖合成变化而导致的细胞壁增厚及细胞壁自溶的降低,D-丙胺酰-D-丙氨酸残基的增加,造成假靶位来"亲和诱捕"或"阻塞"万古霉素[24]。目前未发现VISA典型的遗传特征,但是发现其与一个特殊的同质多象变体(agr Ⅱ)关系密切,在万古霉素存在情况下,agr功能缺失赋予了其选择性的生存优势[25]。
相对于VISA,VRSA的耐药机制更明确,研究证实其耐药基因来源于共同分离的耐万古霉素的肠球菌(vancomycin-resistant enterococci,VRE),由vanA基因组编码,其位于1个11 kb的转座子Tn1546上,由vanH、vanA、vanX和附加基因等组成,合成的蛋白质具有以下作用:将肽聚糖前体末端的D-丙氨酰-D-丙氨酸修饰为D-丙氨酰-D-乳酸,以及清除敏感野生型药物作用靶点。带有D-丙氨酰-D-乳酸末端的前体较带有D-丙氨酰-D-丙氨酸末端的野生型前体对万古霉素的亲和力更低。Tnl546编码9个多肽,这9个多肽协同作用最终形成D-丙氨酰-D乳酸,取代了细菌肽聚糖前体中的D-丙氨酰-D-丙氨酸,使万古霉素失去作用位点,导致金葡菌对万古霉素高水平耐药[26]。这一耐药机制受双组分信号转导系统(vanS和vanR基因)的调控,仅在万古霉素存在的情况下才可激活这一系统[27]。分子学研究表明,如果体内存在MRSA和VRE的混合感染,质粒可以介导Tn1546从VRE到VRSA的水平转移[28]。
环丙沙星引入临床不久,就出现了对喹诺酮类抗生素耐药的MRSA。由于编码DNA旋转酶(gyrA和gyrB)和拓扑异构酶Ⅳ(parC和parE)的核酸突变,导致酶氨基酸的置换,使氟喹诺酮药物不能与靶位结合,从而导致MRSA对喹诺酮的耐药。大多数耐药MRSA至少包含2个突变,一个在parc基因上,一个在gyrA基因上[29]。在某些菌株中,外排泵基因NorA的高表达可导致耐药表型的产生。多种突变及耐药机制的组合可引起对新型喹诺酮药物的交叉耐药[30]。
 唑烷酮类抗菌药物的耐药机制
唑烷酮类抗菌药物的耐药机制 唑烷酮类的代表药物为利奈唑胺,为蛋白质合成抑制剂,作用于翻译的起始阶段,与核糖体50 S亚基结合,抑制mRNA与核糖体连接,阻止70 S起始复合物的形成,从而抑制了细菌蛋白质的合成。研究表明,利奈唑胺结合23 S rRNA V区中心环,即肽基转移酶, 23 S rRNA中G2576U、U2500A、G2447U的替换均可导致利奈唑胺与靶位结合受阻而导致耐药[31]。此外,质粒编码甲基化酶对23 S rRNA A2503残基修饰也可造成MRSA对利奈唑胺的耐药[32]。
唑烷酮类的代表药物为利奈唑胺,为蛋白质合成抑制剂,作用于翻译的起始阶段,与核糖体50 S亚基结合,抑制mRNA与核糖体连接,阻止70 S起始复合物的形成,从而抑制了细菌蛋白质的合成。研究表明,利奈唑胺结合23 S rRNA V区中心环,即肽基转移酶, 23 S rRNA中G2576U、U2500A、G2447U的替换均可导致利奈唑胺与靶位结合受阻而导致耐药[31]。此外,质粒编码甲基化酶对23 S rRNA A2503残基修饰也可造成MRSA对利奈唑胺的耐药[32]。
面对VISA和VRSA的出现,临床上选择万古霉素与二线抗金葡菌药物联合应用治疗VISA或VRSA感染。甲氧苄啶-磺胺甲基异 唑及四环素越来越多的应用于治疗MSSA和MRSA导致的皮肤软组织感染。对甲氧苄啶-磺胺甲基异
唑及四环素越来越多的应用于治疗MSSA和MRSA导致的皮肤软组织感染。对甲氧苄啶-磺胺甲基异 唑的耐药性可使病原菌通过选择性代谢途径以免受合成药物抑制。对甲氧苄啶的耐药机制是染色体突变,而对磺胺甲基异
唑的耐药性可使病原菌通过选择性代谢途径以免受合成药物抑制。对甲氧苄啶的耐药机制是染色体突变,而对磺胺甲基异 唑的耐药机制是获得由转座子编码的drfA基因[33]。对四环素的耐药机制有2个:一是主动外排药物,由质粒携带的基因tetK和tetL参与;一是保护四环素作用靶点,由tetM和tetO参与[34]。夫西地酸作为治疗葡萄球菌皮肤感染局部用药,目前已出现频繁耐药现象。对夫西地酸的耐药性与染色体上的fusA基因突变以及fusB因子的获得有关[35]。
唑的耐药机制是获得由转座子编码的drfA基因[33]。对四环素的耐药机制有2个:一是主动外排药物,由质粒携带的基因tetK和tetL参与;一是保护四环素作用靶点,由tetM和tetO参与[34]。夫西地酸作为治疗葡萄球菌皮肤感染局部用药,目前已出现频繁耐药现象。对夫西地酸的耐药性与染色体上的fusA基因突变以及fusB因子的获得有关[35]。
尽管MRSA对克林霉素的耐药性不断增加,但目前仍作为抗MRSA的备选药物。葡萄球菌属对克林霉素的耐药性与erm基因相关,由erm基因编码产生的红霉素核糖体甲基化酶会导致23 S rRNA的甲基化,降低了对大环内酯-林可胺类-链阳菌素(MLSB)类抗生素与核糖体的结合而产生耐药。在MRSA中,Tn554-like转座子携带的ermA基因在MRSA中最常见[36]。由erm基因介导的耐药又分为结构型和诱导型2种。结构型耐药表型对红霉素和克林霉素均耐药,可通过药敏试验检测;诱导型耐药表型是对红霉素耐药而对克林霉素敏感,对克林霉素是否真的敏感可经大环内酯类药物诱导产生耐药现象的D-试验检测。
大多数MRSA仍对利福平敏感,利福平可用于难治性感染。如果用于单一治疗,久之也会产生耐药性,其耐药性是由于rpoB基因点突变所致,降低药物与利福平的亲和力[33]。
达托霉素是脂肽类抗菌药物。达托霉素耐药罕见,其机制为多因素,现尚未完全明了。染色体编码赖氨酸磷脂酰甘油合成酶基因(mprF),传感器组氨酸激酶基因(yycG),RNA聚合酶亚β和β'单位基因(rpoβ和ropC)的突变可导致对达托霉素敏感性的降低。体外研究显示VISA对达托霉素的敏感性低于万古霉素敏感菌,这种交叉耐药可能归因于VISA增厚的细胞壁阻挡了达托霉素向细胞膜的渗透[37]。
MRSA耐药性日趋严重,且呈现多重耐药现象,耐药谱越来越广,耐药机制复杂,尤其是对万古霉素的耐药性,给临床治疗带来了严峻的挑战。为寻找有效的治疗方法,必须深入研究MRSA的耐药机制及各种机制间的相互作用。检测MRSA的耐药结构可以为临床治疗及感染控制提供依据,值得重视。



























