
明确临床常规检测为肺炎链球菌,但荚膜肿胀试验阴性的分离菌株中是否存在其他草绿色链球菌,分析其抗生素敏感性,为临床选药提供参考。
以105株临床分离但荚膜肿胀试验阴性的肺炎链球菌为研究对象,采用草绿色链球菌的多位点序列分析(MLSA)和基质辅助激光解析电离飞行时间质谱(MALDI-TOF-MS)进行菌种鉴定;采用微量肉汤稀释法检测14种抗菌药物的敏感性。
105株细菌经MLSA确认24株为假肺炎链球菌,其余81株为肺炎链球菌。MALDI-TOF-MS将6株假肺炎链球菌鉴定为肺炎链球菌,3株鉴定为缓症/口腔链球菌;6株肺炎链球菌被鉴定为假肺炎链球菌。全部菌株均对万古霉素、左氧氟沙星和莫西沙星敏感,肺炎链球菌与假肺炎链球菌对头孢呋辛(28.4%和58.4%)、氯霉素(39.5%和4.2%)、红霉素(77.8%和95.8%)和阿奇霉素(75.3%和95.8%)等抗生素不敏感率有明显不同。
临床常规检测可能将部分假肺炎链球菌鉴定为肺炎链球菌,质谱分析也不能确保正确鉴定;荚膜肿胀试验阴性的肺炎链球菌分离株耐药性情况存在差异,鉴定失误可能影响病原菌及其耐药性的评估。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
临床检测中肺炎链球菌培养鉴定的常规方法包括菌落形态(α-溶血环等)、奥普托欣敏感性试验(OP试验)和胆汁溶菌试验。荚膜肿胀试验(Capsular swelling test,又称Quellung reaction)是肺炎链球菌血清分型的鉴定方法,但很少应用于临床检测,主要在肺炎链球菌流行病学监测中使用。早有关于奥普托欣敏感的草绿色链球菌[1,2]和胆汁溶菌试验阴性的肺炎链球菌的报道[3],这些方法在鉴定肺炎链球菌时存在一定缺陷[4]。同时,肺炎链球菌定植的呼吸道也常常可以分离出假肺炎链球菌、缓症链球菌、口腔链球菌等草绿色链球菌,一般常规鉴定容易出现混淆。本课题组在肺炎链球菌流行病学监测中发现,部分常规鉴定的肺炎链球菌菌株在血清分型时荚膜肿胀试验阴性,不能确定血清型,推测其中可能有其他绿色链球菌。为明确这些菌株的菌种,本研究采用多位点序列分析(MLSA)方法[5,6]进行鉴定,并评估目前临床常用的基质辅助激光解吸电离飞行时间质谱(MALDI-TOF-MS)鉴定这些菌株的价值,同时检测这些菌株的抗生素药物敏感性供临床参考。
本研究菌株收集自本课题组既往研究的临床分离肺炎链球菌,共计105株经形态学、奥普托欣试验和胆汁溶菌试验鉴定为肺炎链球菌,但全价Omni血清(丹麦国家血清研究院,哥本哈根,丹麦)荚膜肿胀试验阴性。
将可疑肺炎链球菌菌落(草绿色溶血环、边缘整齐、可有脐窝状凹陷)转种于50 mL/L脱纤维羊血胰蛋白胨大豆琼脂(TSA)培养皿上,贴上Optochin纸片(DD0001B 5 μg optochin,Oxoid Ltd,UK),35 ℃ 50 mL/L二氧化碳(CO2)条件下孵育18~24 h。Optochin(OP)纸片抑菌环直径>14 mm为阳性,即判定为肺炎链球菌。临床常规检测中,若OP抑菌环直径≤14 mm,应结合胆汁溶菌试验结果综合判定,胆汁溶菌试验阳性则可判定为肺炎链球菌[7]。
胆汁溶菌试验:制备1.0 mL 18~24 h待检菌液2管,1管加入100 g/L脱氧胆酸钠溶液0.1 mL(DZ0240脱氧胆酸钠溶液,pH 7.0,雷根生物,北京),另1管加等量9 g/L盐水作对照,混匀,置于37 ℃水浴10~30 min;菌液由浑浊变澄清为阳性结果。
用无菌棉签挑取适量的菌落至含有磷酸缓冲液(PBS)的1.5 mL PCR小管中,混匀形成悬液;取1.5 μL细菌悬液至于载玻片上,另取同等量购自丹麦国家血清研究院(Statens Serum institut,哥本哈根,丹麦)的Omni全价抗血清与之充分混匀,相差显微镜1 000倍下观察细菌大小、荚膜肿胀和凝集状况。与纯菌液比较,荚膜肿胀阳性反应者镜下菌体明显变大,多数还伴不同程度的细菌凝集,阴性结果镜下没有上述变化。
采用硅胶模型™基因组DNA提取试剂盒(北京赛百盛基因技术有限公司,北京)提取菌株DNA,实验步骤参照说明书中革兰阳性菌操作流程进行。本研究采用草绿色链球菌MLSA鉴定方法,通过PCR扩增map、pfl、ppaC、pyk、rpoB、sodA、tuf等7个目的基因。引物和扩增信息参考文献[8]。扩增产物送至北京天一辉远公司双向测序。MLSA使用的数据库下载自网站(http://www.eMLSA.net),其中包含了Bishop等研究中的全部菌株序列信息。
得到测序结果后,使用MEGA 4软件将正反向序列拼接,与数据库中标准序列对比切割获得等长目的序列;再按照map-pfl-ppaC-pyk-rpoB-sodA-tuf顺序串联拼接为总长度3 063 bp序列。使用MEGA 4软件将本研究菌株序列与数据库全部菌株序列构建系统发育树,采用邻位归并法(neighbour-joining tree,NJ tree)重复1 000次建树,得到分析结果,确定检测菌株菌种。
采用法国生物梅里埃公司的MALDI-TOF-MS设备Vitek MS对菌株进行鉴定,按照其操作规程操作。图谱数据使用MYLA软件比对Vitek MS IVD 3.0标准数据库得出结果。大肠埃希菌ATCC8739作为仪器的校准株。
本研究使用微量肉汤稀释法检测菌株对青霉素、头孢曲松、红霉素等14种抗生素的最低抑菌浓度(MIC)。采用Sensititre™ ARIS™ 2X全自动鉴定及药敏分析系统(赛默飞世尔科技有限公司,美国)测定读取MIC值,操作依照系统配套手册。结果判定依据2019年美国临床和实验室标准协会(CLSI)标准,由于假肺炎链球菌没有CLSI标准,敏感性判读使用肺炎链球菌标准。药敏检测质控菌株为肺炎链球菌ATCC49619。
使用MEGA 4软件处理分析菌株MLSA序列,并构建系统发育树,以鉴定菌种。采用SPSS 23.0软件进行统计学分析,计数资料以例数或率表示。率的比较采用χ2检验或Fisher′s精确概率法,P<0.05为差异有统计学意义。
105株常规检测为肺炎链球菌但荚膜肿胀试验阴性菌株的MLSA结果见图1。结果显示其中24株为假肺炎链球菌,其余81株为肺炎链球菌,未发现其他草绿色溶血链球菌。本研究菌株与数据库(http://www.eMLSA.net)中肺炎链球菌与假肺炎链球菌部分MLSA分析所建系统进化树见图2,图中可见2组细菌分支明确,本研究检测的肺炎链球菌和假肺炎链球菌有比较集中的遗传分支,同时也存在一些散在分布。
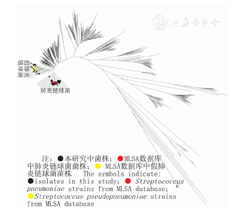

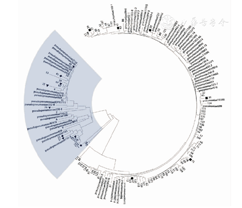

注:■Vitek MS错误鉴定为假肺炎链球菌的肺炎链球菌菌株;▲Vitek MS错误鉴定为肺炎链球菌的假肺炎链球菌;◆Vitek MS错误鉴定为缓症/口腔链球菌的假肺炎链球菌;MLSA:多位点序列分析 ■Streptococcus pneumoniae isolates which were wrongly identified asStreptococcus pseudopneumoniae by Vitek MS;▲Streptococcus pseudopneumoniae isolates which were wrongly identified asStreptococcus pneumoniae by Vitek MS;◆Streptococcus pseudopneumoniae isolates which were wrongly identified asStreptococcus mitis/oralis by Vitek MS;MLSA:multilocus sequence analysis
根据MLSA结果,其中24株假肺炎链球菌菌株的OP纸片抑菌环直径均>14 mm(14~22 mm,中位数为15 mm)。假肺炎链球菌菌株胆汁溶菌试验结果显示有5株为阳性,见表1。
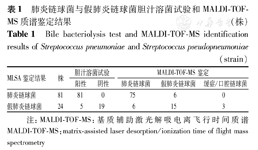
肺炎链球菌与假肺炎链球菌胆汁溶菌试验和MALDI-TOF-MS质谱鉴定结果(株)
Bile bacteriolysis test and MALDI-TOF-MS identification results ofStreptococcus pneumoniae andStreptococcus pseudopneumoniae(strain)
肺炎链球菌与假肺炎链球菌胆汁溶菌试验和MALDI-TOF-MS质谱鉴定结果(株)
Bile bacteriolysis test and MALDI-TOF-MS identification results ofStreptococcus pneumoniae andStreptococcus pseudopneumoniae(strain)
| MLSA鉴定结果 | 株 | 胆汁溶菌试验 | MALDI-TOF-MS鉴定 | |||
|---|---|---|---|---|---|---|
| 阳性 | 阴性 | 肺炎链球菌 | 假肺炎链球菌 | 缓症/口腔链球菌 | ||
| 肺炎链球菌 | 81 | 81 | 0 | 75 | 6 | 0 |
| 假肺炎链球菌 | 24 | 5 | 19 | 6 | 15 | 3 |
注:MALDI-TOF-MS:基质辅助激光解吸电离飞行时间质谱MALDI-TOF-MS:matrix-assisted laser desorption/ionization time of flight mass spectrometry
Vitek MS鉴定结果见表1,24株假肺炎链球菌中,6株被鉴定为肺炎链球菌,3株鉴定为缓症/口腔链球菌;6株肺炎链球菌被鉴定为假肺炎链球菌。图2也显示Vitek MS鉴定错误的菌株之间亲缘关系相距较远,位于明显不同的分支。
抗生素敏感性检测结果见表2。全部菌株均对万古霉素、左氧氟沙星和莫西沙星敏感。肺炎链球菌对头孢呋辛、红霉素和阿奇霉素的不敏感率分别为28.4%、77.8%和75.3%,明显低于假肺炎链球菌的58.4%、95.8%和95.8%,差异均有统计学意义(χ2=105.0、99.3、105.0,均P<0.05),表明假肺炎链球菌对以上抗生素耐药性均高于肺炎链球菌。肺炎链球菌氯霉素不敏感率(39.5%)明显高于假肺炎链球菌(4.2%)。
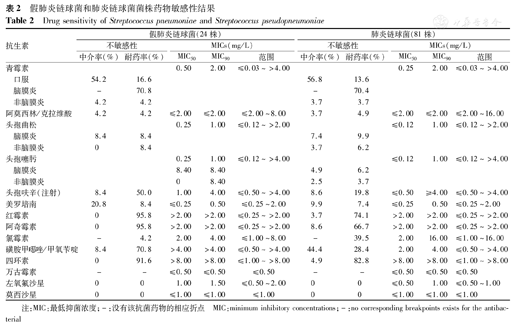
假肺炎链球菌和肺炎链球菌菌株药物敏感性结果
Drug sensitivity ofStreptococcus pneumoniae andStreptococcus pseudopneumoniae
假肺炎链球菌和肺炎链球菌菌株药物敏感性结果
Drug sensitivity ofStreptococcus pneumoniae andStreptococcus pseudopneumoniae
| 抗生素 | 假肺炎链球菌(24株) | 肺炎链球菌(81株) | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 不敏感性 | MICs(mg/L) | 不敏感性 | MICs(mg/L) | ||||||||
| 中介率(%) | 耐药率(%) | MIC50 | MIC90 | 范围 | 中介率(%) | 耐药率(%) | MIC50 | MIC90 | 范围 | ||
| 青霉素 | 0.50 | 2.00 | ≤0.03~>4.00 | 0.25 | 2.00 | ≤0.03~>4.00 | |||||
| 口服 | 54.2 | 16.6 | 56.8 | 13.6 | |||||||
| 脑膜炎 | - | 70.8 | - | 70.4 | |||||||
| 非脑膜炎 | 4.2 | 4.2 | 3.7 | 3.7 | |||||||
| 阿莫西林/克拉维酸 | 4.2 | 4.2 | ≤2.00 | ≤2.00 | ≤2.00~8.00 | 3.7 | 4.9 | ≤2.00 | ≤2.00 | ≤2.00~16.00 | |
| 头孢曲松 | 0.25 | 1.00 | ≤0.12~>2.00 | ≤0.12 | 1.00 | ≤0.12~>2.00 | |||||
| 脑膜炎 | 8.4 | 8.4 | 7.4 | 9.9 | |||||||
| 非脑膜炎 | 0 | 8.4 | 3.7 | 6.2 | |||||||
| 头孢噻肟 | 0.25 | 1.00 | ≤0.12~>4.00 | ≤0.12 | 1.00 | ≤0.12~>4.00 | |||||
| 脑膜炎 | 8.40 | 8.40 | 4.9 | 6.2 | |||||||
| 非脑膜炎 | 0 | 8.40 | 2.5 | 3.7 | |||||||
| 头孢呋辛(注射) | 8.4 | 50.0 | 1.00 | 4.00 | ≤0.50~>4.00 | 8.6 | 19.8 | ≤0.50 | ≥4.00 | ≤0.50~>4.00 | |
| 美罗培南 | 20.8 | 8.4 | ≤0.25 | 0.50 | ≤0.25~2.00 | 9.9 | 7.4 | ≤0.25 | 0.50 | ≤0.25~2.00 | |
| 红霉素 | 0 | 95.8 | >2.00 | >2.00 | ≤0.25~>2.00 | 3.7 | 74.1 | >2.00 | >2.00 | ≤0.25~>2.00 | |
| 阿奇霉素 | 0 | 95.8 | >2.00 | >2.00 | ≤0.25~>2.00 | 8.6 | 66.7 | >2.00 | >2.00 | ≤0.25~>2.00 | |
| 氯霉素 | - | 4.2 | 2.00 | 4.00 | ≤1.00~8.00 | - | 39.5 | 2.00 | 16.00 | ≤1.00~16.00 | |
磺胺甲 唑/甲氧苄啶 唑/甲氧苄啶 | 8.4 | 70.8 | >4.00 | >4.00 | ≤0.50~>4.00 | 44.4 | 28.4 | 2.00 | 4.00 | ≤0.50~>4.00 | |
| 四环素 | 0 | 91.6 | >8.00 | >8.00 | ≤1.00~>8.00 | 4.9 | 82.8 | >8.00 | >8.00 | ≤1.00~>8.00 | |
| 万古霉素 | - | - | ≤0.50 | ≤0.50 | ≤0.50 | - | - | ≤0.50 | ≤0.50 | ≤0.50 | |
| 左氧氟沙星 | 0 | 0 | 1.00 | 1.50 | ≤0.50~2.00 | 0 | 0 | ≤0.50 | 1.00 | ≤0.50~1.00 | |
| 莫西沙星 | 0 | 0 | ≤1.00 | ≤1.00 | ≤1.00 | 0 | 0 | ≤1.00 | ≤1.00 | ≤1.00 | |
注:MIC:最低抑菌浓度;-:没有该抗菌药物的相应折点 MIC:minimum inhibitory concentrations;-:no corresponding breakpoints exists for the antibacterial
草绿色链球菌是一组很难通过表型明确鉴定菌种的一组细菌。针对特定基因,如肺炎链球菌溶血素、自溶素或表面抗原等,设计普通、多重、实时荧光定量PCR或限制性片段长度多态性等方法的鉴定准确性都存在问题[9,10]。就目前来讲,没有单一的方法能够完全准确地鉴定肺炎链球菌,尤其是与亲缘关系接近的假肺炎链球菌和缓症链球菌群,临床应考虑结合不同的方法,以期做到准确诊断。MLSA是基于多个管家基因的串联序列通过构建系统发育树的方法鉴定菌种,研究证明该方法能消除草绿色链球菌中常见的同源重组带来的潜在问题,能准确鉴定菌种。虽然MLSA可作为鉴定的金标准,但由于成本和工作量太大,在临床微生物实验室并不可行[11,12]。
本研究显示临床常规检测为肺炎链球菌但荚膜肿胀试验阴性的菌株中,以MLSA方法确定有假肺炎链球菌,占22.9%。Arbique等[13]2004年根据奥普托欣敏感性和胆汁溶菌试验结果经DNA同源性研究确认,将先前部分鉴定为不典型肺炎链球菌的菌株命名为假肺炎链球菌,假肺炎链球菌鉴定特征为50 mL/L CO2环境中孵育对奥普托欣抵抗,在大气环境中敏感,且胆汁溶菌试验阴性,可与肺炎链球菌区分开。本研究中的24株假肺炎链球菌在50 mL/L CO2环境中均对奥普托欣敏感,且5株菌株胆汁溶菌试验也呈阳性,被常规鉴定为肺炎链球菌。Rolo等[5]对61株假肺炎链球菌的常规检测发现16.4%在50 mL/L CO2环境中培养对奥普托欣敏感,并且36.1%胆汁溶菌试验阳性。上述结果表明常规表型鉴定方法不能完全区分肺炎链球菌和假肺炎链球菌。
MALDI-TOF-MS近年来越来越多应用于临床微生物实验室。van Prehn等[14]分别用Microflex(布鲁克公司,不来梅,德国)和Vitek MS对11株肺炎链球菌和13株假肺炎链球菌分离株进行菌种鉴定,结果显示Microflex无法将2株肺炎链球菌准确鉴定,将3株假肺炎链球菌鉴定为肺炎链球菌;Vitek MS将1株肺炎链球菌鉴定为口腔/缓症链球菌,全部假肺炎链球菌准确鉴定。Angeletti等[15]的类似研究中,Microflex未能鉴别出假肺炎链球菌,Vitek MS也只能鉴定出17个假肺炎球菌中的2个。可见不同品牌的MALDI-TOF-MS在鉴定肺炎链球菌和假肺炎链球菌的能力上存在差别。本研究中也发现Vitek MS并不能完全准确地鉴定荚膜肿胀试验阴性的肺炎链球菌和假肺炎链球菌。随着仪器的更新换代及数据库和图谱处理软件的完善,MALDI-TOF-MS将来可以更准确地区分肺炎链球菌和其他草绿色链球菌。
本研究MLSA鉴定结果表明,大部分荚膜肿胀试验阴性菌株是肺炎链球菌,属于不可分型肺炎链球菌(NTSp)。NTSp产生机制可能包括因环境营养原因未产生荚膜、编码荚膜位点突变不能合成荚膜,或还未制备相关抗清的新型荚膜等[16]。多项早期研究表明,绝大多数NTSp都是缺乏荚膜的即无荚膜肺炎链球菌(NESp)[17,18]。
假肺炎链球菌的致病意义目前尚不清楚,很可能是条件致病菌[19]。有证据表明假肺炎链球菌能够定植于呼吸道和口咽[11]。其致病性在小鼠模型中得到明确证明,腹腔注射假肺炎链球菌可导致100%的小鼠死亡[20],但发现遗传物质的经典实验证明小鼠腹腔注射无荚膜的粗糙型肺炎链球菌不会导致小鼠死亡。临床研究提示假肺炎链球菌对人具有致病性。Keith等[21]描述了美国假肺炎链球菌与慢性阻塞性肺病(COPD)加重的关系,回顾性病例对照研究中,与从痰中分离出肺炎链球菌的患者相比,感染假肺炎链球菌的患者更有可能有COPD病史或COPD加重。近期也有假肺炎链球菌引起的迟发性疱疹相关眼内炎[22]和血培养分离出假肺炎链球菌[23]的报道。
目前假肺炎链球菌的药物敏感性数据十分有限,并且无相应判读标准参考。Laurens等[24]研究中140株假肺炎链球菌对万古霉素、左氧氟沙星和莫西沙星的不敏感率均为零,本研究结果与之一致。本研究红霉素、阿奇霉素、四环素和磺胺甲 唑/甲氧苄啶的耐药率高于国外报道[11],可能是由于本研究菌株均分离自儿童。本研究中肺炎链球菌和假肺炎链球菌对青霉素、阿莫西林/克拉维酸、头孢曲松和头孢噻肟相似的不敏感率,提示临床中的错误鉴定可能并不会影响经验性抗生素治疗的效果。方潮等[25]研究中国9家儿童医院分离肺炎链球菌的耐药性显示对青霉素非脑膜炎和脑膜炎的不敏感率分别为25.1%和86.9%,均高于本研究结果。这一情况可能源于NTSp与有荚膜肺炎链球菌的不同,中国台湾一项NTSp研究中全部菌株均对青霉素敏感[26]。
唑/甲氧苄啶的耐药率高于国外报道[11],可能是由于本研究菌株均分离自儿童。本研究中肺炎链球菌和假肺炎链球菌对青霉素、阿莫西林/克拉维酸、头孢曲松和头孢噻肟相似的不敏感率,提示临床中的错误鉴定可能并不会影响经验性抗生素治疗的效果。方潮等[25]研究中国9家儿童医院分离肺炎链球菌的耐药性显示对青霉素非脑膜炎和脑膜炎的不敏感率分别为25.1%和86.9%,均高于本研究结果。这一情况可能源于NTSp与有荚膜肺炎链球菌的不同,中国台湾一项NTSp研究中全部菌株均对青霉素敏感[26]。
本研究尚存在不足之处。本研究中菌株是实验室多个课题研究过程中积累的菌株,大部分无法获得临床病例的确切诊断,不能对2组菌株是否是临床致病菌株,及其感染类型做出准确推断,因此不能从临床角度评估菌株致病力。另外,虽然对所有菌株进行了准确的菌种鉴定,但对81株荚膜肿胀试验阴性的肺炎链球菌没有检测荚膜基因变异和表达情况,如果从分子角度阐明其未表达荚膜的机制,可进一步确认研究结果,还可能提出菌种鉴定方法。
综上所述,临床常规检测过程中,可能将部分假肺炎链球菌鉴定为肺炎链球菌,质谱分析也不能确保正确区分肺炎链球菌和假肺炎链球菌。这些假肺炎链球菌OP抑菌环直径靠近阳性判定界值,增加胆汁溶菌试验也不能完全避免鉴定失误。荚膜肿胀试验阴性的肺炎链球菌与假肺炎链球菌的耐药性情况也存在差异,其流行病学特征和意义需要进一步监测和研究。
所有作者均声明不存在利益冲突



























