
结核病目前仍是严重危害儿童健康的重要疾病,儿童结核病的早期发现和诊断仍面临诸多问题,如传统检测方法检出率低、漏诊率高,儿童咳痰困难、痰标本细菌载量低,肺外结核病诊断困难。因此,迫切需要发现新的结核病诊断标志物以建立快速敏感且高效的结核病诊断方法,而生物标志物则为我们指明了方向。现从转录组学、蛋白质组学、代谢组学和microRNA等4个方面,对儿童结核病新型生物标志物的应用研究进展进行阐述。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
由结核分枝杆菌(Mycobacterium tuberculosis,MTB)引起的结核病是严重危害人类健康的重要传染病之一。2019年世界卫生组织(World Health Organization,WHO)全球结核病报告显示,2018年全球新发病例1 000万,其中儿童结核病约占110万;约145万人死于结核病,结核病已经成为全世界由单一传染性病原体引起死亡的最大杀手[1]。众所周知,快速准确诊断对于指导治疗至关重要,但目前整体的结核病病例检出率和报告率很低,其中65%的儿童结核病无法被识别或报告[2],诊断不足为结核病的传播提供了更多机会,尤其是在经济不发达和地理位置偏僻的国家或地区。另外,儿童痰标本采集困难,因此儿童肺结核和肺外结核病诊断面临着更多问题[3]。
据WHO估计,在结核病新发病例背后隐藏着约20亿的MTB隐性感染者。这些结核潜伏感染(latent tuberculosis infection,LTBI)个体中约10%会最终发展为活动性结核病。目前用于结核病诊断的方法主要包括影像学、细菌学、免疫学和分子生物学等方面,这些方法或者检测周期长、检出率低、漏诊率高(如细菌培养),或不能区分LTBI和活动性结核病[如γ-干扰素释放试验(interferon-gamma release assays,IGRAs)],或受限于儿童痰液等标本细菌载量低的特点,检测的敏感性不如成人标本[如Xpert MTB/利福平(Rifampicin,RIF)],或因设备、技术和成本等原因不适合中低收入及结核病高负担国家,因此迫切需要开发新型结核病诊断标志物,建立快速敏感且高效的结核病诊断方法,用以区分LTBI和活动性结核病,或进行结核病发病风险的预测和治疗效果(包括治疗失败或复发)的监测,以及为新药和疫苗的临床试验提供候选靶标[4]。现从转录组学、蛋白组学、代谢组学和micro-RNA(miRNA)等4个方面对结核病新型生物标志物进行阐述,以了解该方向的最新研究进展。
转录组学是从RNA水平研究基因表达的情况,是研究细胞表型和功能的一个重要手段。主要通过筛选出与疾病相关的具有诊断意义的特异性差异表达,从而发现新的生物标志物用于疾病的诊断和风险预测。目前可利用的技术包括RNA测序、微阵列(芯片)、反转录多重连接探针扩增技术(reverse transcriptase multiplex ligation-dependent probe amplification,RT-MLPA)或反转录定量聚合酶链反应(reverse transcription quantitative polymerase chain reaction,RT-qPCR)等。
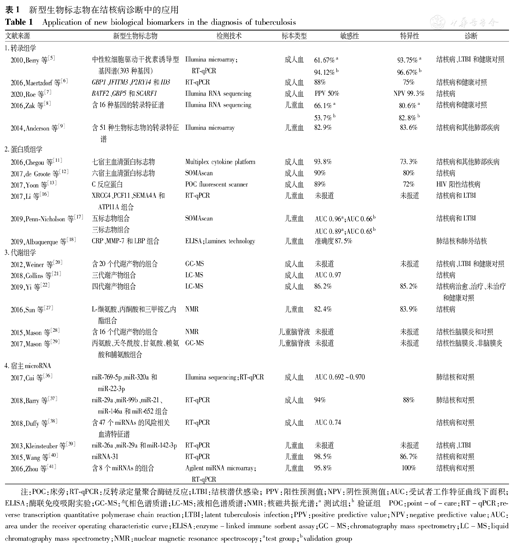
针对成人结核病的诊断和风险预测,研究者们发现了一些重要的基于转录组学的新型生物标志物。2010年Berry等[5]利用转录组学确定了1个可用来区分活动性结核病和其他感染性疾病的393种生物标志物转录特征谱,主要构成为中性粒细胞驱动干扰素诱导型基因谱[eutrophil-driven interferon (IFN)-inducible gene profile],该研究为结核病新型疫苗和诊断性抗原的开发提供了基础数据,相关成果发表在Nature杂志上。2016年Maertzdorf等[6]鉴定出1个由GBP1、IFITM3、P2RY14和ID3 4个基因组成的转录特征谱,诊断结核病的敏感性为88%,特异性为75%。2019年Roe等[7]采用前瞻性队列研究对333名英国结核病成人接触者[人类免疫缺陷病毒(HIV)阴性]平均随访346 d,验证了该研究小组之前在南非儿童人群中[8]筛选出来的3个基因(BATF2、GBP5和SCARF1)转录特征谱的结核病风险预测性能,发现该转录特征谱的阳性预测值和阴性预测值分别为50.0%和99.3%,远高于同一队列外周血IGRAs 5.6%的阳性预测值。
研究者们也将转录组学新型生物标志物应用于儿童结核病的诊断和风险预测。2014年Anderson等[9]采用前瞻性队列研究在非洲疑似肺结核患儿中确定了含51种生物标志物的转录特征谱,其诊断敏感性和特异性分别为82.9%和83.6%,该成果发表在N Engl J Med杂志。2016年Zak等[8]采用前瞻性队列研究对6 363例南非LTBI青少年随访2年,在46例最终发展为活动性肺结核的儿童和107例健康对照儿童全血中确定了1个含16种基因的风险相关转录特征谱;经在肺结核患者家庭接触者中验证,该特征谱的敏感性和特异性分别为53.7%和82.8%,其中由BATF2、GBP5和SCARF1 3个基因组成的转录特征谱阳性预测值为23%,远高于IGRAs或结核菌素皮肤试验,该成果发表在Lancet杂志。综上,Roe等[7]和Zak等[8]针对发病风险最大的结核病接触者,分别从成人和儿童角度提供了结核病接触者疾病风险分级方面的数据,对结核病的早期诊治具有重要意义。以上数据的总结见表1。
新型生物标志物在结核病诊断中的应用
Application of new biological biomarkers in the diagnosis of tuberculosis
新型生物标志物在结核病诊断中的应用
Application of new biological biomarkers in the diagnosis of tuberculosis
| 文献来源 | 新型生物标志物 | 检测技术 | 标本类型 | 敏感性 | 特异性 | 诊断 | |
|---|---|---|---|---|---|---|---|
| 1.转录组学 | |||||||
| 2010,Berry等[5] | 中性粒细胞驱动干扰素诱导型基因谱(393种基因) | Illumina microarray; RT-qPCR | 成人血 | 61.67%a | 93.75%a | 结核病、LTBI和健康对照 | |
| 94.12%b | 96.67%b | ||||||
| 2016,Maertzdorf等[6] | GBP1、IFITM3、P2RY14和ID3 | RT-qPCR | 成人血 | 88% | 75% | 结核病和健康对照 | |
| 2020,Roe等[7] | BATF2、GBP5和SCARF1 | Illumina RNA sequencing | 成人血 | PPV 50% | NPV 99.3% | 结核病 | |
| 2016,Zak等[8] | 含16种基因的转录特征谱 | Illumina RNA sequencing | 儿童血 | 66.1%a | 80.6%a | 结核病和健康对照 | |
| 53.7%b | 82.8%b | ||||||
| 2014,Anderson等[9] | 含51种生物标志物的转录特征谱 | Illumina microarray | 儿童血 | 82.9% | 83.6% | 结核病和其他肺部疾病 | |
| 2.蛋白质组学 | |||||||
| 2016,Chegou等[11] | 七宿主血清蛋白标志物 | Multiplex cytokine platform | 成人血 | 93.8% | 73.3% | 结核病和其他肺部疾病 | |
| 2017,de Groote等[12] | 六宿主血清蛋白标志物 | SOMAscan | 成人血 | 90% | 80% | 结核病 | |
| 2017,Yoon等[13] | C反应蛋白 | POC fluorescent scanner | 成人血 | 89% | 72% | HIV阳性结核病 | |
| 2017,Li等[16] | XRCC4、PCF11、SEMA4A和ATP11A组合 | RT-qPCR | 儿童血 | 未报道 | 未报道 | 结核病和LTBI | |
| 2019,Penn-Nicholson等[17] | 五标志物组合 | SOMAscan | 儿童血 | AUC 0.96a;AUC 0.66b | 结核病和LTBI | ||
| 三标志物组合 | AUC 0.89a;AUC 0.65b | ||||||
| 2019,Albuquerque等[18] | CRP、MMP-7和LBP组合 | ELISA;Luminex technology | 儿童血 | 准确度87.5% | 肺结核和肺外结核 | ||
| 3.代谢组学 | |||||||
| 2012,Weiner等[20] | 含20个代谢产物的组合 | GC-MS | 成人血 | 未报道 | 未报道 | 结核病、LTBI和健康对照 | |
| 2018,Collins等[21] | 三代谢产物组合 | LC-MS | 成人血 | AUC 0.97 | 结核病 | ||
| 2019,Yi等[22] | 四代谢产物组合 | LC-MS | 成人血 | 86.2% | 85.2% | 结核病治愈、治疗、未治疗和健康对照 | |
| 2016,Sun等[27] | L-缬氨酸、丙酮酸和三甲铵乙内酯组合 | NMR | 儿童血 | 82.4% | 83.9% | 结核病 | |
| 2015,Mason等[28] | 含16个代谢产物的组合 | NMR | 儿童脑脊液 | 未报道 | 未报道 | 结核性脑膜炎和对照 | |
| 2017,Mason等[29] | 丙氨酸、天冬酰胺、甘氨酸、赖氨酸和脯氨酸组合 | GC-MS | 儿童脑脊液 | 未报道 | 未报道 | 结核性脑膜炎、非脑膜炎 | |
| 4.宿主microRNA | |||||||
| 2017,Cui等[36] | miR-769-5p、miR-320a和miR-22-3p | Illumina sequencing;RT-qPCR | 成人血 | AUC 0.692~0.970 | 肺结核和对照 | ||
| 2018,Barry等[37] | miR-29a、miR-99b、miR-21、miR-146a和miR-652组合 | RT-qPCR | 成人血 | 94% | 88% | 肺结核和对照 | |
| 2018,Duffy等[38] | 含47个miRNAs的风险相关血清特征谱 | RT-qPCR | 成人血 | AUC 0.74 | 结核病和对照 | ||
| 2013,Kleinsteuber等[39] | miR-26a、miR-29a和miR-142-3p | RT-qPCR | 儿童血 | 未报道 | 未报道 | 结核病、LTBI | |
| 2015,Wang等[40] | miRNA-31 | RT-qPCR | 儿童血 | 98.5% | 86.7% | 结核病和对照 | |
| 2016,Zhou等[41] | 含8个miRNAs的组合 | Agilent miRNA microarray;RT-qPCR | 儿童血 | 95.8% | 100% | 结核病和对照 | |
注:POC:床旁;RT-qPCR:反转录定量聚合酶链反应;LTBI:结核潜伏感染;PPV:阳性预测值;NPV:阴性预测值;AUC:受试者工作特征曲线下面积;ELISA:酶联免疫吸附实验;GC-MS:气相色谱质谱;LC-MS:液相色谱质谱;NMR:核磁共振光谱;a测试组;b验证组 POC:point-of-care;RT-qPCR:reverse transcription quantitative polymerase chain reaction;LTBI:latent tuberculosis infection;PPV:positive predictive value;NPV:negative predictive value;AUC:area under the receiver operating characteristic curve;ELISA:enzyme-linked immune sorbent assay;GC-MS:chromatography mass spectrometry;LC-MS:liquid chromatography mass spectrometry;NMR:nuclear magnetic resonance spectroscopy;atest group;bvalidation group
上述多数所确定的转录特征谱准确度>80%,具有很好的应用前景。但有些研究具有局限性,如采用的是回顾性病例对照研究、处于Ⅱ期诊断试验研究阶段、属于小样本研究以及在一个地点进行的研究,因此需要研究者们采用前瞻性、大样本量的研究方法并在多个地点进行验证。转录组学可从全局的角度研究基因的功能和结构,其所利用的技术已经发展到比较成熟的阶段,但在高负担和资源受限的国家或地区,研究者们需要考虑建立快速检测平台及进行实验人员技术培训所带来的设备和人力的问题,以及应用实验技术,如RNA测序和RT-qPCR验证所带来的成本问题等。
由于IGRA无法区分LTBI和活动性结核病,该诊断方法并不适合在结核潜伏感染率高的环境中应用,因此急需开发可用于结核病诊断的新型MTB抗原。研究者们虽已鉴定出多个抗原和宿主标志物[10],但这些试验一般是基于抗原过夜孵育的体外刺激试验,不适用于床旁的快速诊断,因此研究者开发了基于蛋白组学的新型生物标志物。目前可利用的技术包括蛋白质微阵列(芯片)、SOMAscan检测技术等。
针对成人结核病,研究者们发现了一些基于蛋白组学的新型生物标志物。2016年Chegou等[11]利用前瞻性研究鉴定出一个可诊断结核病的七宿主血清蛋白标志物[包括C反应蛋白(C-reactive protein,CRP)、转甲状腺素蛋白、γ-干扰素等],敏感性和特异性分别为93.8%和73.3%。2017年de Groote等[12]利用SOMAscan技术在7个结核病流行国家的1 470份血清样品中,鉴定出一个可诊断结核病的六宿主血清蛋白标志物(包括SYWC、丝氨酸蛋白酶抑制剂、补体C9等),敏感性和特异性分别为90%和80%。2017年Yoon等[13]在Lancet Infect Dis杂志发表了一篇前瞻性研究,针对已开始抗反转录病毒治疗的1 177例乌干达获得性免疫缺陷综合征患者,评价了一项床旁CRP(point-of-care-Creactive protein,POC-CRP)在筛查HIV感染者患活动性结核病方面的性能,该试验的敏感性为89%,特异性为72%。作者指出,CRP作为单一标志物是一种方便有效的HIV感染者结核病筛查工具,其对于及早发现HIV感染者的结核感染和提高结核病的预防治疗效果非常有意义。另外研究者还针对痰标本,发现了一些结核病患者特有的蛋白组学特征[14,15]。
针对儿童结核病患者,2017年首都医科大学附属北京儿童医院的申阿东课题组在19例活动性结核病患儿、16例LTBI儿童、17例炎性疾病对照患儿和20例健康对照儿童中筛选出了122种血浆差异蛋白;经验证发现X射线修复交叉互补蛋白-4(X-ray repair cross-complementing protein-4,XRCC4)、PCF11、信号素4A(semaphorin-4A,SEMA4A)和腺苷三磷酸酶11A(ATP11A)4蛋白组合在LTBI向活动性结核病的转化过程中发挥作用[16]。2019年Penn-Nicholson等[17]基于前期研究成果[8],对6 363例南非LTBI青少年随访2年,在46例最终发展为活动性肺结核的儿童和106例健康对照儿童中筛选出361种血浆差异蛋白,确定了1个五标志物[B细胞抗原受体复合物相关蛋白α链CD79A、神经细胞黏附分子(neuronal cell-adhesion mo-lecule,NrCAM)、胰岛素样生长因子结合蛋白-2(insulin-like growth factor-binding protein 2,IGFBP-2)、基质重塑相关蛋白-7(matrix-remodeling associated 7 protein,MXRA-7)和补体C9]和1个三标志物[补体C1q肿瘤坏死因子相关蛋白3(complement C1q tumor necrosis factor-related protein 3,C1qTNF3)、肌酸激酶MB亚型(creatine kinase type M/type B,CK-MB)和补体C9]组合;经在肺结核患者家庭接触者中验证,发现这2种组合均可较好地预测结核病家庭接触儿童1年内进展为结核病的风险,该研究首次从风险预测方面描述了结核病患者的蛋白组学特征。另外,Albuquerque等[18]发现,血浆中CRP、基质金属蛋白酶7(matrix metalloproteinase-7,MMP-7)和脂多糖结合蛋白(lipopolysaccharide-binding protein,LBP)等介质水平是鉴别儿童结核病不同临床疾病状态(如肺结核和肺外结核)的有效生物标志物。以上数据的总结见表1。
蛋白组学针对的是生物所能表达的所有蛋白质,它是寻找疾病分子标记和药物靶标最有效的方法之一。但蛋白质组学难以像基因组学一样形成比较一致的研究方法,有时蛋白组学和转录组学数据的重合度并不高,所以研究者们越来越重视蛋白质组学与其他学科的整合交叉研究。如有研究者在结核病中开展了转录组学和蛋白组学相结合的研究。2017年Scriba等[19]采用前瞻性研究方法比较了44例最终发展为活动性肺结核的青少年和106例健康对照儿童的全血RNA测序数据和血浆蛋白组学数据,发现在结核病诊断前的18个月,患儿的Ⅰ/Ⅱ型干扰素信号转导和补体级联反应升高,辅助性T淋巴细胞17(Th17)反应被抑制,该研究展示了机体进展为结核病之前所表现的一系列炎性反应变化和免疫学改变,对促进转录组学和蛋白组学在结核病诊治中的应用具有重要意义。
代谢组学是继基因组学和蛋白质组学之后发展起来的一门系统生物学,通过对某一生物或细胞在一特定生理时期内所有低分子量代谢产物进行定性和定量分析,进而判断生物体的病理生理状态、找出可能相关的生物标志物,为疾病预警提供相关信号。目前可利用的平台包括磁共振光谱[nuclear magnetic resonance (NMR) spectroscopy]、气相色谱飞行时间质谱(gas chromatography time-of-flight mass spectrometry,GCTOFMS)、液相色谱高分辨质谱(liquid chromatography high-resolution mass spectrometry,LC-HRMS)和超高效液相色谱-电喷雾离子化四极杆飞行时间质谱(ultrahigh-performance liquid chromatography-electrosprayionisation-quadrupole time-of-flight mass spectrometry,UHPLC-ESIQTOFMS)等。
针对成人结核病患者,2012年Weiner等[20]确定了1个可有效区分活动性结核病的二十代谢产物标志物组合,为结核病的代谢组学生物标志物研究奠定了基础。2018年Collins等[21]则确定了1个可区分活动性肺结核病的MTB三代谢产物组合[磷脂酰甘油(phosphatidylglycerol,PG)、溶血磷脂酰肌醇(lysophosphatidylinositol,Lyso-PI)和单酰化磷脂酰肌醇甘露糖苷(acylphosphatidylinositol mannoside,Ac1PIM1)],受试者工作特征曲线(ROC曲线)下面积(AUC)为0.97。2019年Yi等[22]建立了1个基于L-组氨酸、花生四烯酸、胆绿素和L-半胱氨酸-谷胱甘肽二硫化物4种代谢产物的结核病治愈诊断模型,该模型的AUC为0.909,敏感性为86.2%,特异性为85.2%。另外,研究者们还在脑脊液[23]、尿液[24]及胸腔积液[25]等其他标本中进行了代谢组学新型生物标志物的研究。将转录组学和代谢组学结合则可更准确地预测LTBI到活动性结核病的进展[26]。
将代谢组学应用于儿童结核病诊断方面的研究较少。2016年Sun等[27]筛选出17个血浆代谢产物用于区分儿童活动性结核病;建立了基于L-缬氨酸、丙酮酸和三甲铵乙内酯3个代谢产物的诊断模型,经验证其敏感性为82.4%,特异性为83.9%,该研究为血浆代谢产物在儿童结核病诊断中的应用奠定了基础。结核性脑膜炎(tuberculosis meningitis,TBM)是儿童肺外结核病中最严重的一种疾病类型,Mason等[28,29]先后利用NMR和GC-MS平台,针对TBM和非脑膜炎患儿展开了脑脊液氨基酸代谢组学方面的研究,发现丙氨酸、天冬酰胺、甘氨酸、赖氨酸和脯氨酸5种氨基酸水平在TBM患儿的脑脊液中显著升高,可作为诊断TBM的代谢组学生物标志物。上述研究为TBM患儿的早期诊断、早期治疗和临床治疗效果评估提供了研究数据。结果见表1。
上述多数研究采用的是病例对照研究,因此这些候选代谢物在结核病中的诊断潜力仍需大规模验证。与前面2种组学相比,代谢组学的发展较为滞后,它主要研究生物样品中代谢物谱的变化规律,其放大了基因和蛋白表达的微小变化,检测也更为容易。随着结核病中越来越多的代谢组学生物标志物被发现,这些代谢产物涉及葡萄糖、脂肪和氨基酸等多个代谢途径,让研究者们对机体感染MTB后产生的宿主代谢反应及MTB胞内存活机制也有了进一步深入的了解。
miRNA是一类存在于真核生物中长21~23个核苷酸的非编码单链RNA分子。miRNA通过与靶mRNA结合,在转录后水平降解mRNA或抑制mRNA的翻译从而调控基因的表达。由曼彻斯特大学研究人员开发的miRBase数据库(http://www.mirbase.org/)已收录了来自200多个物种的38 589个miRNAs信息(Release 22.1:October 2018)。人类外周血中存在着大量的miRNAs,具有稳定性高、易于检测的特点,miRNA分子作为新型生物标志物已经在临床多种疾病(如癌症、心脏疾病、糖尿病及各种感染性疾病)的诊断、发生发展及预后等方面显现出良好的应用潜力[30,31,32,33,34,35]。目前,研究者们主要利用测序、微阵列(芯片)或RT-qPCR的方法检测宿主miRNA。
针对成人结核病患者,2017年Cui等[36]发现血浆中的miR-769-5p、miR-320a和miR-22-3p具有较好的活动性结核病诊断价值,AUC在0.692~0.970;另外该研究还发现耐药结核病患者的miR-320a水平低于敏感结核病患者(AUC=0.882),其可能是耐药结核病的生物标志物之一。2018年Barry等[37]发现,miR-29a、miR-99b、miR-21、miR-146a和miR-652组合诊断结核病的敏感性为94%,特异性为88%,AUC为0.976;另外,部分miRNAs水平可在治疗成功后恢复到基线水平,因此在治疗效果预测方面也具有较好的应用价值。Duffy等[38]则针对54例非洲结核病家庭接触者开展了一项随访2年的前瞻性研究,确定了一组含47个miRNAs的风险相关血清特征谱,其预测结核病家庭接触者6个月和24个月内进展为活动性结核病的AUC值为0.74和0.66。
目前,将miRNA分子用于儿童结核病方面的研究较少。2013年Kleinsteuber等[39]开展了一项前瞻性队列研究,发现与LTBI儿童相比,结核病患儿全血中的miR-26a、miR-29a和miR-142-3p表达较低,其在治疗及恢复期逐渐增加,可考虑将miRNA作为结核病干预策略中的新靶点。2015年Wang等[40]研究了中国儿童肺结核患者外周血单个核细胞中miRNA-31的诊断价值,发现肺结核患儿的miRNA-31表达明显低于健康儿童,其表达水平与血清白细胞介素(IL)-6呈负相关,诊断敏感性为98.5%,特异性为86.7%。2016年Zhou等[41]利用微阵列(芯片)的方法在活动性肺结核患儿外周血中筛选出了29个与对照组表达不同的miRNAs,其中14个miRNAs关系密切;经验证发现,miR-150、miR-146a和miR-125b等8个miRNAs组合具有较高的结核病诊断价值,其中诊断价值最高的单个miRNA为miR-150,见表1。以上研究多数属于小样本病例对照研究且缺乏队列验证实验,因此限制了上述研究结果的进一步应用。
在结核病领域应用新型生物标志物进行疾病的诊断,其具有的巨大优势是检测不依赖于痰标本,这对于无法咳痰的儿童肺结核和肺外结核病具有非常重要的意义。将新型生物标志物应用于结核病的诊断中,仍需面临或解决一些问题。首先,面对不同学者所确定的如此众多新型生物标志物及其特征谱,如何进行选择或将其转换为一个全球通用的含最少生物标志物的特征谱,是目前研究者面临的重要问题,这也在一定程度上降低了新型生物标志物的应用价值。但研究者一直在努力解决这一问题,如研究者利用美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)的GEO(Gene Expression Omnibus database)数据库,开展了基因表达特征谱已有数据的再发掘、分析和验证等工作。2016年Sweeney等[42]发表在Lancet Respir Med杂志上的研究,利用14个已有的数据集合(覆盖了10个国家2 572份样本),筛选出1个对活动性肺结核具有高度诊断价值的3基因(GBP5、DUSP3和KLF2)转录特征谱并进行了验证,结果显示该组合可将活动性肺结核患者与健康对照者、LTBI及其他疾病患者(AUC分别为0.90、0.88和0.84)区别诊断开来,且不受HIV感染状况、细菌耐药性或卡介苗接种的干扰。该转录特征谱良好的诊断性能也被Warsinske等[43]的系统比较研究所证实。
其次,已建立的生物标志物特征谱应用于其他种族人群中是否同样具有较好的诊断或预测能力,也是研究者们需要解决的问题。2018年Leong等[44]利用基因差异表达分析和聚类分析等方法检测了已有的7个数据集合对南印度的28例活动性肺结核患者和16例LTBI疾病状态的预测能力,发现这些不同的转录特征谱在南印度人群中也显示较强的预测能力(AUC=0.900 7~0.987 9)。
此外,研究者在开发新型生物标志物时,还需要面临一些挑战,如商业压力可能会妨碍最佳生物标记的选择,市场竞争是阻碍合作的重要因素;另外,在广泛的浓度范围内或在非常低的浓度下测量多个标志物,检测平台的成本很高,并不适合在结核病高负担的国家开展[45]。因此,开发新型生物标志物虽具有很好的远期应用前景,但需要有持续的资金支持以应对市场竞争压力和检测成本压力。
总之,基于转录组学、蛋白质组学和代谢组学及针对miRNA的新型生物标志物研究,根据其所分析的目标产物和所用具体技术的不同,造成其各自具有相应的特点或优劣势。鉴于单一组学或技术的局限性,研究者们开始重视各个组学之间的交叉验证、组学与其他学科之间的交叉应用、各种技术方法之间的整合互补以及海量数据的分析整合和解释,这是新型生物标志物未来发展的方向,也是研究者们未来努力的方向。结核病根除需要依靠积极的病例发现和筛查策略,而新型生物标志物为研究指明了方向。
所有作者均声明不存在利益冲突



























