
明确导致猩红热的A族链球菌(GAS)流行型别,比较不同emm型别GAS的基因结构及基因变异性,阐明猩红热病原学的流行、进化规律。
回顾性研究。收集2016年1月1日至2018年5月31日深圳市儿童医院诊断为猩红热的儿童咽拭子标本,保存GAS菌株,进行emm基因分型,选取在分型上具有代表性的菌株进行全基因组测序,通过比较基因组学分析,描述GAS菌株基因组多态性,并基于单核苷酸多态性(SNP)位点分析构建遗传进化树,阐明菌株之间的进化关系。2组比较采用秩和检验。
在导致儿童猩红热的176株GAS分离株中,共检测到8种emm型别,emm12.0及其亚型(108/176株,61.4%)占比最高,其次为emm1.0及其亚型(53/176株,30.1%),两者共占91.5%。以GCA-900984775为参考序列进行比较基因组分析显示,GAS菌株基因组之间存在丰富的SNP和插入或缺失(InDel)多态性,emm12.0型菌株SNP数[183(163,213)个]大于emm1.0型菌株SNP数[63(54,75)个],差异有统计学意义(P<0.05);InDel分析中,emm12.0型菌株基因序列插入数[4(3,6)个]和缺失数[8(6,10)个]大于emm1.0型菌株[1(0,2)个,5(3,7)个]。以MGAS5005参考序列,基于SNP的分析结果构建进化树得出18株菌株及参考菌株处于2个分支。
引起儿童猩红热的GAS菌株型别以emm12.0型、emm 1.0型多见,不同型别菌株基因组组成之间存在差异,emm12.0型菌株存在较高的遗传多样性。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
A族链球菌(Group A Streptococcus,GAS)是导致儿童感染性疾病的病原菌之一,GAS除可引起咽炎、扁桃体炎、皮肤化脓性感染外,还可引起坏死性筋膜炎等侵袭性疾病以及风湿热、肾小球肾炎等免疫性疾病[1,2]。猩红热为GAS所致疾病中常见的病种之一,具有传染性,易引起暴发流行,且易导致链球菌中毒性休克综合征(streptococcal toxic shock syndrome,STSS)、败血症等并发症[3,4]。导致菌株显著多样性和疾病多样性的细菌遗传因素值得被关注。全基因组测序是指对物种基因组序列进行测定,用来确定生物的遗传组成。Salvà-Serra等[5]对GAS进行全基因组测序,并对基因组组分进行分析,预测到噬菌体、CRISPR系统、毒力基因及抗菌药物耐药基因,并对细菌重要基因、遗传变异、群体进化展开分析,进一步有效地为传染病诊断提供有价值的分类学和基因组学参考。本研究综合运用emm分型和基因组技术,对深圳市致猩红热GAS的基因型进行回顾性分析,明确主要流行型别,比较不同型别GAS基因变异性,阐明导致猩红热GAS的流行、进化规律,对深圳市猩红热病原学监测和防控具有一定意义。
选择深圳市儿童医院生物样本库收集保存的176株GAS分离株,分离自2016年1月1日至2018年5月31日在深圳市儿童医院门诊或住院的猩红热患儿(0~18岁)。对176株GAS分离株进行emm分型,选取具有代表性的18株GAS分离株进行全基因组测序。本研究已通过深圳市儿童医院医学伦理委员会批准(批准文号:202104702),患儿监护人均知情同意,并签署知情同意书。
18株GAS菌株选择标准:(1)来自不同年份emm1.0、emm12.0主型GAS菌株各5株;(2)各年度emm1.0和emm12.0亚型各选1株,其中emm1.22、emm12.19型分别选取来自不同年份2株;(3)各年度菌株数相同,其中2016年、2017年、2018年各6株。本研究共选取导致深圳市儿童猩红热的emm1.0型(包括亚型)GAS菌株9株,emm12.0型(包括亚型)GAS菌株9株。
通过PCR扩增及测序获得emm基因的5′端可变区序列并上传至分型数据库(https://www2.cdc.gov/vaccines/biotech/strepblast.asp)进行比对分析,确定emm型别。
基因组测序由深圳华大基因公司使用lllumina Hiseq4000系统(美国lllumina公司)完成,测序后序列使用SOAPdenovo v1.05软件组装。
(1)共有基因和特有基因:将所有待分析菌株的基因集进行聚类分析,将得到的基因集作为总基因集,提取聚类结果中每个样品都有的序列为核心基因集,特有的序列为特有基因集。(2)单核苷酸多态性(SNP)及插入或缺失(InDel)检测:SNP检测与注释基于组装结果和参考序列比对进行,首先对过滤后的序列进行组装,将组装结果与参考序列进行比对,进行SNP及基因组的某个位置上所发生的小片段序列的InDel。(3)基于SNP位点分析构建遗传进化树:按照相同顺序将所有SNP相连,获得相同长度的序列(其中一个为参考序列),作为输入文件,用TreeBeST软件的最大似然法算法构建系统进化树。
采用SPSS 21.0统计软件,符合正态分布的计量资料以 ±s表示,非正态分布计量资料以M(Q1,Q3)表示,计数资料以百分比或率表示,率的比较采用χ2检验、校正χ2检验,非正态分布计量资料比较采用秩和检验,P<0.05为差异有统计学意义。
±s表示,非正态分布计量资料以M(Q1,Q3)表示,计数资料以百分比或率表示,率的比较采用χ2检验、校正χ2检验,非正态分布计量资料比较采用秩和检验,P<0.05为差异有统计学意义。
176株GAS分离株中,emm12.0及其亚型(108/176株,61.4%)检出最高,其次为emm1.0及其亚型(53/176株,30.1%)。结果见表1。
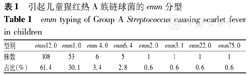
引起儿童猩红热A族链球菌的emm分型
emm typing of Group A Streptococcus causing scarlet fever in children
引起儿童猩红热A族链球菌的emm分型
emm typing of Group A Streptococcus causing scarlet fever in children
| 型别 | emm12.0 | emm1.0 | emm 4.0 | emm6.4 | emm2.0 | emm3.1 | emm22.0 | emm75.0 |
|---|---|---|---|---|---|---|---|---|
| 株数 | 108 | 53 | 6 | 5 | 1 | 1 | 1 | 1 |
| 占比(%) | 61.4 | 30.1 | 3.4 | 2.8 | 0.6 | 0.6 | 0.6 | 0.6 |
基于测序数据组装得到18株GAS基因组,由27~82个片段组成,大小为1.82~1.93 Mb,GC含量为39.03%~39.15%,编码区占基因总长的86%以上,18株GAS基因组基本特征与参考序列基因组特征相似,见表2。
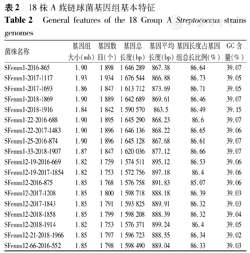
18株A族链球菌基因组基本特征
General features of the 18 Group A Streptococcus strains genomes
18株A族链球菌基因组基本特征
General features of the 18 Group A Streptococcus strains genomes
| 菌株名称 | 基因组大小(mb) | 基因数目(个) | 基因总长度(bp) | 基因平均长度(bp) | 基因长度占基因组总长比例(%) | GC含量(%) |
|---|---|---|---|---|---|---|
| SFemm1-2016-865 | 1.90 | 1 898 | 1 646 289 | 867.38 | 86.64 | 39.07 |
| SFemm1-2017-1117 | 1.93 | 1 934 | 1 676 544 | 866.88 | 86.73 | 39.05 |
| SFemm1-2017-1693 | 1.86 | 1 847 | 1 613 712 | 873.69 | 86.71 | 39.05 |
| SFemm1-2018-1869 | 1.90 | 1 889 | 1 642 689 | 869.61 | 86.46 | 39.07 |
| SFemm1-2018-1916 | 1.84 | 1 842 | 1 590 570 | 863.5 | 86.49 | 39.15 |
| SFemm1-22-2016-688 | 1.90 | 1 895 | 1 645 290 | 868.23 | 86.6 | 39.07 |
| SFemm1-22-2017-1483 | 1.90 | 1 896 | 1 646 136 | 868.22 | 86.65 | 39.06 |
| SFemm1-25-2016-874 | 1.90 | 1 896 | 1 645 128 | 867.68 | 86.61 | 39.07 |
| SFemm1-33-2018-1907 | 1.87 | 1 847 | 1 620 036 | 877.12 | 86.66 | 39.07 |
| SFemm12-19-2016-669 | 1.82 | 1 759 | 1 574 511 | 895.12 | 86.53 | 39.06 |
| SFemm12-19-2017-1854 | 1.82 | 1 753 | 1 572 756 | 897.18 | 86.4 | 39.06 |
| SFemm12-2016-875 | 1.85 | 1 768 | 1 576 758 | 891.83 | 85.07 | 39.06 |
| SFemm12-2017-1208 | 1.85 | 1 800 | 1 598 718 | 888.18 | 86.39 | 39.03 |
| SFemm12-2017-1843 | 1.85 | 1 791 | 1 593 825 | 889.91 | 86.32 | 39.03 |
| SFemm12-2018-1858 | 1.85 | 1 799 | 1 598 208 | 888.39 | 86.32 | 39.04 |
| SFemm12-2018-1914 | 1.82 | 1 753 | 1 576 371 | 899.24 | 86.4 | 39.05 |
| SFemm12-21-2018-1966 | 1.85 | 1 797 | 1 596 723 | 888.55 | 86.34 | 39.02 |
| SFemm12-66-2016-552 | 1.85 | 1 798 | 1 598 490 | 889.04 | 86.33 | 39.03 |
通过与参考序列GCA-900984775进行比对,发现18株GAS存在非常丰富的SNP。以GCA-900984775菌株为参考序列,emm1.0型SNP分析见表3。以GCA-900984775菌株为参考序列,emm12.0型SNP分析见表4。
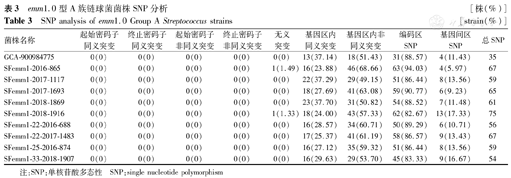
emm1.0型A族链球菌菌株SNP分析 [株(%)]
SNP analysis of emm1.0 Group A Streptococcus strains [strain(%)]
emm1.0型A族链球菌菌株SNP分析 [株(%)]
SNP analysis of emm1.0 Group A Streptococcus strains [strain(%)]
| 菌株名称 | 起始密码子同义突变 | 终止密码子同义突变 | 起始密码子非同义突变 | 终止密码子非同义突变 | 无义突变 | 基因区内同义突变 | 基因区内非同义突变 | 编码区SNP | 基因间区SNP | 总SNP |
|---|---|---|---|---|---|---|---|---|---|---|
| GCA-900984775 | 0(0) | 0(0) | 0(0) | 0(0) | 0(0) | 13(37.14) | 18(51.43) | 31(88.57) | 4(11.43) | 35 |
| SFemm1-2016-865 | 0(0) | 0(0) | 0(0) | 0(0) | 1(1.49) | 16(23.88) | 46(68.66) | 63(94.03) | 4(5.97) | 67 |
| SFemm1-2017-1117 | 0(0) | 0(0) | 0(0) | 0(0) | 0(0) | 22(37.29) | 29(49.15) | 51(86.44) | 8(13.56) | 59 |
| SFemm1-2017-1693 | 0(0) | 0(0) | 0(0) | 0(0) | 0(0) | 18(27.69) | 41(63.08) | 59(90.77) | 6(9.23) | 65 |
| SFemm1-2018-1869 | 0(0) | 0(0) | 0(0) | 0(0) | 0(0) | 23(37.70) | 31(50.82) | 54(88.52) | 7(11.48) | 61 |
| SFemm1-2018-1916 | 0(0) | 0(0) | 0(0) | 0(0) | 1(1.33) | 18(24.00) | 43(57.33) | 62(82.67) | 13(17.33) | 75 |
| SFemm1-22-2016-688 | 0(0) | 0(0) | 0(0) | 0(0) | 0(0) | 16(28.57) | 34(60.71) | 50(89.29) | 6(10.71) | 56 |
| SFemm1-22-2017-1483 | 0(0) | 0(0) | 0(0) | 0(0) | 0(0) | 17(25.37) | 41(61.19) | 58(86.57) | 9(13.43) | 67 |
| SFemm1-25-2016-874 | 0(0) | 0(0) | 0(0) | 0(0) | 0(0) | 16(27.12) | 35(59.32) | 51(86.44) | 8(13.56) | 59 |
| SFemm1-33-2018-1907 | 0(0) | 0(0) | 0(0) | 0(0) | 0(0) | 16(29.63) | 29(53.70) | 45(83.33) | 9(16.67) | 54 |
注:SNP:单核苷酸多态性 SNP:single nucleotide polymorphism
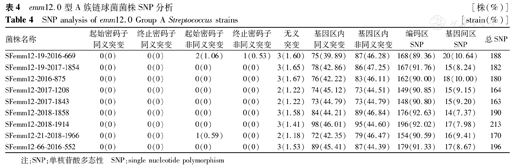
emm12.0型A族链球菌菌株SNP分析 [株(%)]
SNP analysis of emm12.0 Group A Streptococcus strains [strain(%)]
emm12.0型A族链球菌菌株SNP分析 [株(%)]
SNP analysis of emm12.0 Group A Streptococcus strains [strain(%)]
| 菌株名称 | 起始密码子同义突变 | 终止密码子同义突变 | 起始密码子非同义突变 | 终止密码子非同义突变 | 无义突变 | 基因区内同义突变 | 基因区内非同义突变 | 编码区SNP | 基因间区SNP | 总SNP |
|---|---|---|---|---|---|---|---|---|---|---|
| SFemm12-19-2016-669 | 0(0) | 0(0) | 2(1.06) | 1(0.53) | 3(1.60) | 75(39.89) | 87(46.28) | 168(89.36) | 20(10.64) | 188 |
| SFemm12-19-2017-1854 | 0(0) | 0(0) | 0(0) | 0(0) | 3(1.65) | 78(42.86) | 86(47.25) | 167(91.76) | 15(8.24) | 182 |
| SFemm12-2016-875 | 0(0) | 0(0) | 0(0) | 0(0) | 3(1.67) | 76(42.22) | 83(46.11) | 162(90.00) | 18(10.00) | 180 |
| SFemm12-2017-1208 | 0(0) | 0(0) | 0(0) | 0(0) | 2(1.22) | 74(45.12) | 73(44.51) | 149(90.85) | 15(9.15) | 164 |
| SFemm12-2017-1843 | 0(0) | 0(0) | 0(0) | 0(0) | 2(1.22) | 73(44.79) | 73(44.79) | 148(90.80) | 15(9.20) | 163 |
| SFemm12-2018-1858 | 0(0) | 0(0) | 0(0) | 0(0) | 3(1.58) | 84(44.21) | 89(46.84) | 176(92.63) | 14(7.37) | 190 |
| SFemm12-2018-1914 | 0(0) | 0(0) | 0(0) | 0(0) | 3(1.41) | 98(46.01) | 95(44.60) | 196(92.02) | 17(7.98) | 213 |
| SFemm12-21-2018-1966 | 0(0) | 0(0) | 1(0.59) | 0(0) | 2(1.18) | 72(42.35) | 79(46.47) | 154(90.59) | 16(9.41) | 170 |
| SFemm12-66-2016-552 | 0(0) | 0(0) | 0(0) | 0(0) | 3(1.53) | 89(45.41) | 87(44.39) | 179(91.33) | 17(8.67) | 196 |
注:SNP:单核苷酸多态性 SNP:single nucleotide polymorphism
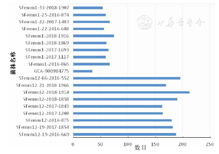
emm12.0型菌株SNP数[183(163,213)个]大于emm1.0型菌株SNP数[63(54,75)个],差异有统计学意义(P<0.05),见图1。

通过与参考序列GCA-900984775进行比对,InDel类型统计和InDel引起的突变类型分见表5、表6。InDel分析中,emm12.0型菌株插入数[4(3,6)]个,缺失数[8(6,10)]个大于emm1.0型菌株插入数[1(0,2)]个,缺失数[5(3,7)]个,见图2。
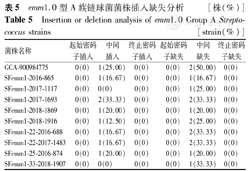
emm1.0型A族链球菌菌株插入缺失分析 [株(%)]
Insertion or deletion analysis of emm1.0 Group A Streptococcus strains [strain(%)]
emm1.0型A族链球菌菌株插入缺失分析 [株(%)]
Insertion or deletion analysis of emm1.0 Group A Streptococcus strains [strain(%)]
| 菌株名称 | 起始密码子插入 | 中间插入 | 终止密码子插入 | 起始密码子缺失 | 中间缺失 | 终止密码子缺失 |
|---|---|---|---|---|---|---|
| GCA-900984775 | 0(0) | 1(25.00) | 0(0) | 0(0) | 2(50.00) | 0(0) |
| SFemm1-2016-865 | 0(0) | 1(16.67) | 0(0) | 0(0) | 1(16.67) | 0(0) |
| SFemm1-2017-1117 | 0(0) | 0(0) | 0(0) | 0(0) | 1(25.00) | 0(0) |
| SFemm1-2017-1693 | 0(0) | 2(33.33) | 0(0) | 0(0) | 2(33.33) | 0(0) |
| SFemm1-2018-1869 | 0(0) | 1(20.00) | 0(0) | 0(0) | 1(20.00) | 0(0) |
| SFemm1-2018-1916 | 0(0) | 1(12.50) | 0(0) | 0(0) | 2(25.00) | 0(0) |
| SFemm1-22-2016-688 | 0(0) | 1(16.67) | 0(0) | 0(0) | 2(33.33) | 0(0) |
| SFemm1-22-2017-1483 | 0(0) | 1(16.67) | 0(0) | 0(0) | 2(33.33) | 0(0) |
| SFemm1-25-2016-874 | 0(0) | 1(20.00) | 0(0) | 0(0) | 1(20.00) | 0(0) |
| SFemm1-33-2018-1907 | 0(0) | 0(0) | 0(0) | 0(0) | 1(33.33) | 0(0) |
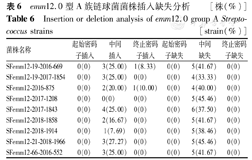
emm12.0型A族链球菌菌株插入缺失分析 [株(%)]
Insertion or deletion analysis of emm12.0 group A Streptococcus strains [strain(%)]
emm12.0型A族链球菌菌株插入缺失分析 [株(%)]
Insertion or deletion analysis of emm12.0 group A Streptococcus strains [strain(%)]
| 菌株名称 | 起始密码子插入 | 中间插入 | 终止密码子插入 | 起始密码子缺失 | 中间缺失 | 终止密码子缺失 |
|---|---|---|---|---|---|---|
| SFemm12-19-2016-669 | 0(0) | 3(25.00) | 1(8.33) | 0(0) | 5(41.67) | 0(0) |
| SFemm12-19-2017-1854 | 0(0) | 3(25.00) | 0(0) | 0(0) | 4(33.33) | 0(0) |
| SFemm12-2016-875 | 0(0) | 2(20.00) | 1(10.00) | 0(0) | 4(40.00) | 0(0) |
| SFemm12-2017-1208 | 0(0) | 0(0) | 0(0) | 0(0) | 5(45.46) | 0(0) |
| SFemm12-2017-1843 | 0(0) | 4(25.00) | 0(0) | 0(0) | 6(37.50) | 0(0) |
| SFemm12-2018-1858 | 0(0) | 2(16.67) | 0(0) | 0(0) | 5(41.67) | 0(0) |
| SFemm12-2018-1914 | 0(0) | 1(7.69) | 0(0) | 0(0) | 5(38.46) | 0(0) |
| SFemm12-21-2018-1966 | 0(0) | 3(27.27) | 0(0) | 0(0) | 5(45.46) | 0(0) |
| SFemm12-66-2016-552 | 0(0) | 3(25.00) | 0(0) | 0(0) | 5(41.67) | 0(0) |
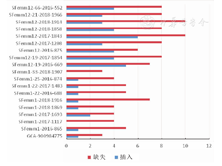

以化脓性链球菌改为A族链球菌菌株MGAS5005基因组为参考序列,18株菌株为比对菌株基于SNP的分析结果构建进化树,显示参考菌株与18株比对菌株分别位于2个进化分支上,MGAS5005参考序列与emm 1.0型菌株有更近的进化距离,结果见图3。
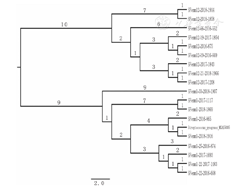

注:SNP:单核苷酸多态性 SNP:single nucleotide polymorphism
研究表明,多克隆emm12.0和emm1.0型别是2011年中国流行性猩红热相关GAS菌株的主要emm型别[6]。2014年英国猩红热疫情的流行病学监测显示,区域性暴发是由多种(emm3.0、emm12.0、emm1.0和emm4.0)基因类型及系统发育谱系引起的,单基因ssa与猩红热患病率显著增加相关[7]。本研究为回顾性研究,分析了2016年至2018年深圳市儿童医院生物样本库收集保存的176株猩红热患儿来源的GAS分离株,emm分型结果显示,深圳地区致猩红热患儿GAS的型别主要为emm12.0型及emm1.0型,这与之前国内报道[8]的流行型别相符。另有一些研究发现[9],近10年来,emm1.0型的比例有上升趋势,emm12.0型则有下降趋势。但总体来讲,emm12.0型和emm1.0型GAS菌株依然是导致深圳地区儿童猩红热的主要菌株,这为深圳地区猩红热病原学监测和防控提供了参考意义,也为未来疫苗研究提供依据。
本研究对代表性菌株进行全基因组测序发现,猩红热儿童来源的emm1.0和emm12.0型GAS菌株大小为1.82~1.93 Mb,GC含量为39.03%~39.15%,与参考序列GCA-900984775相似。对不同型别菌株基因组组成之间进行全基因组序列比较发现,不同型别之间存在遗传变异,其中emm12.0型GAS菌株SNP及InDel数均多于emm1.0型GAS菌株,说明其存在较高的遗传多样性。基因组变异、重组、移动元件横向转移可能是导致GAS菌株遗传多态性的内在动力[10]。一些研究认为,emm12.0型GAS引起猩红热的能力在流行病学上与新的噬菌体的存在有关,包括编码分泌的超抗原SSA和SpeC的噬菌体ΦHKU.vir和DNaseSpd1[11]。噬菌体在GAS种群形成、遗传转移和毒力中起关键作用,可通过消除易感血清型来改变种群分布,也可通过转导作为毒力基因和抗生素耐药性遗传转移的关键介质[12]。GAS前噬细胞已被证明是该病原体毒力的主要促成因素[13],且噬菌体具有携带毒力基因、全基因组调控等作用。本研究未进一步对毒力基因及噬菌体等遗传元件进行分析,本课题组另一项研究[14]对中国儿童emm1.0和emm12.0菌株进行全基因组测序和系统发育分析发现,这些菌株对大环内酯类抗生素和四环素的耐药率和耐药水平很高,主要表达ermB和tetM,vanuG和毒力基因的分布与emm类型有关。先前报道的重要噬菌体,如φHKU16.vir、φHKU488.vir、Φ5005.1、Φ5005.2和Φ5005.3编码链球菌毒素以及整合性结合元件(integrative conjugative elements,ICEs),如ICE-emm12和ICE-HKU397编码大环内酯类和四环素耐药基因,在深圳的emm1.0或emm12.0克隆群中存在。本研究测序的基因组数据为中国GAS分子监测提供重要参考,但未进一步对SNP对应的毒力基因、耐药基因及噬菌体作进一步分析,未来需继续关注以明确菌株遗传多样性对于疾病分布及转归的影响。
全基因组测序技术对于发病率增加的疾病,从病原菌遗传进化的角度作出了很好解释[15],也增进了对新遗传元件的出现和与疾病相关的基因表达的相互作用的认识。随着国际上GAS基因组序列数据库的扩展,除了为GAS种群基因组学提供新的见解外,还能够生成一个比较基因组学的迭代框架[16],全基因组测序仍然是绘制新兴GAS菌株图谱的重要工具。但仍需进一步的研究来描述GAS毒力基因的致病机制,并揭示监测和治疗这些疾病的新策略。
所有作者均声明不存在利益冲突



























