
探讨坐骨神经损伤后长链非编码RNA(LncRNA)差异表达的基因,以及LncRNA MX1对大鼠雪旺细胞迁移、增殖能力的影响。
选取10周龄无特定病原体级雄性SD大鼠24只,随机分为0 d(T0)、3 d(T1)、7 d(T2)和14 d(T3)4组,每组6只,建立坐骨神经损伤动物模型。分别于模型建立后第0、3、7、14天取相应组大鼠坐骨神经损伤处的残端组织提取RNA,制备RNA基因芯片进行Heatmap聚类分析,筛选出各个时间点发生差异表达的基因,并选择其中差异表达显著的基因LncRNA MX1。培养iCell-r030大鼠雪旺细胞,分为对照组、Control siRNA组(siRNA组)和LncRNA MX1 siRNA组(siRNA-MX1组),使用相应试剂进行转染。转染后采用实时荧光定量聚合酶链反应(qRT-PCR)检测3组雪旺细胞的LncRNA MX1mRNA表达情况,采用Transwell小室检测雪旺细胞的迁移能力,采用5-乙炔基-2'-脱氧尿苷(EDU)实验检测雪旺细胞的增殖能力。
各组大鼠均造模成功,实验过程中无大鼠死亡。大鼠损伤坐骨神经组织LncRNA差异基因Heatmap聚类分析的热图显示,与T0组比:T1组共3 066个LncRNA基因发生差异性表达,其中1 634个LncRNA基因表达上调,1 432个LncRNA基因表达下调;T2组共2 498个LncRNA基因发生差异性表达,其中1 634个LncRNA基因表达上调,864个LncRNA基因表达下调;T3组3 567个LncRNA基因发生差异性表达,其中有1 643个LncRNA基因表达上调,1 924个 LncRNA基因表达下调。各组差异表达的LncRNA基因中LncRNA MX1的差异倍数数值较大,差异表达显著。大鼠雪旺细胞qRT-PCR结果显示,转染LncRNA MX1 siRNA后,siRNA-MX1组LncRNA MX1的相对表达量为1.0±0.2,低于对照组的2.3±0.2和siRNA组的2.2±0.2,差异有统计学意义(F=78.47,P<0.001);而对照组和siRNA组组间差异无统计学意义(P>0.05)。迁移、增殖试验结果显示,siRNA-MX1组细胞迁移数量为(24.1±4.2)个、EDU阳性细胞与DAPI阳性细胞的比率为27.5%±2.8%,低于对照组的(50.3±7.8)个、44.1%±7.2%和siRNA组的(49.2±6.2)个、41.8%±7.0%,差异均有统计学意义(F=93.15、121.26,P值均<0.001);而对照组和siRNA组间差异均无统计学意义(P值均>0.05)。
大鼠坐骨神经损伤后LncRNA MX1差异表达显著,下调LncRNA MX1在大鼠雪旺细胞中的表达可显著降低细胞的迁移、增殖能力。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
周围神经损伤(peripheral nerve injury,PNI)是目前临床上常见的疾病之一,患者常表现为持续性的运动和感觉障碍,对日常生活和工作带来巨大的影响[1]。当下手术治疗PNI的主流方法是采用显微外科技术进行神经外膜的缝合;而对于范围较大的周围神经病灶,常常选择利用患者自体神经进行移植修复,由于这种操作方式会一定程度的损伤患者自体神经,因此其临床使用受到较大的限制[2]。
雪旺细胞作为周围神经系统(peripheral nervous system,PNS)中重要的神经胶质细胞,其高度可塑性可以极大地影响周围神经损伤后的再生能力[3]。长链非编码RNA(long non-coding RNA,LncRNA)是长度大于200个核苷酸且没有编码蛋白质功能的核酸序列[4]。随着RNA测序技术的发展,研究人员发现LncRNA在表观遗传学和翻译水平上调控基因表达,调节多种病理生理过程[5]。然而,有关LncRNA在PNI后雪旺细胞表型调节中的特定作用的研究较少。Pan等[6]通过微阵列分析测量了PNI后不同时间点的坐骨神经远端节段中LncRNA的表达,并预测有其中LncRNA ENSMUSG00000075555、NONMMUG036610和NONMMVG014942等可能参与了PNI相关的生物学过程,但对LncRNA的具体功能和机制没有进一步探索。本研究中,通过构建大鼠坐骨神经损伤模型,提取大鼠坐骨神经损伤后不同时间点的差异基因进行汇总、分析和筛选,探讨LncRNA调控雪旺细胞促进坐骨神经损伤修复的分子机制,旨在为临床PNI患者的治疗和康复提供新的思路。
选用10周龄无特定病原体级雄性SD大鼠24只,体质量323~376 g,由徐州医科大学动物实验中心提供,实验动物许可证号:SYXK(苏)2020-0048。采用数字表法随机将SD大鼠分为4组,分别为PNI后0 d(T0)、3 d(T1)、7 d(T2)和14 d(T3)组,每组6只。本实验所有操作均遵循实验动物使用的相关规定。
PCR引物、总RNA提取试剂盒、Lipofectamine转染试剂为美国Invitrogen公司产品;Control siRNA和LncRNA MX1 siRNA为美国赛默飞公司产品;CWBIO Ultra SYBR Mixture试剂盒、Prime-Script RT Mixture试剂盒、iCell-r030大鼠雪旺细胞为上海生物工程有限公司产品;PrimeScrite RT reagent Kit with gDNA Eraser试剂盒为大连TaKaRa公司;LncRNA MX1原位杂交试剂盒、DMEM培养基、RPMI-1640培养基为江苏凯基技术股份有限公司产品;SC培养基、4',6-二脒基-2-苯基吲哚(4',6-diamidino-2-phenylindole,DAPI)为上海碧云天生物有限公司产品;5-乙炔基-2'-脱氧尿苷(5-ethynyl-2'-deoxyuridine,EDU)溶液为北京天根生化有限公司产品。CKX53荧光显微镜、多功能光学倒置显微镜为日本Olympus公司产品;iScan芯片扫描仪为美国Illumina公司产品;医用核酸分子杂交仪为江苏迅迪仪器科技有限公司产品。
将大鼠四肢外展固定于操作台面,腹腔注射5%水合氯醛(0.9 mL/100 g)麻醉。取大鼠左下肢根部做长约2 cm切口,血管钳钝性分离显露大鼠左下肢坐骨神经,使用持针器尖端挤压此部位坐骨神经约30 s。以坐骨神经损伤处呈现半透明状、神经外膜大致完整为标准,判定坐骨神经损伤模型制备成功[7]。缝合切口,无菌纱布包扎。
T0组在建立坐骨神经损伤模型后立即取样,T1、T2、T3组分别在模型建立后第3、7、14天取样。取样方法:各组大鼠常规麻醉固定后开胸,心脏注射生理盐水处死大鼠。从原手术切口进入显露坐骨神经损伤处,并取坐骨神经残端组织约100 mg。将坐骨神经组织加入约1 mL的Trizol试剂,匀浆后转至EP管,室温下孵育5 min;加入氯仿溶液300 µL,充分混匀后室温下孵育5 min;12 000×g离心10 min后,取沉淀物即为坐骨神经组织的RNA。
取各组大鼠提取的RNA进一步纯化,使用Cy3-CTP标记Mix进行荧光标记,片段化mix芯片杂交后,采用iScan芯片扫描仪扫描记录。使用R语言将扫描得到的原始数据转换为可供识别的Data frame格式,运用AFFY 1.60.1软件(http://bioconductor.org/packages/release/bioc/html/affy.html)中的RMA算法将不同时间点的基因芯片扫描数据归一化,以减少不同时间点基因信息间的背景差异。将归一化后的数据进行样本主成分分析和样本Heatmap聚类分析,选择造模后不同时间点发生显著差异表达的LncRNA MX1进行后续实验。
取iCell-r030大鼠雪旺细胞接种于6孔板,分为对照组、Control siRNA组(siRNA组)和LncRNA MX1 siRNA组(siRNA-MX1组),每组2个复孔。将各组细胞在37 ℃、5% CO2培养箱中培养36 h,待雪旺细胞融合度达70%时进行转染。在siRNA组和siRNA-MX1组细胞中分别加入5 µL Lipofectamine试剂室温下充分混合摇匀,再分别加入Control siRNA和LncRNA MX1 siRNA试剂避光静置20 min。PBS清洗6孔板后,siRNA组和siRNA-MX1组细胞中分别加入上述试剂重复转染2次,转染时间为8、48 h。对照组操作步骤与siRNA组和siRNA-MX1组相同,每次均加入RPMI-1640培养基。
取转染后的对照组、siRNA组和siRNA-MX1组雪旺细胞,按Prime-Script RT Mixture试剂盒说明提取RNA。各组细胞提取RNA后,按照PrimeScrite RT reagent Kit with gDNA Eraser试剂盒说明书合成cDNA,反转录条件:37 ℃ 15 min,85 ℃ 15 min。目的基因引物序列见表1。将反转录所得cDNA按PCR试剂盒说明进行qRT-PCR检测。qRT-PCR反应条件:50 ℃ 2 min,95 ℃ 10 min,95 ℃ 15 s,60 ℃ 1 min,40个循环。根据PCR反应曲线得到目的基因的荧光阈值(cycle threshold,Ct),以GAPDH为内参,按照2-ΔΔCt法计算出各组雪旺细胞LncRNA MX1的相对表达量。实验重复3次,结果取平均值。

目的基因引物名称、序列和长度
目的基因引物名称、序列和长度
| 引物名称 | 引物序列 | 长度(bp) |
|---|---|---|
| LncRNA MX1 | 235 | |
| 正向引物 | 5’-GCAAGACACAAAGCAAGCAC-3’ | |
| 反向引物 | 5’-CAGTGGGTGAGTGAGGACAC-3’ | |
| GAPDH | 147 | |
| 正向引物 | 5’-GAAGGTCGGAGTCAACGGATT-3’ | |
| 反向引物 | 5’-CGCTCCTGGAAGATGGTGAT-3’ |
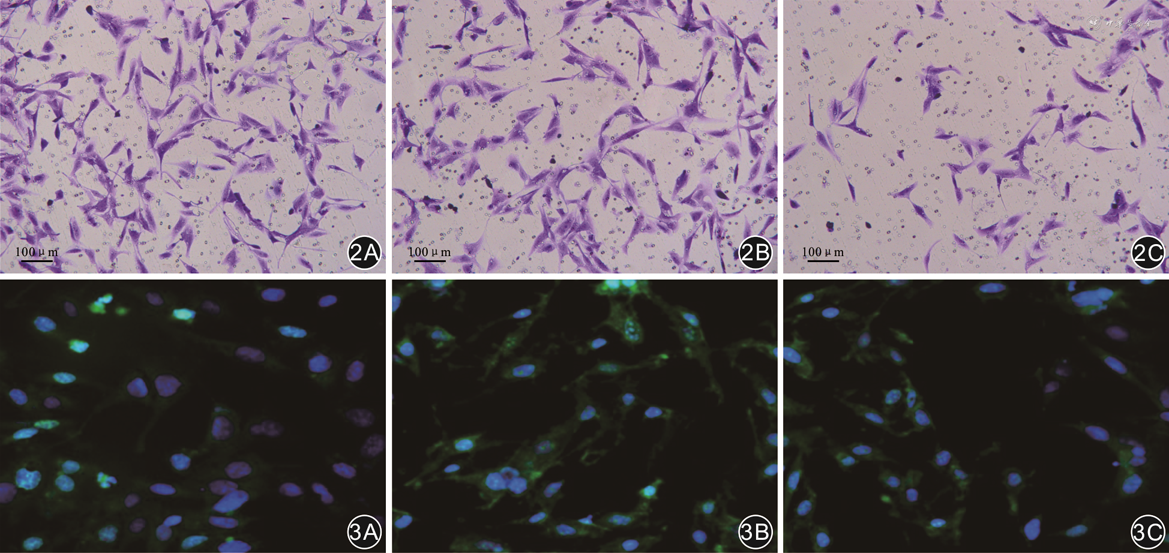
取转染后的对照组、siRNA组和siRNA-MX1组雪旺细胞,收集细胞,调整细胞浓度为2×105/mL的细胞悬液。在小室膜的底面涂敷10 µg/mL纤连蛋白,Transwell上腔室中加入500 µL雪旺细胞悬液,下腔室中加入含10%PBS的RPMI-1640培养基。将Transwell小室于37 ℃、5% CO2培养箱中孵育24 h,用棉拭子擦去上层小室滤过膜上表面的细胞,无水甲醇固定滤过膜下表面细胞30 min,0.1%结晶紫染色。使用倒置显微镜随机选取9个视野,对侵袭细胞数目进行计数。实验重复3次,结果取平均值。
EDU溶液和无血清培养基按1∶1 000稀释制备成EDU培养基。取转染后的对照组、siRNA组和siRNA-MX1组雪旺细胞,调整细胞浓度为3×104/mL,加入EDU培养基在37 ℃、5% CO2培养箱中培养12 h。PBS溶液清洗细胞2次,4%多聚甲醛固定后加入甘氨酸脱色摇床孵育20 min,5% Triton X-100渗透30 min,室温下进行EDU染色。将EDU染色后的雪旺细胞使用PBS溶液清洗5 min,室温下DAPI工作液染色20 min,PBS溶液漂洗2次后封片。荧光显微镜下随机选择5个视野观察、拍照。计算EDU阳性细胞与DAPI阳性细胞的比率,比率越高提示细胞增殖能力越低。EDU阳性细胞为细胞核染色成绿色的细胞,DAPI阳性细胞为细胞核染色成蓝色的细胞。实验重复3次,结果取平均值。
应用GraphPad Prism 5统计学软件对数据进行分析。服从或近似服从正态分布的计量资料以±s表示,大鼠坐骨神经损伤后不同时间点的差异基因采用贝叶斯检验,多组间比较采用方差分析,组间两两比较采用Student’s-t检验。以P<0.05为差异有统计学意义。
各组大鼠造模成功,实验过程中无大鼠死亡。Heatmap聚类热图显示:与T0组比,T1组共有3 066个LncRNA基因发生差异性表达,其中1 634个LncRNA基因表达上调,1 432个LncRNA基因表达下调;T2组共有2 498个LncRNA基因发生差异性表达,其中1 634个LncRNA基因表达上调,864个LncRNA基因表达下调;T3组3 567个LncRNA基因发生差异性表达,其中有1 643个LncRNA基因表达上调,1 924个LncRNA基因表达下调。见图1。对各组差异表达的LncRNA基因进行汇总分析,其中LncRNA MX1的差异倍数值较大,表达差异有统计学意义,最终选择LncRNA MX1进行后续的实验。各组差异倍数前10的LncRNA基因见表2。


注:X轴为基因样本名称;Y轴为基因探针聚类;红色方块为表达上调的基因;绿色方块为表达下调的基因;T0为坐骨神经损伤后0 d;T1为坐骨神经损伤后3 d;T2为坐骨神经损伤后7 d;T3为坐骨神经损伤后14 d
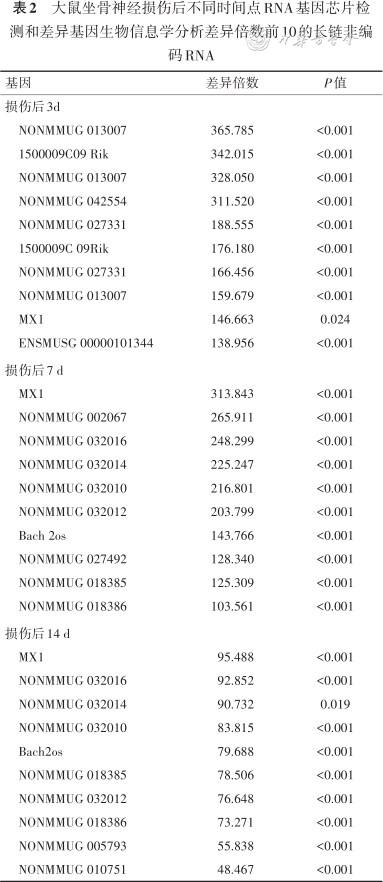
大鼠坐骨神经损伤后不同时间点RNA基因芯片检测和差异基因生物信息学分析差异倍数前10的长链非编码RNA
大鼠坐骨神经损伤后不同时间点RNA基因芯片检测和差异基因生物信息学分析差异倍数前10的长链非编码RNA
| 基因 | 差异倍数 | P值 |
|---|---|---|
| 损伤后3d | ||
| NONMMUG 013007 | 365.785 | <0.001 |
| 1500009C09 Rik | 342.015 | <0.001 |
| NONMMUG 013007 | 328.050 | <0.001 |
| NONMMUG 042554 | 311.520 | <0.001 |
| NONMMUG 027331 | 188.555 | <0.001 |
| 1500009C 09Rik | 176.180 | <0.001 |
| NONMMUG 027331 | 166.456 | <0.001 |
| NONMMUG 013007 | 159.679 | <0.001 |
| MX1 | 146.663 | 0.024 |
| ENSMUSG 00000101344 | 138.956 | <0.001 |
| 损伤后7 d | ||
| MX1 | 313.843 | <0.001 |
| NONMMUG 002067 | 265.911 | <0.001 |
| NONMMUG 032016 | 248.299 | <0.001 |
| NONMMUG 032014 | 225.247 | <0.001 |
| NONMMUG 032010 | 216.801 | <0.001 |
| NONMMUG 032012 | 203.799 | <0.001 |
| Bach 2os | 143.766 | <0.001 |
| NONMMUG 027492 | 128.340 | <0.001 |
| NONMMUG 018385 | 125.309 | <0.001 |
| NONMMUG 018386 | 103.561 | <0.001 |
| 损伤后14 d | ||
| MX1 | 95.488 | <0.001 |
| NONMMUG 032016 | 92.852 | <0.001 |
| NONMMUG 032014 | 90.732 | 0.019 |
| NONMMUG 032010 | 83.815 | <0.001 |
| Bach2os | 79.688 | <0.001 |
| NONMMUG 018385 | 78.506 | <0.001 |
| NONMMUG 032012 | 76.648 | <0.001 |
| NONMMUG 018386 | 73.271 | <0.001 |
| NONMMUG 005793 | 55.838 | <0.001 |
| NONMMUG 010751 | 48.467 | <0.001 |
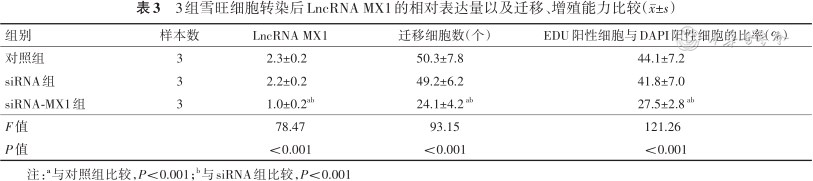
qRT-PCR结果显示,转染LncRNA MX1 siRNA后,siRNA-MX1组LncRNA MX1的相对表达量低于对照组、siRNA组,差异有统计学意义(P值均<0.001),而对照组和siRNA组间差异无统计学意义(P>0.05)。见表3。
3组雪旺细胞转染后LncRNA MX1的相对表达量以及迁移、增殖能力比较(±s)
3组雪旺细胞转染后LncRNA MX1的相对表达量以及迁移、增殖能力比较(±s)
| 组别 | 样本数 | LncRNA MX1 | 迁移细胞数(个) | EDU阳性细胞与DAPI阳性细胞的比率(%) |
|---|---|---|---|---|
| 对照组 | 3 | 2.3±0.2 | 50.3±7.8 | 44.1±7.2 |
| siRNA组 | 3 | 2.2±0.2 | 49.2±6.2 | 41.8±7.0 |
| siRNA-MX1组 | 3 | 1.0±0.2ab | 24.1±4.2 ab | 27.5±2.8 ab |
| F值 | 78.47 | 93.15 | 121.26 | |
| P值 | <0.001 | <0.001 | <0.001 |
注:a与对照组比较,P<0.001;b与siRNA组比较,P<0.001
迁移、增殖实验结果显示,转染LncRNA MX1 siRNA后siRNA-MX1组雪旺细胞迁移数量和EDU阳性细胞与DAPI阳性细胞的比率均低于对照组、siRNA组,差异均有统计学意义(P值均<0.001),而对照组和siRNA组间差异均无统计学意义(P值均>0.05)。结果提示,下调LncRNA MX1在雪旺细胞中的表达会显著降低雪旺细胞的迁移、增殖能力。见表3和图2、3。

相比于中枢神经系统,PNS具有较高的修复和再生能力,PNI后雪旺细胞可以转化为再生表型,从而促进基膜的形成,并释放大量信号来触发神经元再生反应[8]。在PNI后几小时,病灶周围的轴突和髓鞘会发生退化,同时巨噬细胞会逐渐迁移到神经损伤部位;当组织碎片在雪旺细胞和巨噬细胞的共同作用下被清除后,雪旺细胞有规则地排列成柱状,为周围神经轴突再生提供了一个富含营养因子的微环境[9]。尽管已经发现雪旺细胞在PNI后的再生过程中保持着高度的增殖活性,雪旺细胞的迁移可以控制突触的形成,促进轴突修复,引导轴突再生,但调控雪旺细胞增殖与迁移的分子信号仍不清楚[10]。因此,研究雪旺细胞在PNI修复中的调控机制对于PNI的治疗具有重要意义。
目前,LncRNA已被证明在中枢和外周神经损伤部位差异表达,并在神经损伤和再生中具有调节作用[11]。实验表明,有322个LncRNA在缺氧、缺血性脑病中差异表达,其中LncRNA BC088414在脑损伤后显著上调,提示其参与中枢神经系统损伤的相关机制[12]。此外,有105个差异表达的LncRNA在缺氧、缺血性脑损伤后显著上调,提示其参与中枢神经系统损伤的发病机制,表明LncRNA在中枢神经损伤中起重要作用[13]。然而,关于LncRNA在PNI修复中的作用研究仍然相对较少。经高通量RNA-seq测序分析,编码LncRNA MX1的基因位于21号染色体(CRCH38,chr21:41581432e41582698),其转录本全长1 182 nt[14]。目前,LncRNA MX1已被证实在前列腺肿瘤组织和前列腺肿瘤细胞系中高表达,敲除LncRNA MX1可以抑制前列腺肿瘤细胞的增殖和侵袭性,这表明LncRNA MX1与前列腺肿瘤的发生和发展密切相关,在评估前列腺肿瘤的恶性程度和转移预测等方面具有重要的临床价值[15]。然而在PNI研究领域,LncRNA MX1仍未有相关文献报道。因此,在本研究中探讨大鼠坐骨神经损伤后LncRNA差异表达的基因及其调控雪旺细胞促进坐骨神经损伤修复的分子机制,旨在为临床PNI患者的治疗和康复提供新的思路。
聚类分析可以筛选、提取、降维处理基因芯片中的数据,通过颜色展示多样本多个基因表达量的高低,直观反应出数据间的复杂关系,已被广泛应用于分子生物学领域。在本研究中,通过建立大鼠坐骨神经损伤模型,对不同时间点的坐骨神经残端组织提取RNA,进行聚类分析,最终选择出差异表达显著的基因LncRNA MX1。
研究表明,雪旺细胞的高度可塑性是PNI后神经再生的重要因素[16]。在神经修复过程中,雪旺细胞被损伤诱导信号迅速激活,使受损的神经脱髓鞘,通过增殖并上调前髓鞘基因,重新分化为髓鞘细胞,使再生的轴突重新髓鞘化;同时,雪旺细胞的迁移还可以引导轴突神经移植和控制突触的形成来加强轴突修复[16]。因此,雪旺细胞的增殖和迁移能力在PNI后的修复过程中具有重大意义。本研究通过qRT-PCR结果证实,转染LncRNA MX1 siRNA后,siRNA-MX1的表达量低于对照组和siRNA组,进而通过Transwell和EDU细胞功能实验证实下调LncRNA MX1在雪旺细胞中的表达会显著降低其迁移和增殖能力。这一研究结果在证实了沉默LncRNA MX1可以抑制雪旺细胞的增殖和迁移的同时,也在基因层面上补充了雪旺细胞增殖和迁移的调控机制。
综上所述,大鼠坐骨神经损伤后基因LncRNA MX1差异表达显著,下调LncRNA MX1在大鼠雪旺细胞中的表达可显著降低细胞的迁移、增殖能力。本研究样本数较少,对LncRNA MX1促进雪旺细胞的增殖和迁移的具体机制未行进一步研究。后续将增加样本量,对其作用机制进一步深入探讨。
所有作者声明不存在利益冲突



























