
分析阿尔茨海默病(AD)小鼠脑组织长链非编码RNA(LncRNA)和信使RNA(mRNA)表达谱,构建竞争内源性RNA(ceRNA)调控网络,探讨差异表达LncRNA在AD发病机制中的潜在作用。
选取3只10月龄雄性APP/PS1转基因小鼠作为AD组,3只年龄及体质量相匹配的普通C57小鼠作为对照组。使用基因芯片技术检测2组小鼠脑组织LncRNA和mRNA的表达,筛选出差异表达的LncRNA和mRNA。对部分差异表达的LncRNA进行实时定量聚合酶链反应(qRT-PCR)。对差异表达的mRNA进行基因本体论(GO)和京都基因、基因组百科全书(KEGG)通路分析。随机挑选6个差异表达LncRNA构建ceRNA网络,进行AD的靶基因功能预测分析。
与对照组相比,AD组小鼠脑组织差异表达1.5倍以上的LncRNA有933个,其中上调222个,下调711个;差异表达1.5倍以上的mRNA有529个,其中上调189个,下调340个。qRT-PCR检测结果显示,AD组与对照组比较,7个差异表达的LncRNA上调或下调趋势与基因芯片结果一致,差异均有统计学意义(P值均<0.05)。GO和KEGG通路分析结果显示,差异表达基因主要参与氨基酸代谢、炎症反应和免疫反应。ceRNA调控网络靶基因的功能富集分析显示,LncRNA在胰岛素抵抗以及糖尿病并发症中的AGE-RAGE信号通路中显著富集。
AD小鼠脑组织LncRNA表达谱发生显著变化,由LncRNA Dgkb、Svip等构建的ceRNA调控网络有助于增进对AD发病分子机制的研究,差异表达的LncRNA或通路有可能成为潜在的治疗靶点。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
阿尔茨海默病(Alzheimer's disease,AD)是由内嗅皮层开始并进展到海马和大部分大脑皮层的神经退行性疾病[1, 2],其特征是β淀粉样蛋白(amyloid β,Aβ)沉积形成的老年斑和tau蛋白过度磷酸化形成的神经元纤维缠结,主要表现为进行性学习记忆水平下降、认知功能障碍和行为异常[3]。研究表明,淀粉样前体蛋白、早老蛋白-1等基因在AD中具有高表达,因此,阐明介导AD发生相关的基因变化至关重要。
非编码RNA,如长链非编码RNA(long non-coding RNA,LncRNA)和微小RNA(microRNA,miRNA)是没有蛋白质编码能力的功能性RNA分子。miRNA可诱导降解并抑制其靶基因的翻译。LncRNA作为基因调控因子,在转录和转录后水平调控相关基因表达[4]。尽管人们对LncRNA在AD动物模型及人类表达谱中的研究已经有很多,但关于LncRNA在AD中的具体表达模式和潜在的生物学功能仍不清楚。研究表明,LncRNA参与AD的病理生理学发展过程,包括Aβ斑块的形成和发展、神经元纤维缠结、突触丢失和神经元死亡[5, 6]。最近的研究揭示了AD与LncRNA相关的竞争内源性RNA(competing endogenous RNA,ceRNA)网络之间有潜在联系,ceRNA调控网络是预测LncRNA功能的有效策略[7]。因此,本研究利用基因芯片技术检测并分析APP/PS1转基因AD小鼠脑组织LncRNA和mRNA差异表达,同时构建LncRNA-miRNA-mRNA ceRNA网络,分析差异表达LncRNA在AD发病机制中的潜在作用。
主要试剂:Arraystar小鼠LncRNA 芯片(V4.0)、Arraystar基因芯片配套试剂盒、2×PCR master mix试剂盒为美国Arraystar公司产品;Trizol试剂、SuperScriptTM Ⅲ RT master mix试剂盒为美国Invitrogen公司产品;RNeasy Mini Kit试剂盒为美国Qiagen公司产品。
主要仪器、软件:NanoDrop ND-1000分光光度仪为美国NanoDrop公司产品;Agilent DNA微阵列扫描仪、Agilent Feature Extraction 软件(v11.0.1.1)、GeneSpring GX 软件(v12.1)为美国Agilent公司产品;QuantStudioTM 5 Real-time PCR System为美国Applied Biosystems公司产品。
选取3只10月龄雄性APP/PS1转基因小鼠(20~30 g)作为AD组,3只年龄及体质量相匹配的普通C57小鼠作为对照组。实验动物均购自南京君科生物工程有限公司,许可证号:SCXK(苏)2020-0009。采用颈椎脱位法处死2组小鼠,开颅剥离完整脑组织,保存在-80 °C冰箱备用。实验过程中遵循实验动物伦理规定。
按照Trizol试剂说明提取小鼠脑组织总RNA。用NanoDrop ND-1000测定RNA质量和数量,用标准变性琼脂糖凝胶电泳评估RNA完整性。使用Arraystar小鼠LncRNA V4.0芯片、Arraystar基因芯片配套试剂盒进行基因芯片杂交。使用Agilent DNA微阵列扫描仪对杂化阵列进行洗涤、固定和扫描。
采用Agilent Feature Extraction v11.0.1.1软件获取芯片信号强度图和原始数据,并应用GenesSpring GX v12.1软件对原始数据进行归一化处理和差异数据分析。筛选差异表达LncRNA和mRNA标准为:差异倍数(fold change,FC)绝对值(|FC|)≥1.5,P值<0.05。
从差异表达的LncRNA中随机选取7个进行qRT-PCR验证。根据SuperScriptTM Ⅲ RTmaster mix逆转录试剂盒操作步骤,首先将2组小鼠脑组织总RNA逆转录为cDNA。逆转录条件:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s,40个循环。将所得cDNA按照2×PCR master mix试剂盒进行扩增,反应条件:95 ℃ 10 min;95 ℃ 10 s,60 ℃ 60 s,40个循环。使用甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)作为内参,根据反应曲线得出阈值循环参数后,应用QuantStudioTM 5 Real-time PCR 仪分析荧光阈值(cycle threshold,Ct)。2-ΔΔCt法分析目的基因的相对表达量。重复实验3次,结果取平均值。引物序列见表1。

目的基因引物序列和长度
目的基因引物序列和长度
| 目的基因(LncRNA) | 引物序列 | 长度(bp) |
|---|---|---|
| AK034056 | 227 | |
| 正向引物 | 5'-GGAGAGACGCTTATTGGGTGA-3' | |
| 反向引物 | 5'-CATTCTTTCCCTAACACCCTGG-3' | |
| AK047492 | 203 | |
| 正向引物 | 5'-AGTGAGTCATCGGTCTGGTTT-3' | |
| 反向引物 | 5'-CAGGTCCAGTGTTAGAACTTTGA-3' | |
| AK053427 | 88 | |
| 正向引物 | 5'-TGTTGATATCTGGAGACGGTG-3' | |
| 反向引物 | 5'-TGTTTTGAGTAAAGAGAGGGGA-3' | |
| ENSMUST00000099676 | 274 | |
| 正向引物 | 5'-AGTCTTCTGCCATCAAGCCAA-3' | |
| 反向引物 | 5'-AGGCAAGGCAAGTGTCTACT-3' | |
| ENSMUST00000145263 | 87 | |
| 正向引物 | 5'-AACGCGCATCTGTGCCTAG-3' | |
| 反向引物 | 5'-GCAATTGAAGCAAAAGGGTCT-3' | |
| uc056yur.1 | 204 | |
| 正向引物 | 5'-AGGTTGTCATTTTGCCTTTTC-3' | |
| 反向引物 | 5'-GCTCTTTTCACTATGTGCCGT-3' | |
| Map3k8 | 63 | |
| 正向引物 | 5'-AGCATGGAGCTCGGCTACAG-3' | |
| 反向引物 | 5'-CTCCTTCCTAAAACACACTTCTTCT-3' | |
| GAPDH | 124 | |
| 正向引物 | 5'-GCTCTCTGCTCCTCCCTGTTCTA-3' | |
| 反向引物 | 5'-TGGTAACCAGGCGTCCGATA-3' | |
利用GO数据库(http://www.geneontology.org)对差异表达的mRNA进行GO条目分析,从分子功能、细胞成分和生物过程3个方面分析差异表达mRNA显著富集的生物学作用。利用KEGG数据库(http://www.genome.jp/kegg)对差异表达的mRNA进行KEGG通路分析,探讨差异表达的mRNA显著富集的信号通路。
随机挑选与AD相关的差异表达LncRNA(Dgkb、Plcl1、Svip、XLOC_015776、Sox2ot和Rora)构建ceRNA网络。利用miRanda数据库预测与所选差异表达LncRNA相互作用的miRNA(数量限制在1 000个)。应用miRanda和TargetScan数据库预测差异表达miRNA的靶向mRNA。利用所选LncRNA以及预测miRNA和mRNA构建ceRNA网络,使用Cytoscape 3.4.0可视化ceRNA网络,对ceRNA靶基因进行GO和KEGG通路富集分析,以全面了解ceRNA的作用。
应用SPSS 26.0 统计学软件对数据进行统计分析。服从正态分布的计量资料以±s表示,采用独立样本t检验。以P<0.05为差异有统计学意义。
在AD组与对照组脑组织中共鉴定出37 645个LncRNA转录本和22 122个mRNA转录本。对2组LncRNA和mRNA的表达量进行差异分析发现,AD组小鼠脑组织差异表达1.5倍以上的LncRNA有933个,其中上调222个,下调711个;差异表达1.5倍以上的mRNA有529个,其中上调189个,下调340个。见图1、2。差异表达前10位的LncRNA和mRNA见表2。
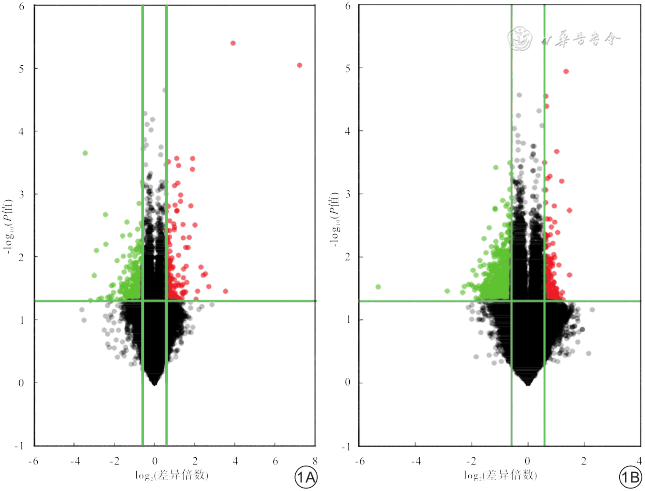

注:红色为显著上调基因;绿色为显著下调基因;黑色为无差异变化的基因
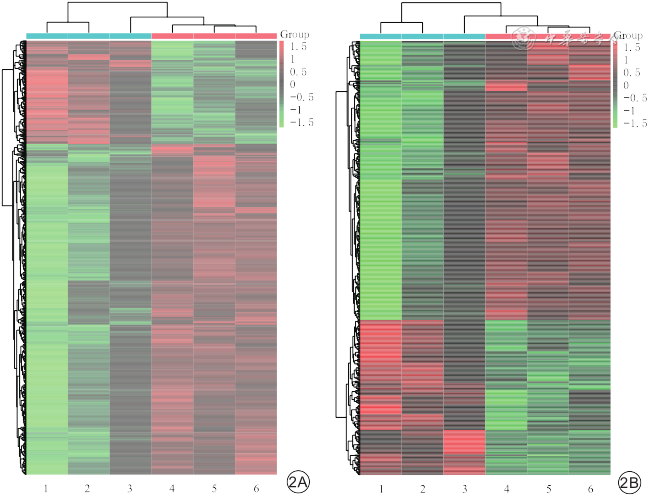

注:1~3为阿尔茨海默病组小鼠脑组织基因样本,4~6为对照组小鼠脑组织基因样本;纵轴为基因聚类,一行代表一个基因;不同颜色为基因的相对表达量,红色代表上调的表达,绿色代表下调的表达,颜色越亮基因相对表达量越高
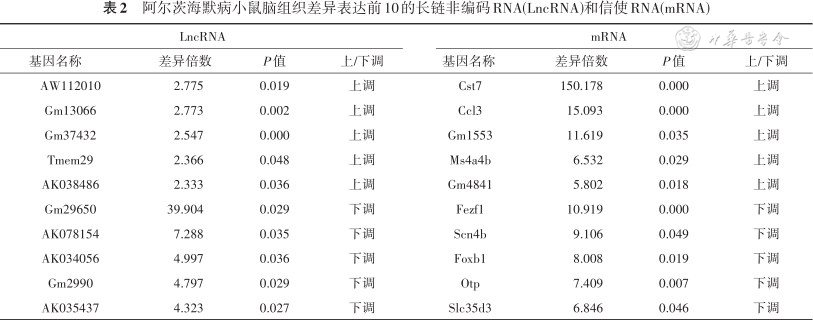
阿尔茨海默病小鼠脑组织差异表达前10的长链非编码RNA(LncRNA)和信使RNA(mRNA)
阿尔茨海默病小鼠脑组织差异表达前10的长链非编码RNA(LncRNA)和信使RNA(mRNA)
| LncRNA | mRNA | |||||||
|---|---|---|---|---|---|---|---|---|
| 基因名称 | 差异倍数 | P值 | 上/下调 | 基因名称 | 差异倍数 | P值 | 上/下调 | |
| AW112010 | 2.775 | 0.019 | 上调 | Cst7 | 150.178 | 0.000 | 上调 | |
| Gm13066 | 2.773 | 0.002 | 上调 | Ccl3 | 15.093 | 0.000 | 上调 | |
| Gm37432 | 2.547 | 0.000 | 上调 | Gm1553 | 11.619 | 0.035 | 上调 | |
| Tmem29 | 2.366 | 0.048 | 上调 | Ms4a4b | 6.532 | 0.029 | 上调 | |
| AK038486 | 2.333 | 0.036 | 上调 | Gm4841 | 5.802 | 0.018 | 上调 | |
| Gm29650 | 39.904 | 0.029 | 下调 | Fezf1 | 10.919 | 0.000 | 下调 | |
| AK078154 | 7.288 | 0.035 | 下调 | Scn4b | 9.106 | 0.049 | 下调 | |
| AK034056 | 4.997 | 0.036 | 下调 | Foxb1 | 8.008 | 0.019 | 下调 | |
| Gm2990 | 4.797 | 0.029 | 下调 | Otp | 7.409 | 0.007 | 下调 | |
| AK035437 | 4.323 | 0.027 | 下调 | Slc35d3 | 6.846 | 0.046 | 下调 | |
qRT-PCR结果显示,AD组与对照组相比,LncRNA AK034056、AK047492、AK053427和uc056yur.1 表达下调,LncRNA AW112010、Gm13066和Map3k8表达上调,差异均有统计学意义(P值均<0.05),见表3。检测结果与芯片结果相符。
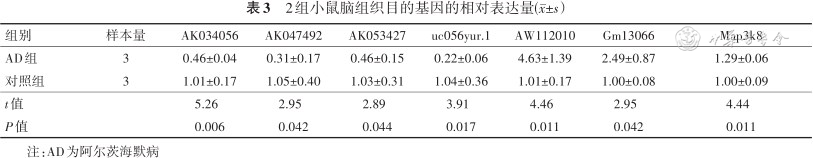
2组小鼠脑组织目的基因的相对表达量(±s)
2组小鼠脑组织目的基因的相对表达量(±s)
| 组别 | 样本量 | AK034056 | AK047492 | AK053427 | uc056yur.1 | AW112010 | Gm13066 | Map3k8 |
|---|---|---|---|---|---|---|---|---|
| AD组 | 3 | 0.46±0.04 | 0.31±0.17 | 0.46±0.15 | 0.22±0.06 | 4.63±1.39 | 2.49±0.87 | 1.29±0.06 |
| 对照组 | 3 | 1.01±0.17 | 1.05±0.40 | 1.03±0.31 | 1.04±0.36 | 1.01±0.17 | 1.00±0.08 | 1.00±0.09 |
| t值 | 5.26 | 2.95 | 2.89 | 3.91 | 4.46 | 2.95 | 4.44 | |
| P值 | 0.006 | 0.042 | 0.044 | 0.017 | 0.011 | 0.042 | 0.011 |
注:AD为阿尔茨海默病
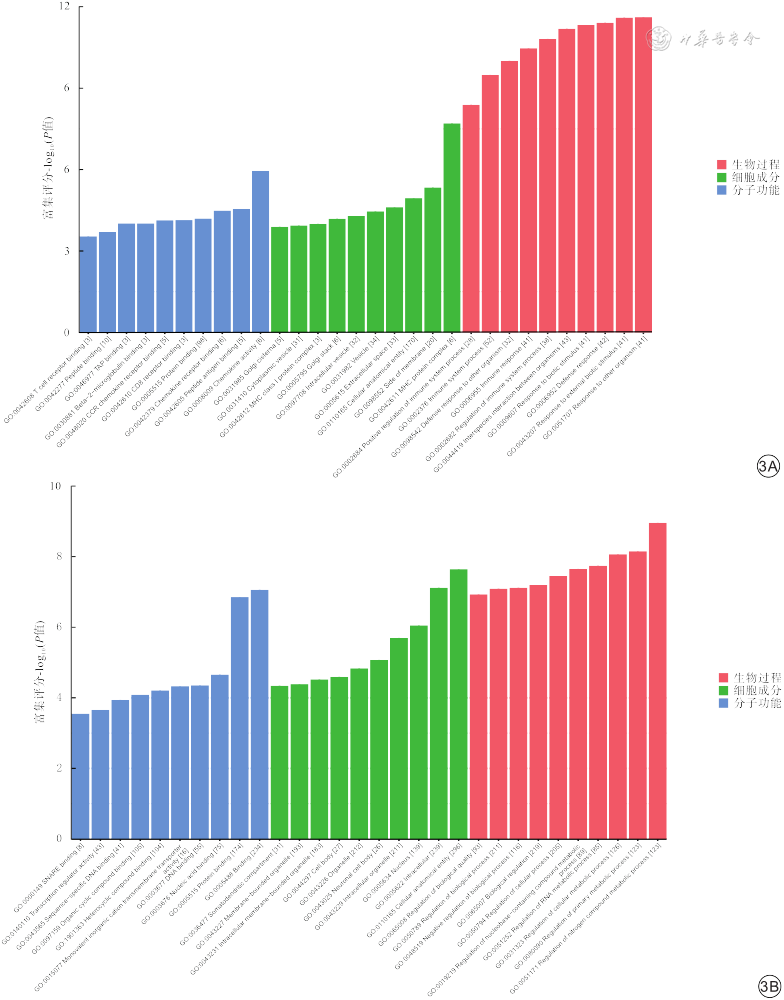
GO富集分析:529个差异表达的mRNA中,表达上调的mRNA参与812个生物学过程,表达下调的mRNA参与870个生物学过程,差异均有统计学意义(P值均<0.05)。上调的差异表达mRNA主要富集于趋化因子活动、MHC蛋白复合物以及对外部生物刺激的反应等生物过程;下调的差异表达mRNA主要富集于蛋白结合、对初级代谢过程的调节、氮化合物代谢过程的调节等生物过程。差异表达mRNA富集评分前10的GO分析条目见图3。

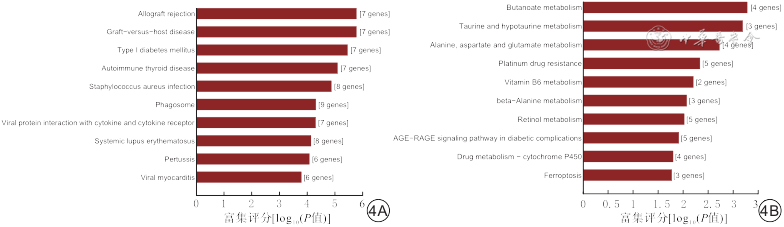
KEGG通路富集分析:529个差异表达的mRNA中,表达上调的mRNA富集得到32条通路,表达下调的mRNA富集得到16条通路,差异均有统计学意义(P值均<0.05)。上调的差异表达mRNA主要富集于异体移植排斥反应、移植物抗宿主病、自身免疫性甲状腺疾病等炎症反应通路;下调的差异表达mRNA主要富集于丁酸盐代谢、牛磺酸和次牛磺酸代谢、谷氨酸代谢等氨基酸代谢途径。KEGG通路分析富集评分前10的通路见图4。

使用miRanda和TargetScan数据库,预测得到miRNA的靶向mRNA 550个。根据所选LncRNA及预测的miRNA和mRNA构建LncRNA-miRNA-mRNAc ceRNA网络,见图5。


注:红色圆圈为微小RNA(miRNA);蓝色圆圈为信使RNA(mRNA);绿色圆圈为长链非编码RNA(LncRNA)
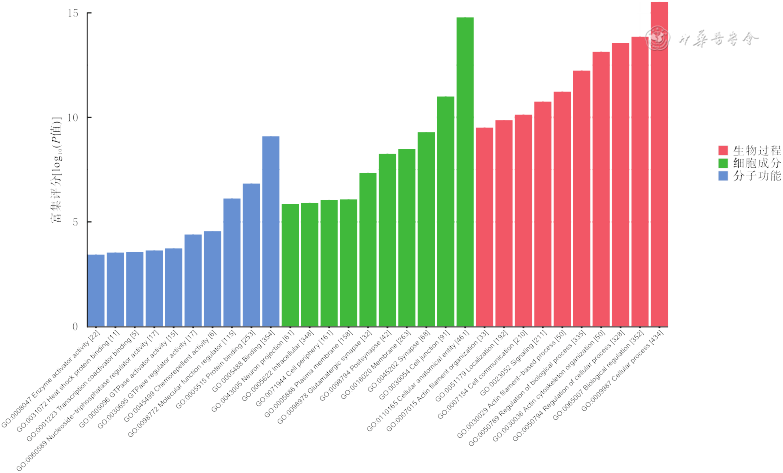
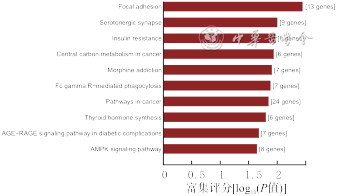
对ceRNA预测的靶基因进行GO和KEGG通路分析:GO分析显示,550个差异表达的mRNA富集得到1 414个GO条目(P值均<0.05),其主要富集于蛋白结合、细胞连接以及生物调节等生物过程;KEGG通路分析发现,550个差异表达的mRNA富集得到27条通路(P值均<0.05),主要富集于胰岛素抵抗、Fcγ介导的吞噬作用、糖尿病并发症中的AGE-RAGE信号通路和AMPK信号通路等。见图6、7。


AD是一种常见的神经退行性疾病,其发生的病理机制尚不完全清楚,主要有遗传因素、炎性机制、免疫机制等。近年来,有学者指出AD的发生与糖尿病小血管病变密切相关,导致Aβ沉积和tau蛋白过度磷酸化[8]。我们课题组对AD和糖尿病的相关性进行了研究,并且发现体外腹腔注射胰岛淀粉样多肽可减轻AD的认知损害[9]。随着基因测序技术的发展,越来越多的研究将非编码RNA作为AD早期诊断的潜在生物标志物[10, 11],关于AD的研究也主要集中在AD发病机制的表观遗传调控方面[12, 13]。本研究结果显示,与对照组相比,AD小鼠脑组织差异表达的LncRNA有933个,差异表达的mRNA有529个;经qRT-PCR验证显示与基因芯片结果一致,证明LncRNA基因芯片检测结果可靠。
本研究GO分析结果显示,其主要参与神经投射、突触传递调控、神经元分化、G蛋白偶联受体结合等生物学过程。这些生物过程与AD的病理发展相关[14]。KEGG分析结果显示,上调的mRNA主要参与炎症/免疫反应,下调的mRNA主要参与氨基酸代谢。免疫炎症反应作为神经退行性疾病的核心特征,影响AD的每个病理变化(包括Aβ沉积,tau蛋白过度磷酸化以及突触和神经元丢失),并使其进一步复杂化[15]。我们观察到许多与炎症反应相关的LncRNA分子表达失调,如lncRNA MEG3、Svip、Map3k8,这与之前研究报告的结果一致[16, 17, 18],表明在AD发生时免疫系统受到破坏,炎症反应被强烈激活。另外,一些氨基酸(如牛磺酸、天冬氨酸和谷氨酸)在大脑中的表达水平高于其他组织,这些氨基酸有助于学习和记忆维持[19]。对AD患者和AD模型小鼠的氨基酸代谢组学分析表明,氨基酸代谢受损与AD发病机制相关,并且改变的氨基酸特征可以作为AD诊断的生物标志物[20, 21]。
ceRNA假说指出,LncRNA可通过竞争共享的miRNA来调节基因表达[7]。在本研究中,我们构建了ceRNA调控网络并对其预测的靶基因进行GO和KEGG分析,结果显示其主要富集于胰岛素抵抗以及糖尿病并发症中的AGE-RAGE信号通路。Ma等[22]对3、6、9月龄AD小鼠大脑皮层进行的关于AD发病机制和治疗策略的LncRNA测序研究中,结果也发现差异表达的LncRNA参与胰岛素抵抗或胰岛素分泌调节。有证据表明,胰岛素抵抗参与了AD的发生发展,这与许多分子机制有关,包括神经炎症、氧化应激、Aβ积聚、tau过度磷酸化和突触衰竭[8,23]。胰岛素可介导神经元和神经胶质细胞的生长、代谢、基因表达、神经递质网络和突触功能[24]。胰岛素缺乏会引起Aβ和淀粉样斑块的积累,增加tau蛋白磷酸化,以及空间记忆缺陷[25]。高血糖也会影响神经元稳态,例如在糖尿病前期或2型糖尿病患者中反复短暂的高血糖发作,可能会影响KATP通道,改变神经元代谢并增加Aβ水平[26]。葡萄糖代谢失调会驱动糖和脂质、蛋白质和核酸的游离氨基之间的不受控制和非酶促反应,产生晚期糖基化终产物。糖基化终产物在患有糖尿病的AD患者中比在非糖尿病AD患者中更丰富。研究证明,糖基化终产物会损害Aβ的正常清除,并有利于Aβ和tau糖基化,从而促进淀粉样蛋白斑块和神经元纤维缠结的形成[27]。以上这些结果表明,LncRNA可通过ceRNA网络调控蛋白编码基因,进而影响多个关键信号通路活性来参与AD的发生和发展。
综上所述,本研究通过LncRNA芯片技术探讨了AD小鼠脑组织LncRNA和mRNA表达谱变化,发现异常调控的LncRNA可能在AD的发生中起重要作用。构建的ceRNA网络有助于进一步增进对AD发病分子机制的研究,差异表达的LncRNA或通路有可能成为潜在的治疗靶点。本研究的局限性:(1)在测序前未对淀粉样前体蛋白/早老蛋白-1转基因AD小鼠进行相关炎症指标检测;(2)仅使用淀粉样前体蛋白/早老蛋白-1小鼠脑组织进行分析,血液和脑脊液中的全局LncRNA和mRNA变化也应在进一步的研究中确定,以更准确地反映AD的病理生理学。后续还需在细胞实验中进一步验证这些ceRNA在AD中的潜在生物学功能,并通过更深入的数据分析和更广泛的LncRNA验证完善研究结果。
李金平, 李小雄, 马琳秋, 等. 阿尔茨海默病小鼠脑组织长链非编码RNA与信使RNA表达谱及调控网络的分析[J]. 中华解剖与临床杂志, 2023, 28(2): 112-120. DOI: 10.3760/cma.j.cn101202-20221030-00331.
所有作者声明不存在利益冲突



























