
MR是一种临床常用的、无创成像技术。由于其采集、成像时间长使其推广应用受到限制,故为了减少扫描时间,基于信号处理的不完整K空间重建技术成为新的发展趋势。随着深度学习在医学成像应用上的成功,基于神经网络的降采样MR图像重建算法受到了广泛的关注,其重建结果及效率优于传统的压缩感知(CS)等重建方法。在此就重建算法的发展历程,对基于深度学习的MR重建算法从模型、数据流、训练方面进行系统阐述。

本刊刊出的文章除特别声明外,不代表主办单位和本刊编委会的观点。本刊如有印刷质量问题,请与本刊编辑部联系调换。
MR是一种无创、无电离辐射的成像技术,通过施加特定频率的射频信号和可变的梯度磁场,可以提供多种软组织成像、组织代谢、生理功能变化等信息,是临床上常用的检查手段[1, 2]。MR通过采集编码空间体素中的质子磁矩变化,将信号填充为K空间(K-Space)。在K空间中获取的矩阵大小与重构MR图像的质量及分辨率成正相关[3]。当空间分辨率或成像区域增加,需要采样的K空间点数也会增加,所需的扫描时间随之加长。在长时间的扫描过程中,图像质量不可避免地受到呼吸、血流、心跳和患者运动等因素影响[4];因此,很多研究者致力于通过减少K空间采样的方式以缩短MR扫描时间。降采样K空间数据会导致图像混叠和低信噪比,在过去的几十年里已开发出一些经典的技术方案来改善这一问题,如并行成像(parallel imaging,PI)[5]、压缩感知(compressed sensing,CS)[3]等。近年来,深度学习在超分辨图像、去噪声、图像分割等医学成像应用上表现出优于传统方法的巨大潜力,为MR图像重建算法方向提供了新思路。以下将从模型类型、训练数据类型和学习模式3种角度对基于深度学习的MR图像重建算法的发展历程进行综述,旨在帮助读者深入了解其优点及局限性。
早期的MR加速利用K空间数据的共轭对称性进行部分采集[6, 7],但这种半扫描的加速方式是有限的。并行成像的出现为MR加速方法提供了新的发展方向[8],其使用多个射频接收线圈以获得比所需更少的数据,利用多个接收线圈的灵敏度编码来恢复未采集的数据,目前已广泛集成在MR系统中。在重建算法上,CS将PI与低秩约束项相结合,利用K空间数据的稀疏性进行重建[9, 10],如线圈敏感度编码(sensitivity encoding,SENSE)[11]和自校准部分采集并行成像(generalized autocalibrating partially parallel acquisitions,GRAPPA)[5]算法。一些基于低秩约束的算法也相继出现[12, 13],但降采倍数较高时,高频振荡伪影仍难以消除。字典学习在图像域和K空间来回交替更新自适应字典[14],可以更好地表现图像的稀疏化。然而这些传统的算法往往涉及复杂、大规模的矩阵、迭代运算,其对计算成本的高要求、重建时间的延长影响了MR的临床应用与推广。
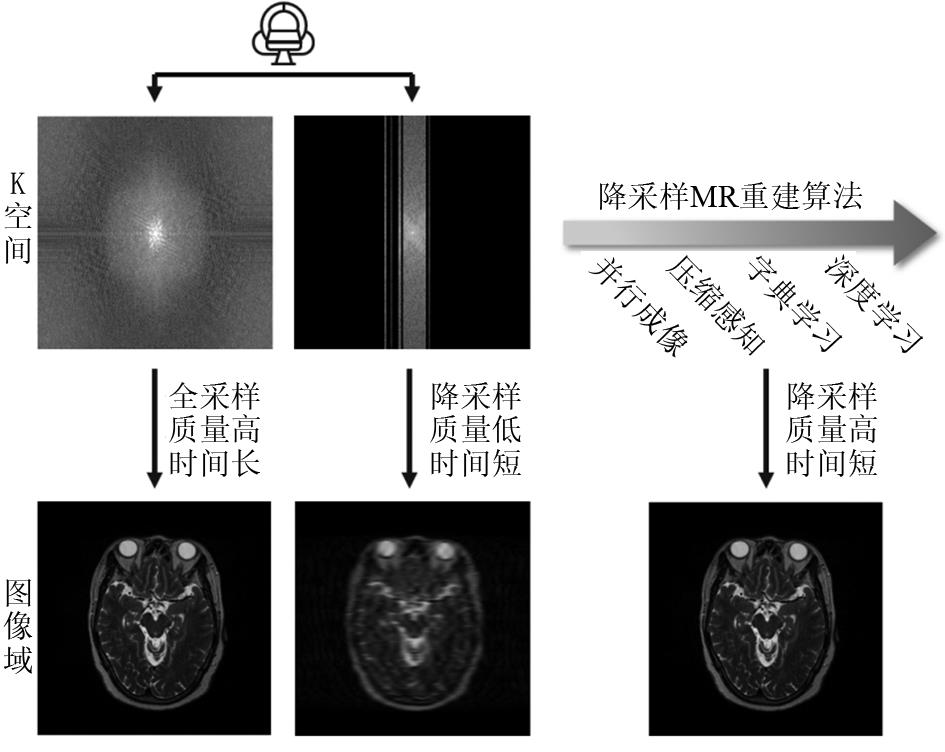
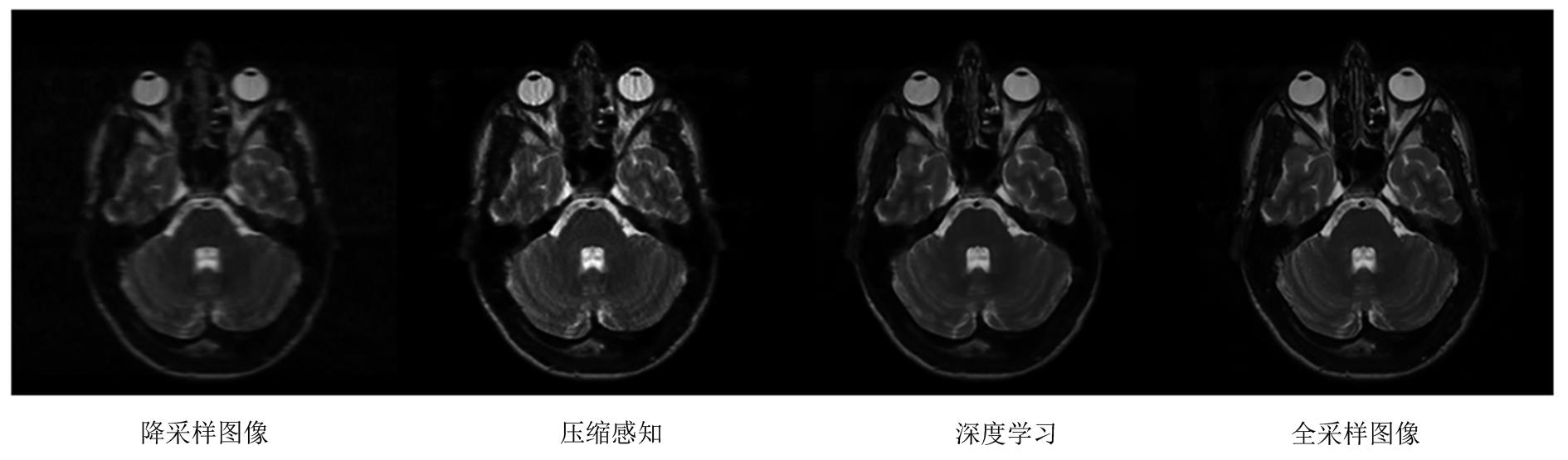
近年来,基于深度学习的方法被提出并成功应用于MR成像,多种针对降采样图像重建的算法相继出现。以大数据作为先验知识,以其更快速的前馈网络处理速度和更好的重建效果,逐渐成为MR重建算法的研究新热点。降采样的MR重建算法经历了半扫描直接重建、基于CS和低秩等方法的传统算法,到基于深度学习的新兴算法(图1),其重建速度逐渐加快,重建效果去伪影、降噪等方面逐渐加强,可适用的降采倍数也逐步提高。以颅脑T2WI为例(图2),在4倍降采样下使用不同类型算法的重建效果,直接重建的图像受到严重的伪影影响,组织结构难以辨认;使用CS的传统算法重建对噪声和伪影有一定的去除作用,但图像质量仍然不佳;使用深度学习算法重建的图像较好地抑制了降采伪影,还原了全采样图像中的解剖结构信息。基于深度学习的MR重建算法将进一步推动医学影像成像速度和质量的提高。


基于深度学习的快速MR重建方法根据驱动类型可分为2类:数据驱动和模型展开。基于数据驱动能够挖掘大数据的先验信息,在模型结构上具有丰富的类型,适用于一些特定的重建任务,但其通常依赖大规模数据集的训练,在采样参数更改的情况下需要重新训练。模型展开则基于解逆问题的数学模型,相比于数据驱动通常能够在数据量较小的情况下进行训练并取得较好结果。
基于数据驱动的重建算法利用大数据信息,训练一个通用网络,以端到端的方式学习欠采样和完全采样数据对之间的非线性映射,当采集方式改变时需要重新训练网络。我们基于深度学习网络结构的不同,对一些具有代表性的算法进行介绍(图3)。
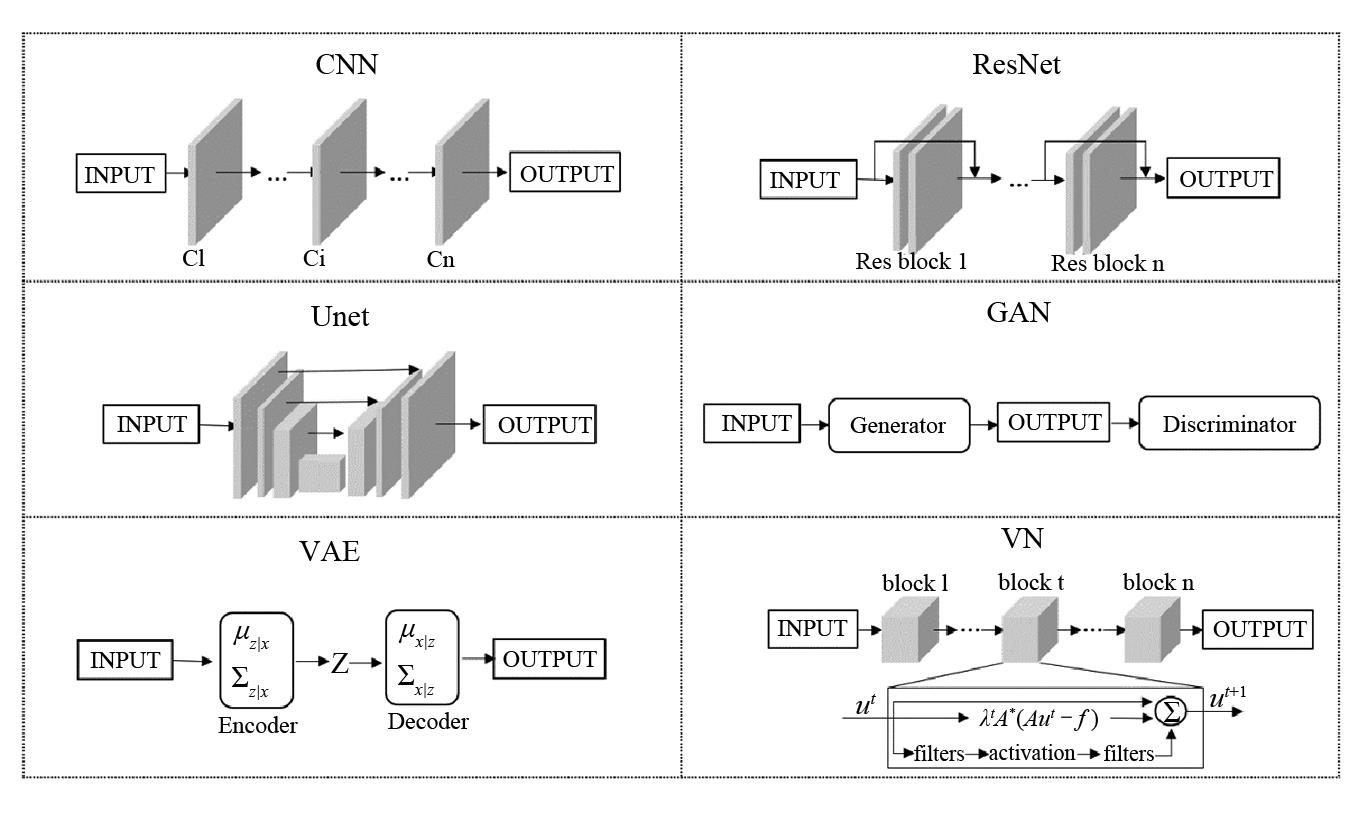

注:CNN:卷积神经网络,ResNet:残差神经网络,Unet:U型神经网络,GAN:对抗生成神经网络,VAE:变分自编码器,VN:变分网络
卷积神经网络(convolutional neural network,CNN)是受到人类视觉神经系统启发的基础网络结构,由卷积层、激活层和池化层构成。卷积层用于提取出深层特征,激活层增加非线性映射能力,池化层将特征空间降维。CNN网络被广泛应用于图像重建生成领域,Schlemper等[15]将降采样的二维动态心脏MR图像应用于CNN网络,能够在11倍降采样下恢复解剖结构。Wang等[16]提出了一种多监督损失函数,对频域信息和重构结果进行不同层次的约束,该方法可以在较短的时间内获得较好的重建结果。Cheng等[17]构建了基于CNN的数据似然模型,能够捕获成像噪声的实际分布。
残差神经网络(ResNet)[18]在CNN的基础上增加了快捷连接分支,以退化方式消除过深网络训练困难问题。Fu等[19]提出了一个适用于放射状采集的MS-ResNet算法,其训练预处理策略使用含有欠采样伪影和不含伪影的多对比度图像,该方法在大脑和膝盖数据集上都有良好的重建效果。U型神经网络(Unet)由编码器与解码器组成,最早应用于医学图像分割领域[20],通过编码器的连续卷积提取出深层维度的特征,由解码器做图像生成,并通过前后的跳跃连接,使不同维度的特征融合。Peng等[21]提出了一种基于Unet的线圈灵敏度函数估计方法,能够完成低分辨到高分辨率灵敏度间的映射,与SENSE方法结合能够更好地抑制混叠和噪声。
对抗生成神经网络(generative adversarial networks,GAN),这种网络常用于图像风格迁移问题,实现像素与像素间的映射关系,且效果较为清晰[22]。网络基础结构主要由生成器与判别器构成。其中,生成器的作用是将输入图像的风格转换为目标图像风格,尽量使生成的图像无限贴近于目标图像;判别器的作用是分辨成对输入的图像究竟是否真实存在;因此,生成器与判别器之间是对抗的关系。基于网络结构、损失函数等方面的改进,有很多基于GAN的改进工作,如DCGAN(deep convolutional generative adversarial networks)[23],WGAN(wasserstein generative adversarial networks)[24],LSGAN(least squares generative adversarial networks)[25]等。GAN网络在降采样图像重建算法中应用广泛,DAGAN(de-aliasing generative adversarial networks)将L2与感知损失相结合[26],GANCS(generative adversarial neural networks for compressive sensing)利用最小二乘的GAN损失和像素级的L1及L2损失的混合[27],都获得了较传统方法更好地重建细节。一些研究也致力于GAN网络结构的变体,RefineGAN利用一个链式网络来进一步提高重构质量,生成器由双重Unet网络组成,分别对应重建步骤和细节优化步骤,并增加了频域的损失函数[28]。Shaul等[29]则是将级联网络分别对应K空间补充和图像域重建步骤。GAN网络是重建算法研究的热点,有大量研究者致力于基于GAN及其变体的工作[30, 31, 32]。
其他种类的深度学习网络也被应用于重建算法中。变分自编码器(variational autoencoder networks,VAE)是一种生成模型,通过编码器对图像进行隐式整合,解码器将低维向量重构。Tezcan等[33]采用VAE隐含编码全采样图像的概率分布,将其作为重建过程中解码的先验项,从而实现任意模式下降采样K空间的重建。变分网络(variational network,VN)是一种超梯度下降迭代算法,滤波核和激活函数等正则项参数都在训练中不断更新,学习后的模型较好地应用于回顾性和前瞻性的欠采样数据上[34, 35, 36]。
基于模型展开是将优化算法的迭代展开到神经网络中,利用正则化等先验知识,使神经网络能够自动学习超参数或转换方法以解决逆问题。Yang等[37, 38]将交替方向乘子法(alternating direction multiplier method,ADMM)优化算法与深度学习结合,将CS模型扩展为具有待定稀疏变换和变换域正则化的模型,取得了较好地重建精度。Lahiri等[39]将CNN网络与字典学习结合,能够较好地保留高频细节,在不同数据集大小和序列下都有相对稳健的重建效果。许多研究者也将深度学习与其他传统重建模型相结合[40, 41, 42],开发出了较原有方法更佳的算法。
根据神经网络输入与输出数据类型,重建算法可以分为3类:(1)降采图像域到满采图像域之间的映射模型;(2)降采K空间到满采K空间的映射模型;(3)K空间到图像域的混合映射模型。
与自然图像的一些工作效果类似,图像域间的映射算法在去噪声和去伪影方面效果较好,但在图像细节的保留还原方面存在不足。目前重建算法领域大多数研究集中在图像域到图像域的映射模型[43, 44, 45]。图像域的重建算法主要使用单层或相邻采集的多层数据作为网络输入,这些数据是以实数形式存储的。Xiao等[46]提出了一种基于复数数据输入的网络,可以同时重建出保真的幅值与相位,基于复数的重建算法可以提供更多的临床价值[47, 48]。
K空间的映射算法可以更好的保留图像的精细结构,模型对数据集的依赖性小。RAKI(Robust Artificial-neural-networks for K-space Interpolation)[49]使用非线性的神经网络估计缺失的K空间数据,在降采样方式改变后,提供了一种无需重新训练的深度学习重建方法。SPARK(Scan-specific Artifact Reduction in K-space)[50]训练特定于扫描的模型来估计和纠正K空间中的重建均方误差。Han等[51]将CS中基于消除滤波器的低秩汉克尔矩阵方法和深度学习联合,以实现对降采样K空间的插值,且适用于不同的采样轨迹。
部分研究者致力于跨域的重建工作,以期望均衡以上2种方法的优劣。KIKI-net使用交替的K空间CNN网络模块与图像域网络模块,在3~4倍的降采能够较好重建出人脑影像[52]。Zhu等[53]通过有监督的卷积网络和全连接网络实现K空间到图像域之间的映射,应用于对K空间MR采集和相应的图像空间,证明了对噪声和重建伪影的免疫力。Shaul等[29]提出了一个双域的GAN网络,串联K空间生成器和图像域生成器,在多个数据集上均取得良好结果。此外,也有研究者致力于跨域的图像重建工作[16,54, 55, 56, 57, 58],并在不同数据集上取得了较好的重建结果。
在大多数重建算法的研究中,研究者专注于单一对比度图像的生成,模型的多通道输入是相邻层数据[29],相邻帧数据[15]或多通道线圈数据[57]。
在临床应用中,常采用多种MR序列进行疾病诊断。虽然不同序列的图像在对比上有所不同,但同一患者的图像携带相似的结构信息,在神经网络重建过程中会共享部分信息。基于此种相似性生成的图像会获得不属于原对比度图像的信息。近年也有部分研究者致力于通过多对比度的输入获得更佳重建结果。Song等[59]提出一个基于相似特征捕捉和自适应字典去伪影的模型,对2种降采样图像同时进行重建。Dar等[32]提出了一种基于GAN网络的多对比MRI重建方法,输入为多通道的满采样图像与降采样图像,相比于正常的单通道重建或单通道生成,在多重对比度的输入表现出了更好的结果。Sun等[60]特征共享单元与数据保真度单元相结合组成推理块,允许在不同深度的网络上进行有效的信息传输共享,重建准确率和效率优于单对比度方法。Yan等[61]提出一种基于模型驱动的注意力网络,设计交叉对比先验的重构模型,利用采样时间较短的图像指导重建高度欠采样图像。在其他的一些研究中[62, 63, 64, 65, 66, 67, 68],多对比度图像重建算法展现出极高的重建质量和效率,将成为未来更高降采倍数下重建算法的发展方向。
基于深度学习的重建算法期望能够获得降采图像到非降采图像之间的映射关系。由于目前大多数重建算法都是回顾性研究,通常使用标准图像进行傅里叶逆变换并伪降采样;而监督学习的训练集要求包含成对的输入输出。因此,大多数的研究都是基于有监督学习的训练方式。真实降采样采集的数据往往难以获得其对应的金标准全采样图像,无监督学习更适用于此种情形下的模型设计;且无监督模型的拓展性更高,不易受到数据集偏移分布的影响。Zhang等[69]使用自监督循环神经网络,提出了用于估计和校正运动伪影的4D图像重建算法。Narnhofer等[70]提出了一种迭代重建方法,使用无监督的GAN网络学习全采样数据的概率分布,在多线圈膝关节数据集上取得较好结果。其他研究者也着力于对基于无监督的重建算法的开发[33],同时半监督、弱监督等学习模式也逐渐显露出发展潜力,是未来重要的重建算法研究方向。
MR加速成像一直是MR领域研究的重点,基于信号处理的不完整K空间重建技术是其中研究的方向之一。降采样的方案可以大幅度减少扫描采集时间,但传统的重建方法计算复杂度高、耗时长。深度学习为降采样图像重建算法带来了新的发展方向,逐渐展现出在下一代快速MR成像技术中的巨大潜力。相较于传统的重建算法,深度学习提供了更快的处理速度,并且在更高的加速倍数下依然保持良好的重建效果。
从不同分类角度下,每类重建算法均具有不同的优势(图4)。数据是建立模型的核心,基于K空间数据的重建算法往往能对影像的细节信息做到较好的恢复,避免图像过于平滑;而基于图像域的算法在去噪和去降采伪影方面较为突出,这可能与深度学习在自然图像处理的特性有关。深度学习模型的训练方法则为算法提取大数据中高维信息提供指导,有监督网络的训练效果较其他方式更优,但对数据集的依赖程度及敏感度较高,需要数量大、质量高的数据集作为输入,金标准的存在也使评估阶段较为容易。无监督和半监督则将数据集对算法的影响降低,避免过拟合等因素,挖掘数据最核心的信息,但也提高了衡量算法准确率的难度。深度学习网络则是算法的核心,基于数据驱动的网络为重建带来了更多的可能性,高可塑性使其能够应用于多种任务,基于模型展开的网络能够将深度学习与传统数学模型相结合,算法的可解释性与可迁移性有了进一步的提升。
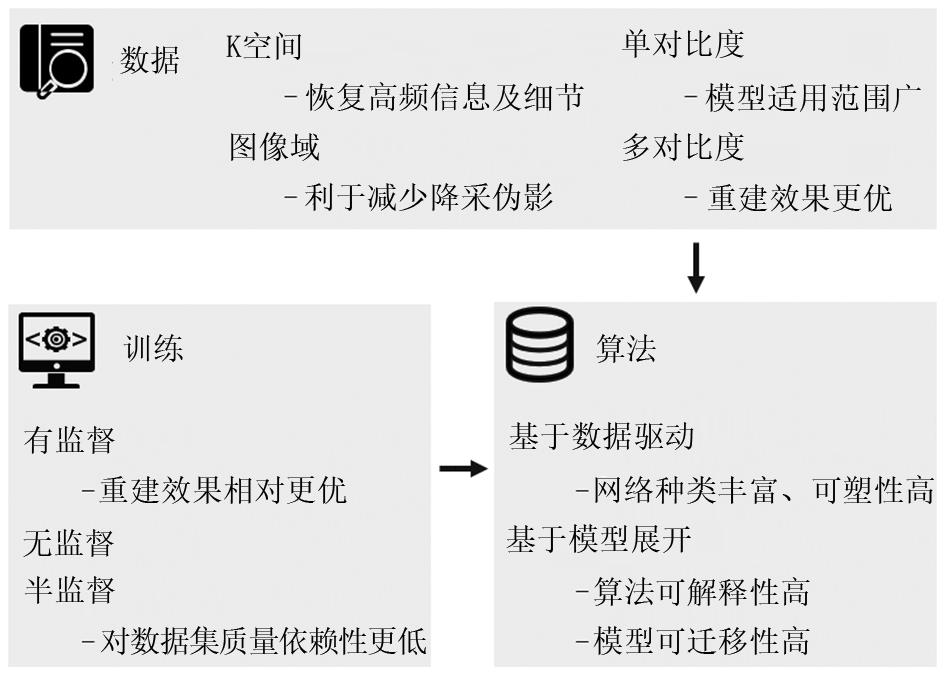

尽管目前的研究成果展现出广阔的应用前景,但依然有着很多局限性。在数据层面,当测试数据发生变化时,存在使用某一特定数据集训练的模型是否能良好地适用于其他数据集、是否需要重新进行训练的问题。端到端的模型往往对数据的一些特性依赖度高,欠采样数据和全采样数据之间的映射关系易随着数据集的变化而变化,欠缺对重建原理的解释性。基于传统迭代模型的重建算法可解释性相对较高,一些研究者也致力于提出解决低秩重建的通用算法,但仍然难以完全地解释深度学习模块的原理。
此外,大数据将为重建算法提供更高的发展空间,而目前单中心医学数据集难以达到自然图像处理项目的规模。模型的泛化能力也是评估其能力的一项指标,一些工作往往在单一的数据集上训练验证,也有工作是基于不同部位数据集甚至自然图像数据集,这从某种程度缓解了对数据集规模的依赖。同时,来自多中心的数据联合将充分挖掘模型的潜力,有助于寻找更具有数据普适性的解决方案,但多中心研究也伴随着数据隐私和安全性等问题。
如何准确地评估重建结果也是基于深度学习重建算法的一大问题,目前多采用统计层面的信噪比、相似度等指标。这些评价指标没有具体的可接受阈值,常随着MR采集参数(序列,回波时间等)变化。用于诊断的重建图像应当是稳定且准确的,仅有部分工作会基于特定疾病的数据集设计特定评估方法。因此,如何构建一套完整的、具有临床应用价值的评估方式亟待解决。



























