
版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
胆囊癌是指发生于胆囊(包括胆囊底部、体部、颈部以及胆囊管)的恶性肿瘤,是胆管系统最常见的恶性肿瘤。我国胆囊癌发病率占同期胆管疾病的0.4%~3.8%,为我国消化道肿瘤发病率第6位。胆囊癌预后差,患者5年生存率仅5%[1]。世界范围内,胆囊癌发病率的地理、种族、民族分布存在显著差异,提示遗传、环境因素在疾病发生发展中有重要作用[2]。随着近年高通量技术的快速发展,对肿瘤的分子生物学研究日趋深入,胆囊癌即在其列。目前胆囊癌的诊断与治疗尚无突破性进展,人们普遍认识到,要想真正改善胆囊癌的诊断和治疗,必须对其分子生物学特性进行充分的研究。现就胆囊癌的分子生物学研究现状和前景概述如下。
TP53是经典的抑癌基因,其在维持基因组完整性、稳定性、维持正常细胞凋亡、阻止血管新生等方面均有重要意义,TP53下调也与恶变高度相关。TP53突变在晚期肿瘤中更为多见。胆囊癌中TP53多发生错义突变。国际上报道,大约40%的胆囊癌患者存在TP53突变[3],我们对国内人群的一项研究发现47.1%胆囊癌患者存在此突变,与国际水平相仿。突变位点较为多样,研究表明5号、8号外显子突变多见,发生于31%~70%的患者中[4]。
原癌基因KRAS是与肿瘤相关众多信号通路的关键分子,KRAS激活引起肿瘤生长相关通路的过度激活。4%~13%的胆囊癌患者中发现存在KRAS基因突变[3],而我们发现对国内人群的研究发现7.8%胆囊癌患者存在此突变。胰胆管汇合异常是胆囊癌的危险因素之一,而有研究表明这一群体携带KRAS突变的频率更高。但仍有一些研究未发现胆囊癌存在KRAS突变,表明胆囊癌存在遗传异质性[5]。由腺瘤发展而来的胆囊癌中未发现KRAS突变,提示胆囊癌的癌前病变的形态学与基因表达相关[6]。胆囊癌组织中KRAS的致病突变主要位于第12、第13或第61位密码子,集中于第12位(表1)。有研究表明,第12位密码子突变是肝外胆管癌预后的独立危险因素[7]。

胆囊癌相关的主要致病基因低通量研究
胆囊癌相关的主要致病基因低通量研究
| 基因 | 主要结果 | 研究方法 | 研究人群 |
|---|---|---|---|
| TP53 | 基因突变 | PCR-RFLP,直接测序,免疫组化 | 日本、智利 |
| 基因突变、基因过表达、杂合性丢失 | PCR-RFLP,直接测序,免疫组化 | 希腊、日本、智利 | |
| KRAS | 第12位密码子突变(8%) | PCR-RFLP | 印度 |
| 第12位密码子突变(29%~30%) | PCR-RFLP | 智利 | |
| 第12位密码子突变(39%) | PCR-RFLP | 日本 | |
| 第12位密码子突变(0~59%) | PCR-RFLP,直接测序 | 日本 | |
| 第12位密码子突变(50%~80%) | PCR-SSCP,直接测序 | 日本 | |
| p16 | 基因突变、基因删除 | PCR-RFLP,直接测序,免疫组化 | 希腊、日本、智利 |
原癌基因ERBB2(又称HER2)属于表皮生长因子家族,具酪氨酸激酶活性。我们在国际上率先发现11.8%胆囊癌患者存在ERBB2基因突变[8]。
同时,我们团队利用转基因小鼠模型,证明了胆管上皮细胞基底膜中ERBB2过表达致使所有受试小鼠发展至胆囊癌。此后国际范围对该分子与胆囊癌的关系表明,ERBB2在高分化Ⅱ~Ⅳ期胆囊癌的阳性率约为9.4%;ERBB2在晚期胆囊癌中阳性率约14%。在进行吉西他滨化疗的胆囊癌患者中,EGFR/HER2通路靶向治疗可以改善患者预后。我们通过一系列体内体外实验发现ERBB2在胆囊癌发生发展中有重要作用。由于胆囊癌的KRAS突变在国内人群较为少见,ERBB2对国内人群胆囊癌发生发展的作用尤为明显。ERBB2在胆囊癌中的分子机制的阐明对理解胆囊癌有着关键作用,以此为治疗靶点的药物开发也存在着广阔的前景。目前多种ERBB受体的靶向药物已在我院处于临床试验阶段[6]。
微阵列技术作为高通量技术的一种,可以同时检测上千种分子的表达情况,建立全基因表达谱。胆囊癌相关的基因表达谱研究总结参见表2。
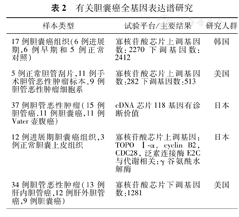
有关胆囊癌全基因表达谱研究
有关胆囊癌全基因表达谱研究
| 样本类型 | 试验平台/主要结果 | 研究人群 |
|---|---|---|
| 17例胆囊癌组织(6例进展期,6例早期和5例正常对照) | 寡核苷酸芯片上调基因数:2270下调基因数:2412 | 韩国 |
| 5例正常胆管刮片,11例手术胆管恶性肿瘤标本,9例胆管恶性肿瘤细胞系 | 寡核苷酸芯片上调基因数:282下调基因数:513 | 美国 |
| 37例胆管恶性肿瘤(15例胆管癌,11例胆囊癌,11例Vater壶腹癌) | cDNA芯片118基因有诊断价值 | 日本 |
| 12例进展期胆囊癌组织,3例正常胆囊上皮组织 | 寡核苷酸芯片上调基因:TOPO Ⅰ-α,cyclin B2, CDC28,泛素连接酶E2C与代谢相关:γ谷氨酰水解酶 | 日本 |
| 34例胆管恶性肿瘤(13例肝内胆管癌,12例肝外胆管癌,9例胆囊癌) | 寡核苷酸芯片下调基因数:1281 | 美国 |
近年来高通量测序技术的发展降低了测序成本,使得同时对上万个特定基因进行序列测定和突变检测成为可能,成本效益好。然而胆囊癌领域中基于高通量测序的研究还较为有限。我们基于国内人群的全外显子测序研究首先发现,胆囊癌细胞ErbB通路多种基因突变频率较高[8]。Javle等[10]发现了胆囊癌TP53和PIK3CA中26个无义突变位点。一项基于印度人群的研究发现胆囊癌细胞中PIK3CA和KRAS通路基因突变在当地人群中常见[11]。我们认为几项研究结果的差异表明了胆囊癌本身的遗传异质性。虽然疾病表现相似,但是其发生发展过程内在的分子机制存在差异。胆囊癌高通量测序研究总结参见表3。

基于高通量测序技术发现的胆囊癌相关基因突变研究
基于高通量测序技术发现的胆囊癌相关基因突变研究
| 试验平台 | 样本量(例) | 研究人群 | 目标基因 | 主要结果 |
|---|---|---|---|---|
| MassARRAY质谱分析 | 石蜡样本49 | 印度 | 30个基因上的390个突变 | PIK3CA (4%), KRAS (2%), CTNNB1 (4%), TP53 (18%) |
| 质谱分析 | 石蜡样本57 | 美国安德森癌症中心 | 33个基因上的159个突变 | 9个样本中14个高发突变位点,影响基因包括KRAS,NRAS,PIK3CA,IDH1,ALK和MET;15个样本中26个突变位点 |
| 二代测序 | 石蜡样本15 | 美国安德森癌症中心 | 182个肿瘤相关基因 | P53,STK11,RICTOR,TSC2,FGF3-TACC融合,FGF10扩增等;PI3K通路 |
| 全外显子测序 | 冰冻样本29 | 日本 | 64个非同义突变 | EGFR, ERBB3, PTEN, ARID2, MLL2, MLL3, APOBEC, TERT APOBEC |
| 外显子测序+目的 基因测序 | 冰冻样本57 | 中国 | 全外显子测序 | TP53(47.1%), KRAS(7.8%)和ERBB3 (11.8%);ERBB通路基因 |
胆囊癌相关的单基因研究数量较多,现有研究结果差异较大。虽然包括我们在内的很多课题组都在此方向进行了大量研究,但相关的诊断、治疗分子靶点研究仍缺乏深入性、系统性,相关成果向临床应用的转化率还是偏低。
杂合性丢失(LOH)即一对杂合的等位基因变成纯合状态,是肿瘤基因组中的一种常见改变。其现象可能来源于基因删除,父源或母源染色体或染色体局部的重复,或因各种染色改变引起。目前发现胆囊癌中杂合性丢失发生在18个不同的染色体区域,包括8p、9p、22q等[12]。这些染色体区域的杂合性丢失与视网膜母细胞瘤、黑色素瘤、喉鳞状细胞癌等相关。另外,在3号染色体短臂有多处编码抑癌基因的位点发生杂合性丢失[13]。一项对日本人群的研究发现一些杂合性丢失与胰胆管汇合异常相关。有研究[6]发现约10%的胆囊癌患者表现高度微卫星不稳定(MSI-H)。胆囊癌相关的杂合性丢失与微卫星不稳定现象研究总结详见表4。此方向上的几项研究仍处于描述性分析阶段,缺乏高质量的研究成果阐明胆囊癌的分子生物学机制。

有关胆囊癌相关的杂合性丢失(LOH)、微卫星不稳定(MSI)研究
有关胆囊癌相关的杂合性丢失(LOH)、微卫星不稳定(MSI)研究
| 杂合性丢失/微卫星不稳定位点 | 研究人群 |
|---|---|
| LOH见于:3p, 6q, 7q, 8p, 9p, 9q, 11q, 12q, 17p, 18q, 19p, 22q和Xq | 智利 |
| LOH见于:2p, 4p, 4q, 8q, 9q, 10p, 14p, 14q, 16p, 19p, 21p和Xp,其中2p24, 14q22和21q22多发 | 日本 |
| p53, 9p.8p, DCC, KRAS, p16, 16q24, 3p,9q, 22q和p161NK4 | 智利、日本 |
| BAT-26 | 希腊 |
| MSI报道于20%~33%病例 | 智利、日本 |
| E-钙黏素相关:2p, 2q, 6q, 7q, 17p | 印度 |
| FHIT相关的MSI-H报道于17.5%病例;FHIT相关的LOH报道于27.5%病例 | 印度 |
| LOH见于1p36 (53%), 9p21 (38%), 13q14 (56%), 16q24 (61%)和17p13 (42%) | 日本 |
| FHIT基因相关位点3p14.2 | 智利 |
| LOH见于3p12, 3p14.2, 5q21, 9p21, 9q, 13q, 17p13和18q,与抑癌基因(DUTT1, FHIT, APC, p16, FCMD, RB1, p53和DCC)相关 | 印度 |
| 基因组不稳定性多见于2p, 2q, 6q, 7q和17p | 印度 |
| DUTT1 (3p12), FHIT (3p14.2), BLU, RASSF1A, SEMA3B和hMLH1 (3p21.3) | 智利 |
抑癌基因启动子的过度甲基化是肿瘤发生发展的重要表观遗传学过程,DNA甲基化状态与肿瘤的诊断、治疗密切相关。一项纳入109例晚期胆囊癌患者的研究发现,p73、MGMT和DCL1基因的甲基化与胆囊癌的生存率相关,而见于其他恶性疾病的CDH13、FHIT基因启动子甲基化则未显示与预后相关[14]。多因素统计分析表明,甲基转移蛋白MGMT的表达水平是胆囊癌的独立预后因素[14]。近期的一项研究中发现,CDH1、CDKN2A-p16、REPRIMO和UCHL1基因启动子甲基化在胆囊癌的发生发展中有重要作用[15]。其他研究见表5。

胆囊癌相关的启动子甲基化研究
胆囊癌相关的启动子甲基化研究
| 基因 | 功能 | 甲基化水平 | 研究人群 |
|---|---|---|---|
| CDH1 | 细胞黏附 | 11%~65% | 日本、智利 |
| FHIT | 调控DNA复制与凋亡 | 30%~57% | 智利 |
| APC | 抑癌基因(与细胞迁 移、黏附、凋亡相关) | 26%~35% | 智利、美国 |
| hMLH1 | 错配修复 | 0~14% | 智利、美国 |
| p16 | 细胞周期调节 | 15%~60% | 智利、美国、德国 |
| p15 | 细胞周期调节 | 22%~44% | 智利 |
| DAPK1 | 丝氨酸-苏氨酸激酶 | 8%~61% | 日本、智利 |
| DLC1 | 激活GTP酶 | 39% | 智利 |
| RASSF1 | 信号转导 | 0~36% | 日本、智利、韩国 |
| MGMT | 甲基转移酶 | 13%~30% | 智利、美国 |
此外,几项研究表明,3p(100%)、8p (100%)、9q (88%)、22q (92%)几个染色体区域过度甲基化在胆囊癌中发生率较高,这些区域与抑癌基因的表达相关。一些经典的抑癌基因表达于3p、5q、8p、13q、18q等染色体区域,其甲基化状态也可能与胆囊癌发生发展相关[6]。
候选基因法用于外显率较低基因的研究,一般通过病例对照研究,对实验组与对照组候选基因外显率进行统计分析,发现与疾病相关性。候选基因往往选择已知通路上的关键基因,通路如凋亡相关、细胞周期相关、DNA修复相关、炎症、物质代谢或激素相关等与肿瘤发生发展相关的通路。此外,一些基因也与疾病的遗传性有关。
目前大多被研究的蛋白都是一些经典通路上的限速酶,以及与脂代谢、类固醇合成、脂质转运、胆汁酸合成、胆管转运、胆囊收缩、细胞周期、DNA修复或炎症相关的蛋白。胆囊癌领域中,已发现重复性较好的基因多态性包括OGG1rs1052133, TP53rs1042522, GSTM1的无效基因多态性和CYP1A1rs1048943基因多态性等,然而由于研究尚少,仍有待进一步证实。
一项胆囊癌全基因组关联分析发现,DCC基因上rs7504990位点的单核苷酸多态性在日本人群中增加6倍胆囊癌患病风险。而此前直肠癌中的研究表明,DCC基因低表达与肿瘤生长、分化、转移均密切相关[16]。但另一项基于印度人群的全基因组关联分析并未发现rs7504990位点与胆囊癌的相关性,但对DCC基因的单体型分析发现Grs2229080-Ars4078288-Crs7504990 Ars714单体型在胆囊癌患病中有累积效应[17]。
至今胆囊癌仍缺少可靠的肿瘤标志物。目前所应用的癌胚抗原(CEA)和糖类抗原19-9(CA19-9)通常在晚期胆囊癌中升高,但缺乏特异性,因此诊断效力不强[18]。其他肿瘤标志物,如CA242、CA125等,也曾有相关研究,但结果差异较大。一些有报道的其他分子标志物包括RCAS1、CA15-3、Mac-2BP、CYFRA21-1,但临床应用价值仍未得到确证。
胆囊癌的发生发展与环境、遗传因素密切相关,分子生物学机制复杂。由于发病率低,标本量小,目前对胆囊癌研究的开展相较于胃癌、胰腺癌等仍较少,内容上深度尚显不足。单基因研究受限于病例偏少,样本不足,证据效力普遍偏低。基于高通量技术的研究目前也较有限。对胆囊癌分子机制的有限了解使得近年在胆囊癌的诊断、治疗方面尚无突破性进展。
我们最终对胆囊癌治疗的突破很可能依赖于新药的开发和基因治疗的成熟,而新药的开发和基因治疗的成熟又必将依赖于对胆囊癌发病机制的认识,要认识胆囊癌的发病机制,必然要研究胆囊癌的生物学特性,所以胆囊癌的最终解决离不开基础研究。我们团队多年来致力于胆囊癌的精准及靶向治疗,国际上首次报道胆囊癌相关外显子突变谱,发现ErbB信号通路突变与胆囊癌进展密切相关。并在这基础上,对我国自主研发的第二代不可逆的酪氨酸激酶抑制剂,主要作用于ErbB家族相关靶点的新药进行Ⅰ期临床试验,为我们的后续研究以及开展对胆囊癌的治疗新方法的探索提供了条件。
新兴技术的应用也为理解胆囊癌的分子生物学机制提供新的实验依据。利用单克隆抗体治疗胃癌,结肠癌等消化道肿瘤已初见成效。随着基因组研究技术的不断发展以及基因芯片技术的改进,我们可以根据病理标本进行基因表达,药敏试验和靶向药物靶点检测等分析。目前,国内多家临床研究中心正在开展胆囊癌一线化疗药物和分子靶向药物的临床研究,以期将胆囊癌个体化治疗在将来成为可能。
随着实验技术的不断更新发展,各临床中心的勉力合作,相信对胆囊癌的复杂分生物学研究会不断深入,胆囊癌的早期诊治水平一定会上一个更高的台阶,并可作为诊断,预后判断和评价肿瘤生物学行为的客观指标,尤其对指导临床采用基因治疗提供依据具有更重要的意义。
本综述对胆囊癌相关的流行病学、发病机制和分子生物学的进展进行了系统的总结,希望有助于本领域研究者了解胆囊癌研究现状,为进一步研究打下基础。


























