
使用布鲁克基质辅助激光解吸电离飞行时间质谱仪对临床分离的病原菌进行鉴定并评价其准确性。
收集福建医科大学附属第一医院2015年11月至2016年12月分离得到的21 270株病原菌,分别以布鲁克质谱仪以及VITEK-Ⅱ/API表型鉴定系统进行鉴定,并以分子生物学结果为金标准,验证鉴定结果的准确性。
对临床常见病原菌,质谱系统的检出率>95%、表型系统的检出率>90%,具有较高的一致性。对43株厌氧菌,质谱有90.7%能鉴定到种,97.7%被鉴定到属。表型系统有65.1%被鉴定到种,69.8%被鉴定到属,差异有统计学意义(χ2=6.76,P<0.01);1 558株真菌中,丝孢酵母和念珠菌的检出率差异均无统计学意义(均P>0.05),质谱对78株丝状真菌鉴定的准确率为76%;18株放线菌、诺卡菌、分枝杆菌和军团菌都能被鉴定到属。
蛋白质谱鉴定技术操作简单、灵敏度高,能够对临床分离的常见病原菌、厌氧菌、真菌等提供快速、准确的鉴定。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
基质辅助激光解吸电离飞行时间质谱(MALDI-TOF)作为一种快速的微生物鉴定新方法,可广泛应用于临床诊断、院感监测、环境检测以及菌种分类的研究中。而VITEK-Ⅱ是目前临床实验室较为常用的一种全自动微生物鉴定和药敏系统,它与API(Analytic Products INC)鉴定系统都属于表型鉴定方法。本研究旨在全面了解质谱仪对临床分离病原菌的鉴定能力。
收集福建医科大学附属第一医院2015年11月至2016年12月来自临床各种标本的分离株,按要求进行接种、分离培养和染色镜检,菌种保存于-80 ℃。
大肠埃希菌ATCC25922、金黄色葡萄球菌ATCC29213、铜绿假单胞菌ATCC27853。质谱仪质控品为IVD BTS溶液。
革兰染液、抗酸染液(台湾BASO公司);沙保罗培养基、马铃薯葡萄糖琼脂培养基(郑州贝瑞特公司);哥伦比亚血琼脂培养基、巧克力营养琼脂、VITEK-Ⅱ全自动微生物鉴定系统及配套的鉴定卡以及API微生物鉴定系统及配套的试剂(法国梅里埃公司);MALDI-TOF及配套的试剂和靶板(德国Bruker公司及美国Sigma公司);16sRNA引物及ITS1/ITS4引物由上海生工公司合成,PCR反应体系使用美国Promega公司产品。PCR产物由上海生工公司进行测序,在NCBI库中进行Blast比对后确定菌株。
依据分离菌的菌落形态、染色结果等选择相应的鉴定卡(包括阳性菌、阴性菌、苛养菌和酵母菌,但不包括丝状真菌)。根据产品操作手册,选取经纯培养的待测菌落配制菌悬液进行检测,获取结果所需时间为3~10 h,鉴定结果由系统自动判读。采用数值分类法进行计算,检测值(T)≥89%时,鉴定可靠。
依据分离菌的菌落形态、染色结果、预方向实验等选择相应的鉴定条,包括阳性菌、阴性菌、苛养菌、厌氧菌和棒状菌。根据产品操作手册,配制相应麦氏浊度的菌悬液进行检测,获取结果所需时间为4~24 h,检测值(ID)≥89%,鉴定可靠。
根据仪器说明书与《中国临床微生物质谱应用专家共识》[1,2]进行质量控制和操作。待测菌分别接种于哥伦比亚血平板、巧克力营养琼脂、沙保罗培养基、马铃薯葡萄糖琼脂培养基等适宜的培养基上,以适宜的培养条件孵育至菌株生长最茂盛时。用无菌牙签蘸取少量的单个待检菌点样到靶板上,彻底干燥后将1 μl甲酸涂敷于样品点上,等彻底干燥后再覆盖1 μl HCCA基质液,室温干燥后进行检测。对于常规方法难以获得结果的目标菌采用乙醇/甲酸萃取法来制备样本:挑取单个菌落(丝状真菌需尽量挑取菌丝并尽量避免孢子和色素产生)放入盛有300 μl超纯水的Eppendorf管内吹打混匀,再加入900 μl无水乙醇混匀。离心后去除上清。在沉淀颗粒中加入50 μl 70%的甲酸吹打混匀,再加入50 μl的乙腈制成混合悬液。离心后吸取1 μl上清液到靶板上,干燥后再覆盖1 μl HCCA基质液,室温干燥后进行检测;抗酸菌采用0.5 mm氧化锆珠处理后获得分析样品。检测结果由MALDI Biotyper IVD2.3(5989)分析软件自动判读。鉴定分值≥2.0分时,鉴定结果到种水平;1.7分≤分值<2.0分时鉴定到属水平;分值<1.7分时鉴定结果不可靠。利用MALDI Biotyper进行鉴定所需时间大约为30 min。当表型系统的检测值<89%或质谱鉴定分值<2.0分时,需重新分纯鉴定,质谱系统另采用乙醇/甲酸萃取法、氧化锆珠法进行鉴定。鉴定或复检结果的检测值≥89%(表型系统)且≥2.0分(质谱系统),两者一致结果确认;均无可靠的鉴定结果就判读为不可鉴定。
对于MALDI Biotyper与VITEK-Ⅱ、API系统的鉴定结果不一致;或MALDI Biotyper鉴定分值<2.0分且VITEK-Ⅱ、API系统的概率百分比<89%的分离菌采用分子生物学的方法进行最终确认。使用核酸提取试剂盒提取相应菌株的DNA后进行PCR扩增[3,4]。扩增产物经纯化后送上海生工进行双向测序,测序结果拼接后利用BLAST进行比对从而确定鉴定结果。利用分子生物学获取结果所需时间至少为3 d。
采用SPSS Statistics 24.0进行统计学分析,两种鉴定系统的检出率比较采用χ2检验,Tmin<1或N<40时用Fisher精确概率法。P<0.05为差异具有统计学意义。
共收集21 270株分离株,分属于162个菌属,其中阳性菌5 712株,阴性菌13 941株,厌氧菌43株,真菌1 558株,放线菌、诺卡菌、分枝杆菌、军团菌等17株(表1)。采用分子生物学方法复核的菌株共936株,包括常见菌846株、厌氧菌17株、酵母菌37株、丝状真菌18株以及少见菌(分离率<1%)18株。
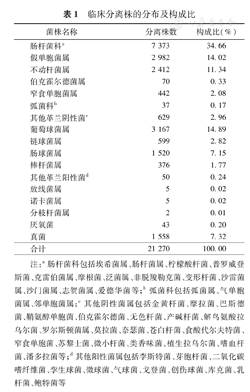
临床分离株的分布及构成比
临床分离株的分布及构成比
| 菌株名称 | 分离株数 | 构成比(%) |
|---|---|---|
| 肠杆菌科a | 7 373 | 34.66 |
| 假单胞菌属 | 2 982 | 14.02 |
| 不动杆菌属 | 2 412 | 11.34 |
| 伯克霍尔德菌属 | 70 | 0.33 |
| 窄食单胞菌属 | 442 | 2.08 |
| 弧菌科b | 37 | 0.17 |
| 其他革兰阴性菌c | 629 | 2.96 |
| 葡萄球菌属 | 3 167 | 14.89 |
| 链球菌属 | 599 | 2.82 |
| 肠球菌属 | 1 520 | 7.15 |
| 棒杆菌属 | 376 | 1.77 |
| 其他革兰阳性菌d | 50 | 0.24 |
| 放线菌属 | 5 | 0.02 |
| 诺卡菌属 | 5 | 0.02 |
| 分枝杆菌属 | 2 | 0.01 |
| 厌氧菌 | 43 | 0.20 |
| 真菌 | 1 558 | 7.32 |
| 合计 | 21 270 | 100.00 |
注:a肠杆菌科包括埃希菌属、肠杆菌属、柠檬酸杆菌、普罗威登斯菌、克雷伯菌属、摩根菌、泛菌属、非脱羧勒克菌、变形杆菌、沙雷菌属、沙门菌属、志贺菌属、爱德华菌等;b弧菌科包括弧菌属、气单胞菌属、邻单胞菌属;c其他阴性菌属包括金黄杆菌、摩拉菌、巴斯德菌、鞘氨醇单胞菌、伯克霍尔德菌、无色杆菌、产碱杆菌、解鸟氨酸拉乌尔菌、罗尔斯顿菌属、莫拉菌、奈瑟菌、苍白杆菌、食酸代尔夫特菌、窄食单胞菌、苏黎士菌、微小杆菌、类香味菌、植生拉乌尔菌、嗜血杆菌、潘多拉菌等;d其他阳性菌属包括李斯特菌、芽胞杆菌、二氧化碳嗜纤维菌、孪生球菌、微球菌、气球菌、戈登菌、创伤球菌、库克菌、乳杆菌、鲍特菌等
对表1中的临床分离菌,除了真菌,MALDI Biotyper对放线菌属、诺卡菌属、分枝杆菌属的检出率>95%,表型鉴定系统(VITEK-Ⅱ及API)的检出率>91%。对埃希菌属、枸橼酸杆菌属、沙门菌属、伯克霍尔德菌属、窄食单胞菌属、葡萄球菌属、肠球菌属及链球菌属,两种鉴定系统的检出率差异无统计学意义(表2)。对于肠杆菌及不动杆菌VITEK-Ⅱ的鉴定结果多为复合菌群,MALDI Biotyper能够鉴定出约23种不动杆菌以及5种肠杆菌,检出率差异有统计学意义;除了对纹带棒杆菌(MALDI和APIcorney)的鉴定存在高度一致外,MALDI Biotyper能够更准确地对嗜血杆菌及其他棒状杆菌进行鉴定。VITEK-Ⅱ/API 20NE无法区分嗜水/豚鼠气单胞菌,需补充赖氨酸脱羧酶实验进行确认;而MALDI Biotyper不能区分志贺菌和大肠埃希菌;有18种细菌是由MALDI Biotyper完成的鉴定,包括神户肠杆菌、耳炎假单胞菌、黄褐假单胞菌、极端东方化假单胞菌、拜尔利氏不动杆菌、医院不动杆菌、食酸代尔夫特菌、苏黎士菌、结核硬脂酸棒杆菌、溶血嗜血菌、pettenkoferi葡萄球菌、小肠肠球菌、苏云金芽孢杆菌、ovis创伤球菌、居肺潘多拉菌、乳酪短杆菌、Corynebacteriumfalsenii和Herbaspirillumaquaticum。
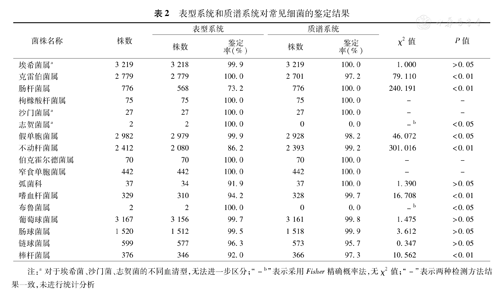
表型系统和质谱系统对常见细菌的鉴定结果
表型系统和质谱系统对常见细菌的鉴定结果
| 菌株名称 | 株数 | 表型系统 | 质谱系统 | χ2值 | P值 | ||
|---|---|---|---|---|---|---|---|
| 株数 | 鉴定率(%) | 株数 | 鉴定率(%) | ||||
| 埃希菌属a | 3 219 | 3 218 | 99.9 | 3 219 | 100.0 | 1.000 | >0.05 |
| 克雷伯菌属 | 2 779 | 2 779 | 100.0 | 2 701 | 97.2 | 79.110 | <0.01 |
| 肠杆菌属 | 776 | 568 | 73.2 | 776 | 100.0 | 240.191 | <0.01 |
| 枸橼酸杆菌属 | 75 | 75 | 100.0 | 75 | 100.0 | - | - |
| 沙门菌属a | 27 | 27 | 100.0 | 27 | 100.0 | - | - |
| 志贺菌属a | 2 | 2 | 100.0 | 0 | 0.0 | -b | <0.05 |
| 假单胞菌属 | 2 982 | 2 979 | 99.9 | 2 928 | 98.2 | 46.072 | <0.05 |
| 不动杆菌属 | 2 412 | 2 080 | 86.2 | 2 393 | 99.2 | 301.016 | <0.01 |
| 伯克霍尔德菌属 | 70 | 70 | 100.0 | 70 | 100.0 | - | - |
| 窄食单胞菌属 | 442 | 442 | 100.0 | 442 | 100.0 | - | - |
| 弧菌科 | 37 | 34 | 91.9 | 37 | 100.0 | 1.390 | >0.05 |
| 嗜血杆菌属 | 329 | 310 | 94.2 | 328 | 99.7 | 16.708 | <0.01 |
| 布鲁菌属 | 2 | 2 | 100.0 | 0 | 0.0 | -b | <0.05 |
| 葡萄球菌属 | 3 167 | 3 156 | 99.7 | 3 161 | 99.8 | 1.475 | >0.05 |
| 肠球菌属 | 1 520 | 1 512 | 99.5 | 1 518 | 99.9 | 3.612 | >0.05 |
| 链球菌属 | 599 | 577 | 96.3 | 573 | 95.7 | 0.347 | >0.05 |
| 棒杆菌属 | 376 | 346 | 92.0 | 366 | 97.3 | 10.562 | <0.01 |
注:a对于埃希菌、沙门菌、志贺菌的不同血清型,无法进一步区分;"-b"表示采用Fisher精确概率法,无χ2值;"-"表示两种检测方法结果一致,未进行统计分析
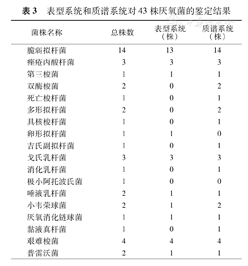
表型系统和质谱系统对43株厌氧菌的鉴定结果
表型系统和质谱系统对43株厌氧菌的鉴定结果
| 菌株名称 | 总株数 | 表型系统(株) | 质谱系统(株) |
|---|---|---|---|
| 脆弱拟杆菌 | 14 | 13 | 14 |
| 痤疮丙酸杆菌 | 3 | 3 | 3 |
| 第三梭菌 | 1 | 1 | 1 |
| 双酶梭菌 | 2 | 0 | 2 |
| 死亡梭杆菌 | 1 | 0 | 1 |
| 多形拟杆菌 | 2 | 0 | 2 |
| 具核梭杆菌 | 1 | 0 | 1 |
| 卵形拟杆菌 | 1 | 1 | 0 |
| 吉氏副拟杆菌 | 1 | 0 | 1 |
| 戈氏乳杆菌 | 3 | 3 | 3 |
| 消化乳杆菌 | 1 | 0 | 1 |
| 极小阿托波氏菌 | 1 | 0 | 0 |
| 唾液乳杆菌 | 2 | 1 | 1 |
| 小韦荣球菌 | 2 | 1 | 2 |
| 厌氧消化链球菌 | 1 | 1 | 1 |
| 黏液真杆菌 | 1 | 0 | 1 |
| 艰难梭菌 | 4 | 4 | 4 |
| 普雷沃菌 | 2 | 1 | 1 |
对于43株厌氧菌,两个系统的检出率差异有统计学意义(χ2=6.76,P<0.01)。质谱有90.7%鉴定到种,97.7%被鉴定到属,表型系统有65.1%鉴定到种,69.8%被鉴定到属。分子生物学方法检测17株,与表型系统一致的有2株,与质谱系统一致的有14株。其中双酶梭菌、死亡梭杆菌、多形拟杆菌、吉氏副拟杆菌、具核梭杆菌、消化乳杆菌、黏液真杆菌都是通过MALDI Biotyper完成的鉴定。MALDI Biotyper未能鉴定到种水平的有卵形拟杆菌、极小阿托波氏菌各1株,但在属水平均能准确鉴定。另有1株Prevotellaheparinolytica未得到正确鉴定。
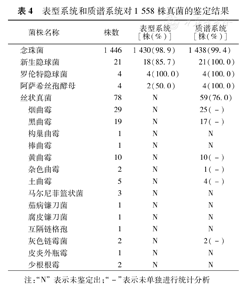
表型系统和质谱系统对1 558株真菌的鉴定结果
表型系统和质谱系统对1 558株真菌的鉴定结果
| 菌株名称 | 株数 | 表型系统[株(%)] | 质谱系统[株(%)] | ||
|---|---|---|---|---|---|
| 念珠菌 | 1 446 | 1 430(98.9) | 1 438 | (99.4) | |
| 新生隐球菌 | 21 | 18(85.7) | 21 | (100.0) | |
| 罗伦特隐球菌 | 4 | 4(100.0) | 4 | (100.0) | |
| 阿萨希丝孢酵母 | 4 | 2(50.0) | 4 | (100.0) | |
| 丝状真菌 | 78 | N | 59(76.0) | ||
| 烟曲霉 | 29 | N | 25(-) | ||
| 黑曲霉 | 19 | N | 17(-) | ||
| 构巢曲霉 | 1 | N | N | ||
| 棒曲霉 | 1 | N | N | ||
| 黄曲霉 | 10 | N | 10(-) | ||
| 杂色曲霉 | 2 | N | 1(-) | ||
| 土曲霉 | 5 | N | 4(-) | ||
| 马尔尼菲篮状菌 | 3 | N | N | ||
| 茄病镰刀菌 | 1 | N | N | ||
| 腐皮镰刀菌 | 1 | N | N | ||
| 互隔链格孢 | 1 | N | N | ||
| 灰色链霉菌 | 2 | N | 2(-) | ||
| 皮炎外瓶霉 | 1 | N | N | ||
| 少根根霉 | 2 | N | N | ||
注:"N"表示未鉴定出;"-"表示未单独进行统计分析
共1 558株真菌,两种系统对丝孢酵母和念珠菌的检出率均>95%,差异无统计学意义(χ2=2.689,1.436,均P>0.05)。但是VITEK-Ⅱ容易将新生隐球菌鉴定为罗伦特隐球菌,需要用API 20C AUX进行进一步确认。MALDI Biotyper对78株丝状真菌的鉴定率为76%,它能够鉴定部分曲霉菌、青霉菌、链霉菌,对链格孢、马儿尼菲篮状菌、茄病镰刀菌、腐皮镰刀菌、毛霉和根霉等依然需要借助形态学以及分子生物学方法进行鉴定。
两个系统对18株少见菌(分离率<1%)均能鉴定到属水平(表5)。经分子生物学复核,MALDI Biotyper能鉴定到种水平的共有8种11株菌(62.5%)。表型系统能够检出2株奴卡菌(12.5%)。均无法鉴定的有5株(28%)。
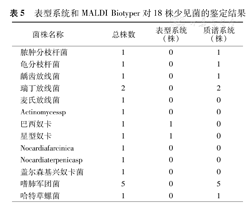
表型系统和MALDI Biotyper对18株少见菌的鉴定结果
表型系统和MALDI Biotyper对18株少见菌的鉴定结果
| 菌株名称 | 总株数 | 表型系统(株) | 质谱系统(株) |
|---|---|---|---|
| 脓肿分枝杆菌 | 1 | 0 | 1 |
| 龟分枝杆菌 | 1 | 0 | 1 |
| 龋齿放线菌 | 1 | 0 | 1 |
| 瑞丁放线菌 | 2 | 0 | 2 |
| 麦氏放线菌 | 1 | 0 | 0 |
| Actinomycessp | 1 | 0 | 0 |
| 巴西奴卡 | 1 | 1 | 0 |
| 星型奴卡 | 1 | 1 | 0 |
| Nocardiafarcinica | 1 | 0 | 0 |
| Nocardiaterpenicasp | 1 | 0 | 0 |
| 盖尔森基兴奴卡菌 | 1 | 0 | 0 |
| 嗜肺军团菌 | 5 | 0 | 5 |
| 哈特草螺菌 | 1 | 0 | 1 |
及时、准确的病原学诊断能够帮助临床医生选择合适的抗感染药物从而降低病死率,缩短住院时间。但目前临床微生物室的常规检测方法远不能满足临床需要。传统鉴定方法主要有手工法和仪器法,是依据待测菌的生化反应结果进行鉴定,具有较好的稳定性和重复性[5],且自动化仪器进行鉴定的同时还可进行药敏实验,比较方便、高效;基质辅助激光解吸电离飞行时间质谱鉴定技术的出现提供了完全不同的鉴定方法[6],其原理是获取待测菌的蛋白质指纹谱图后通过软件进行处理,再和数据库中已知微生物的标准图谱进行比对鉴定;而基于分子生物学技术的病原微生物鉴定常选择通用引物16sRNA和ITS进行细菌和真菌的鉴定[3]。
本次研究中,表型系统和质谱都能准确鉴定临床95%以上的分离株,对常见细菌具有较高的准确性和重复性。对埃希菌属、沙门菌属、伯克霍尔德菌属、窄食单胞菌属、葡萄球菌属及肠球菌属等,两种鉴定系统的检出率差异无统计学意义。对形态特殊的细菌也都能够进行较好地鉴定,如小菌落突变株金黄色葡萄球菌、粗糙型沙门菌、栖稻黄单胞菌和黏液型铜绿假单胞菌。
表型系统VITEK和API的数据库依然存在一定的局限性,对在适宜培养条件下、处于对数生长期的快生长菌、生化反应典型的菌株鉴定效果较好;不同种属细菌的生长周期、培养条件及产酶能力各不相同,其检出率有所差异。比如易将鹑鸡肠球菌判断为铅黄肠球菌,无法区分流感嗜血杆菌及溶血嗜血杆菌,对部分菌株的鉴定需要补充实验,对复合菌群的鉴别能力较弱等。为获得理想的鉴定效果,需要借助预实验选择最为合适的试剂条/卡;芽孢杆菌BAC卡、厌氧菌/棒杆菌的ANC卡目前仅限于科研或者农业领域,也限制了其鉴定范围;操作时菌株不单纯、生长状态不适宜、菌液浊度不标准、读数头出现问题等均会影响结果。因此使用表型系统时,如果遇到少见菌、疑难菌、检测值<89%、结果与菌落形态或镜下形态差别较大、或与典型生化表型不符时,应综合分析、重新鉴定、宜采用多种方法加以确定,以免造成错误。
而MALDI Biotyper数据库里包含了2 000多种微生物的特征指纹图谱,鉴定范围超过了现有的表型鉴定系统。本研究中得到的多数分离菌的检出率与文献报道基本一致[1,2,7,8,9];共有25种(203株)分离菌是通过MALDI Biotyper完成的鉴定;对阴沟肠杆菌/不动杆菌等复合菌群的鉴别、尤其对厌氧菌(90.7%,65.1%)和分枝杆菌、放线菌、军团菌等少见菌(62.5%,12.5%)的鉴定能力都明显优于表型系统(P<0.05);在真菌方面则主要是用于酵母菌的检测,对部分曲霉菌、青霉、链霉菌由一定的鉴定能力;对于培养条件比较苛刻,生长速度缓慢的分离菌,MALDI Biotyper能够显著缩短鉴定时间:如产单核李斯特菌、阴道加德纳菌、隐球菌、军团菌、艰难梭菌、脓肿分枝杆菌等。
但是对没有纳入现有IVD2.3标准数据库的罕见菌种或新型病原体,如布鲁菌[10]、Prevotellaheparinolytica、Actinomycessp、Nocardiafarcinica、Nocardiaterpenicasp、盖尔森基兴奴卡菌等,以及马尔尼菲篮状菌、茄病镰刀菌、毛霉等大量的丝状真菌都无法进行鉴定。此外不同地区存在的多样性菌株没有被收录,也会影响鉴定的准确性。有报道通过自建库提高了13%的鉴定率[11]。自建库有严格的要求和操作,还需要质量控制和"金标准"[2]。通过购买和升级数据库、不断累积本地自建数据库都可使实验室质谱系统鉴定的范围和准确性不断提高。
而同一菌属中相近的菌种以及当菌株的进化关系很近,指纹图谱差异较小时其鉴别能力也会下降,如肺炎/缓症链球菌、路登葡萄球菌/金黄色葡萄球菌、大肠埃希菌/志贺菌、麦氏弧菌/溶藻弧菌、枯草芽孢杆菌/蜡样芽胞杆菌、星型奴卡菌/奴卡菌、近平滑复合群[12]都易出现鉴定错误或难以鉴别,进行手工实验和分子生物学方法的补充与复核,依然是不可缺少的。
除了数据库的质量,菌株的蛋白提取效果对质谱鉴定是至关重要的。虽然质谱系统测定的是高表达、稳定的核糖体蛋白,受生长环境和状态的影响相对较小。但本研究也发现菌龄、生长条件及培养基种类依然可能影响蛋白质量,如孵育时间<24 h的念珠菌、非CO2条件下培养的肺炎链球菌、形成了芽孢的厌氧梭菌等都出现过鉴定错误[9,13];操作者的技术能力[14]、对前处理方法的选择、基质液保存条件、涂布靶板的厚薄也会影响晶体形成的效果从而影响到鉴定准确性。直接涂抹法最为简单也最常使用,但厚薄不均及细胞破壁不理想都可能导致结晶不均匀,如黏液型铜绿假单胞菌、肺炎克雷伯菌、葡萄球菌、部分非发酵菌、放线菌等[15,16]。采用预处理法比如乙醇/甲酸萃取法,不但可灭活待测菌,得到的肽峰数目及质量均好于直接涂抹法且有很好的重复性,可有效提高鉴定能力[2,17]。三氟乙酸提取法、海沙/丙酮提取法、酸洗玻璃珠提取法等也有助于特殊待检菌蛋白的提取[18]。但目前对于丝状真菌、奴卡菌和分枝杆菌的蛋白提取方法仍需要改进[19,20]。因此使用质谱系统时,如果遇到少见菌、疑难菌、鉴定值<2.0、结果与菌落形态或镜下形态差别较大时,也应综合分析、调整预处理方法、重新鉴定,宜采用多种方法包括手工实验等加以确定,以免造成错误。同时要严格按照标准化操作程序进行培养、检测、质量管理和维护。且只有经过培训和考核后的工作人员才能进行操作。
而分子生物学鉴定技术与这两种鉴定系统相比,不受微生物生长环境和状态的影响,种属限制性小。对临床少见菌、苛养菌、以及固体培养基不生长病原菌的鉴定具有高度的灵敏度和准确性[3],能够更为准确的区分部分复合菌群,形态学及ITS测序仍是真菌鉴定的标准方法。虽然引物16sRNA和ITS的通用性良好,可作为优先检测基因,要得到更准确的鉴定应结合具体情况推荐选用其他功能性基因片段的特异性引物。
本研究证实了以MALDI Biotyper为代表的质谱鉴定系统能够快速、准确、高通量的鉴定临床大部分的分离菌,对混合标本、阳性血培养标本、分离菌耐药性和毒力因子的检测,也拓展了质谱的应用领域[1,2,21]。但是受限于数据库数量和质量以及它无法区分蛋白差异较小的病原菌,因此还不能完全取代传统的表型鉴定系统。临床工作中,可以将基质辅助激光解吸电离飞行时间质谱仪系统作为基本的鉴定手段,当出现鉴定结果不佳时,应联合应用形态学、表型鉴定以及分子生物学等方法做出最准确的结论。































