
探讨CYP19A1 rs2899470、GSTM1、GSTT1、GSTP1 rs1695的基因多态性和子宫内膜异位症(EM)的关系。
通过聚合酶链反应-限制性片段长度多态性和多重等位基因特异聚合酶链式反应方法,检测从2015年10月至2017年10月内蒙古医科大学附属医院收治262例EM患者和275名健康者的CYP19A1 rs2899470、GSTM1、GSTT1、GSTP1 rs1695的基因分型。
携带CYP19A1 rs2899470 TT基因型的个体比携带GG基因型患EM的风险更高(P<0.01),调整后的OR为2.33(95%CI 1.27~4.33);此外CYP19A1 rs2899470 TT基因型的个体将加重EM的病情(P<0.01),OR为2.27(95%CI 1.05~4.90);而GSTM1、GSTT1和GSTP1 rs1695的多态性与EM发病风险没有关系。
CYP19A1 rs2899470多态性与EM发病风险及病情风险均相关。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
子宫内膜异位症(EM)是引发痛经、干扰卵巢排卵、子宫内受精、胚胎着床等多环节的一种顽固性疾病[1,2],近年来其发病率呈明显上升趋势,40%的不孕患者与EM有关[3]。其发病原因不清楚。当特定基因位点出现多态性可引起人体对环境毒素物质代谢异常,引发EM发生[4]。细胞色素P450酶系(CYP19A1)和谷胱苷肽转硫酶系(GST)是体内Ⅰ相及Ⅱ相代谢酶的代表酶,参与环境中毒素物质在体内的代谢过程。EM的发病与CYP19A1和GST基因多态性有一定的关系[4,5],但是不同地区、种族的得到的结论并不一致。EM是一种雌激素依赖性疾病,与局部雌激素生成活跃密切相关,CYP19A1基因的编码产物——芳香化酶即是性腺内、外组织中雌激素合成的关键酶,也是细胞内重要的Ⅰ相代谢酶,在环境毒素物质的解毒和活化过程中起着重要的作用;目前,GST家族中研究较多的是GSTM1、GSTT1和GSTP1基因,其基因多态性可能会造成GST酶活性丧失或降低,导致个体对环境致病因素敏感性增高,引发或加重疾病。本研究探讨EM和CYP19A1、GSTM1、GSTT1和GSTP1基因多态性的关系探讨可能的影响机制,寻找EM易感位点,为EM的预防以及个体化治疗提供必要的实验支持。
选取2015年10月至2017年10月期间在内蒙古医科大学附属医院收治,经病理确诊为EM患者262例(试验组),同时期来本院体检的排除EM的育龄健康女性275名(对照组),所有研究对象在入组之前均签署知情同意书,研究协议符合内蒙古医科大学附属医院的伦理制度要求。试验组入组标准:经病理诊断为EM,月经周期规律、基础激素在正常范围,既往无卵巢手术史,3个月内无服用激素及其他药物病史。排除其他内分泌疾病,按照美国生殖医学会2012年EM病情分期标准,根据病变侵及单双侧卵巢、腹膜病变病灶的位置、大小和深浅,输卵管、卵巢粘连的程度和范围,以及道格拉斯窝封闭的严重程度进行评分分为Ⅰ~Ⅳ期。对照组入组标准:经询问病史、妇科检查及B超检查排除了EM及其他妇科疾患等疾病,并且与试验组无任何血缘关系,以及无直系亲属患有EM的体检健康者。
(1)主要试剂耗材及仪器:血液DNA提取试剂盒(货号DP318,北京天根生化公司)、Plantinum PCR Master Mix(货号KT204,北京天根生化公司)、D2000 DNA marker(货号MD114,北京天根生化公司)、D15000 DNA marker(货号BM012-01:EarthOx)、限制性内切酶FastDigest MspⅠ(货号FD0544,立陶宛Fermentas公司)、限制性内切酶RasⅠ(货号R6371,Promega公司)、NanoDrop2000超微量分光光度计、凝胶成像仪。引物由上海生工生物工程有限公司代为合成,纯度为PAGE级。检测的基因,检测的位点,引物序列及扩增片段大小见表1。(2)方法:采集入组者血液2 ml,并将其保存在-20 ℃冰箱。提取血液DNA后,采用多重等位基因特异聚合酶链式反应(MAS-PCR)方法检测GSTM1和GSTT1基因多态性。使用相应引物对不同基因片段进行PCR扩增,扩增产物通过浓度为2%琼脂糖凝胶进行电泳检测,并判断是否符合预期片段大小。对经琼脂糖电泳检测合格的GSTM1、GSTT1的PCR产物,进行MspⅠ酶切,酶切程序为:37 ℃,60 min;70 ℃,5 min;4 ℃,30 min。采用限制性片段长度多态性聚合酶链反应(RFLP-PCR)方法检测CYP19A1 rs2899470和GSTP1 rs1695基因多态性。对经琼脂糖电泳检测合格的CYP19A1基因PCR产物,进行RasⅠ酶切,酶切程序为:37 ℃,60 min;70 ℃,5 min;4 ℃,30 min。在120 V电压下电泳30 min后在紫外凝胶成像中成像,并记录结果。

引物及其相关信息
引物及其相关信息
| 检测基因 | 检测位点 | 序列(5 ′ ➝3 ′) | 扩增大小(bp) |
|---|---|---|---|
| CYP19A1 | rs2899470 | F:CAGGTAGCTCTGTGG AATAGCC | 340 |
| R:GCAGAAAATTAATGA GGACCACAG | |||
| GSTM1 | present/ Null | F:GAACTCCCTGAAAAGC TAAT | 219 |
| R:GTTGGGCTCAAATATA CGGTGG | |||
| GSTT1 | present/ Null | F:TTCCTTACTGGTCCTCAC ATCTC | 120 |
| R:TCACCGGATCATGGCCA GCA | |||
| GSTP1 | rs1695 | F:GTAGTTTGCCCAAGGTC AAG | 440 |
| R:GAAGAGCCAAGGACAG GTAC | |||
| β-球蛋白基因 | F:CAACTTCATCCACGTTC ACC | 350 | |
| R:GAAGAGCCAAGGACAG GTAC |
根据哈迪-温伯格平衡检验(HWE)检测群体调查资料的可靠性;应用χ2检验分析病例组和对照组各基因多态位点的基因型和等位基因频率差异有无统计学意义,用比值比(OR)及其95%的可信区间表示关联的强度。采用多元逻辑回归分析评估CYP19A1 rs2899470、GSTM1、GSTT1和GSTP1 rs1695和EM的发病风险。一般资料的比较采用SPSS 19.0软件,进行t检验,以P<0.05为差异有统计学意义。CYP19A1 rs2899470及GSTP1 rs1695为多位点分析,进一步做Bonferroni校正,α=0.012 5。
两组对象的人口统计学和临床变量见表2所示。两组年龄(t=1.38,P=0.08)、初潮年龄(t=0.60,P=0.28)、妊娠(t=0.56,P=0.29)和奇偶校验(t=0.74,P=0.23)均差异无统计学意义。
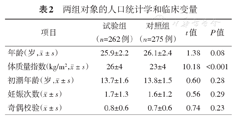
两组对象的人口统计学和临床变量
两组对象的人口统计学和临床变量
| 项目 | 试验组(n=262例) | 对照组(n=275例) | t值 | P值 |
|---|---|---|---|---|
年龄(岁, ± s) ± s) | 25.9±2.2 | 26.1±2.4 | 1.38 | 0.08 |
体质量指数(kg/m2, ± s) ± s) | 26±4 | 23±4 | 10.18 | <0.001 |
初潮年龄(岁, ± s) ± s) | 13.7±1.6 | 13.8±1.5 | 0.60 | 0.28 |
妊娠次数( ± s) ± s) | 1.7±1.3 | 1.6±1.2 | 0.56 | 0.29 |
奇偶校验( ± s) ± s) | 0.8±0.6 | 0.7±0.6 | 0.74 | 0.23 |
提取血液基因组DNA经电泳检测显示基因组DNA条带完整,没有降解。以其为模板,对CYP19A1特异引物进行PCR扩增,片段大小为340 bp,PCR产物通过限制性内切酶RasⅠ进行酶切,CYP19A1基因片段扩增大小为340 bp,等位基因T的纯合子基因型TT(野生型),产生的片段为340 bp,等位基因C的纯合子基因型为GG,产生的片段为200和140 bp,T和C的杂合子基因型TG(杂合型),产生片段为340、200和140 bp,片段大小与预期一致。同理对GSTP1特异引物进行PCR扩增,PCR产物通过限制性内切酶MspⅠ进行酶切,GSTP1基因片段扩增大小为440 bp,野生基因型为AA,产生的片段为440 bp,突变基因型为GG,产生的片段为227和213 bp,因为胶的分辨率的问题,227和213 bp的大小在2%的琼脂糖上显示1条带,杂合基因型为AG,产生片段为340、227和213 bp,片段大小与预期一致。
以上述提取的DNA为模板,使用GSTM1特异引物、GSTT1特异引物和β-球蛋白基因内参引物,通过MAS-PCR检测GSTM1及GSTT1基因多态性,结果β-球蛋白基因片段扩增大小为350 bp,与预期相符,GSTM1的非缺失型基因型扩增片段为219 bp,缺失型没有扩增产物;GSTT1的非缺失型扩增片段为120 bp,缺失型没有扩增产物,均与预期一致。
CYP19A1 rs2899470,GSTM1,GSTT1和GSTP1 rs1695的基因多态性和EM发生风险见表3,CYP19A1 rs2899470基因多态性的TT基因型的个体比携带GG基因型的个体患EM的风险明显增高(P<0.01,经Bonferroni校正,以α=0.012 5为检测标准),调整后OR为2.33(95%CI 1.27~4.33)。携带TG+TT基因型与GG基因型的个体相比患EM的风险P值为0.03(OR=1.48,95%CI 1.03~2.13),但经Bonferroni校正,显示携带这两种基因型的个体相比患EM的风险差异无统计学意义。此外,没有发现GSTM1、GSTT1和GSTP1 rs1695基因多态性与EM风险有任何关联。
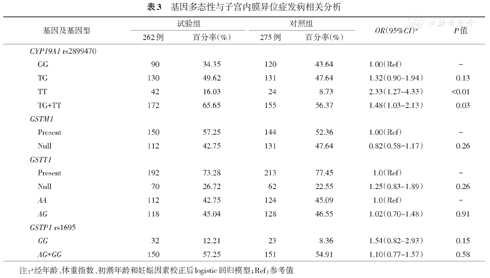
基因多态性与子宫内膜异位症发病相关分析
基因多态性与子宫内膜异位症发病相关分析
| 基因及基因型 | 试验组 | 对照组 | OR(95%CI)a | P值 | |||
|---|---|---|---|---|---|---|---|
| 262例 | 百分率(%) | 275例 | 百分率(%) | ||||
| CYP19A1 rs2899470 | |||||||
| GG | 90 | 34.35 | 120 | 43.64 | 1.00(Ref) | - | |
| TG | 130 | 49.62 | 131 | 47.64 | 1.32(0.90~1.94) | 0.13 | |
| TT | 42 | 16.03 | 24 | 8.73 | 2.33(1.27~4.33) | <0.01 | |
| TG+TT | 172 | 65.65 | 155 | 56.37 | 1.48(1.03~2.13) | 0.03 | |
| GSTM1 | |||||||
| Present | 150 | 57.25 | 144 | 52.36 | 1.00(Ref) | - | |
| Null | 112 | 42.75 | 131 | 47.64 | 0.82(0.58-1.17) | 0.26 | |
| GSTT1 | |||||||
| Present | 192 | 73.28 | 213 | 77.45 | 1.0(Ref) | - | |
| Null | 70 | 26.72 | 62 | 22.55 | 1.25(0.83~1.89) | 0.26 | |
| AA | 112 | 42.75 | 124 | 45.09 | 1.0(Ref) | - | |
| AG | 118 | 45.04 | 128 | 46.55 | 1.02(0.70~1.48) | 0.91 | |
| GSTP1 rs1695 | |||||||
| GG | 32 | 12.21 | 23 | 8.36 | 1.54(0.82~2.93) | 0.15 | |
| AG+GG | 150 | 57.25 | 151 | 54.91 | 1.10(0.77~1.57) | 0.58 | |
注:a经年龄、体重指数、初潮年龄和妊娠因素校正后logistic回归模型;Ref:参考值
CYP19A1 rs2899470、GSTM1、GSTT1和GSTP1 rs1695的基因多态性和EM病情风险见表4,经Bonferroni校正显示CYP19A1 rs2899470基因多态性的TT基因型的个体比携带GG基因型的个体患EM的风险明显增高(χ2=6.69,P<0.01),OR为2.27(95%CI 1.05~4.90)。没有发现GSTM1、GSTT1和GSTP1 rs1695基因多态性与EM的病情有任何关联。
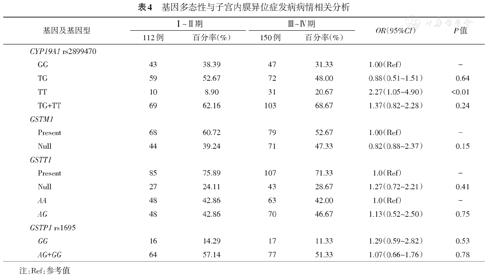
基因多态性与子宫内膜异位症发病病情相关分析
基因多态性与子宫内膜异位症发病病情相关分析
| 基因及基因型 | Ⅰ~Ⅱ期 | Ⅲ~Ⅳ期 | OR(95%CI) | P值 | |||
|---|---|---|---|---|---|---|---|
| 112例 | 百分率(%) | 150例 | 百分率(%) | ||||
| CYP19A1 rs2899470 | |||||||
| GG | 43 | 38.39 | 47 | 31.33 | 1.00(Ref) | - | |
| TG | 59 | 52.67 | 72 | 48.00 | 0.88(0.51~1.51) | 0.64 | |
| TT | 10 | 8.90 | 31 | 20.67 | 2.27(1.05~4.90) | <0.01 | |
| TG+TT | 69 | 62.16 | 103 | 68.67 | 1.37(0.82~2.28) | 0.24 | |
| GSTM1 | |||||||
| Present | 68 | 60.72 | 79 | 52.67 | 1.00(Ref) | - | |
| Null | 44 | 39.24 | 71 | 47.33 | 0.82(0.88~2.37) | 0.15 | |
| GSTT1 | |||||||
| Present | 85 | 75.89 | 107 | 71.33 | 1.0(Ref) | - | |
| Null | 27 | 24.11 | 43 | 28.67 | 1.27(0.72~2.21) | 0.41 | |
| AA | 48 | 42.86 | 63 | 42.00 | 1.0(Ref) | - | |
| AG | 48 | 42.86 | 70 | 46.67 | 1.13(0.52~2.50) | 0.75 | |
| GSTP1 rs1695 | |||||||
| GG | 16 | 14.29 | 17 | 11.33 | 1.29(0.59~2.82) | 0.53 | |
| AG+GG | 64 | 57.14 | 77 | 51.33 | 1.07(0.66~1.76) | 0.78 | |
注:Ref:参考值
EM成因复杂,既有环境因素的作用,又有遗传变异。EM的发生与特定基因位点出现多态性,引起人体对环境毒素物质代谢异常有关。CYP19A1是芳香化酶P450 19A1的编码基因,也是有代表性的Ⅰ相代谢酶,在雌激素合成过程中,芳香化酶(Cytochrome,P450 family)是关键限速酶之一,把C19类固醇转化成雌激素的合成酶。在正常子宫内膜,芳香化酶呈低表达或不表达,而在异位内膜及在位内膜腺体和间质细胞胞质内呈高表达状态,且异位内膜表达高于在位内膜,雌激素合成过程的芳香化酶在EM呈活跃状态,可能参与疾病的发生和发展[4]。GST是一种普遍存在各种物种和细胞内的Ⅱ相代谢酶,并参与多种细胞功能的蛋白,作为与炎症相关的蛋白,内膜异位的组织中其表达水平比正常组织要低得多[5]。目前已经证明基因的多态性与各种各样的疾病有关系,基因多态性的产生根源是基因水平上的变异,这种变异将通过各种不同的机制影响细胞内的生化过程。CYP 19A1和GST基因的多态性也可能对EM的病发及疾病的进程产生影响。
研究表明,rs2899470位于CYP19A1基因内含子部分与循环雌激素、血清中雌二醇(E2)和睾酮(T)比值及E2水平均有相关性[6]。尽管以前有研究证明CYP19A1的表达与EM发病的风险之间的关联,然而不同地区、人种的研究未能取得一致的结果[7]。本研究证明,经Bonferroni校正CYP19A1 rs2899470基因多态性的TT基因型的个体比携带GG基因型的个体患EM的风险明显增高(P<0.01),调整后OR为2.33。但是,携带TG+TT基因型与GG基因型的个体相比患EM的风险P=0.03,经Bonferroni校正显示携带TG+TT基因型与GG基因型的个体相比患EM的风险并没有明显增高。另外,在EM病情分级的试验组中也发现CYP19A1基因rs2899470位点的TT基因型将影响EM病情的发展并加重病情,OR为2.27;这与CYP19A1基因多态性引起雌激素高水平表达,进而引起EM的发病率增高有一定的相似性[8]。本研究证明,CYP19A1基因的rs2899470位点和EM的风险之间以及该位点与EM病情发展存在明显的相关性。
GSTM1、GSTT1和GSTP1基因多态性会造成GST酶活性丧失或降低,可能导致个体对环境致病因素敏感性增高,但是不同研究结论并不统一[9]。一项回顾性分析认为,GSTM1、GSTT1和GSTM1/GSTT1基因多态性和EM易感性之间的可能关联。研究中发现,GSTM1和GSTT1的null缺失与EM的风险,分别为OR=1.56和OR=1.31,GSTM1/GSTT1 (OR=1.68,95%CI:1.29~2.17,P<0.01),显示GST基因多态性增加了EM的风险[10]。伊朗学者研究发现,GSTM1的null缺失和GSTP1基因的313 A/G与EM发病风险有关[11]。在巴西的戈亚斯州人中,GSTT1和GSTM1的present/pull和TP53基因72号氨基酸与EM关联分析发现,GSTT1和GSTM1的present/pull与EM发病风险是不相关的,而与TP53基因72 Arg/Arg联合作用,与EM的风险是相关的[12]。亦有研究发现,GSTP1基因的-313A/G,外显子5的多态性与EM的发病风险没有关系,而GSTP1基因的Val/Ile位点与EM的发病风险有关系[13]。本结果显示,GSTM1、GSTT1和GSTP1基因GSTP1 rs1695位点的多态性与EM发病风险以及病情发展的风险没有任何关联。本结果与其他结论不同的原因可能是由于地区、人群不同,并且研究例数偏少,而EM是一种多因素致病的疾病,因此对于CYP19A1和GSTs基因多态性与EM的发病机制的研究,仍有待扩大样本进行进一步探索。
探讨EM患者多种基因结构、功能及表达的改变,有助于EM的有效防预和早期治疗。本研究对EM不孕患者从源头上明确诊断,筛查异位症易感人群,为EM早期诊断找到可靠的诊断指标。
所有作者均声明不存在利益冲突



























