
分析1株碳青霉烯耐药肺炎克雷伯菌(CRKP)的耐药及毒力特征。
将浙江大学医学院附属第二医院粪便中分离到的1株CRKP命名为肺炎克雷伯菌C35,采用微量肉汤稀释法测定抗菌药物的最低抑菌浓度;全基因组测序和基因组分析确定菌株所携带耐药基因和毒力基因;核心基因组单核苷酸多态性(SNP)分型分析CRKP菌株之间的同源性关系;采用接合试验评估耐药基因的转移能力和效率;采用大蜡螟毒力实验测定菌株的毒力表型。
肺炎克雷伯菌C35对大多数受试药物耐药,尤其碳青霉烯类、舒巴坦和多黏菌素。SNP分型显示肺炎克雷伯菌C35与多株分离自本院不同病房的CRKP菌株具有很高的同源性。该菌属于ST11型,携带包括blaKPC-2、blaCTX-M-199、mcr-1和tet(A)变异体等在内的13种耐药基因。blaKPC-2基因位于>69 800 bp的IncFⅡ型质粒上,blaCTX-M-199和mcr-1基因共同位于>64 800 bp的IncI2型质粒上,tet(A)变异体位于83 628 bp的不可分型质粒上,3种质粒均为可接合性质粒。肺炎克雷伯菌C35还携带rmpA和rmpA2毒力基因以及气杆菌素(aerobactin)相关基因iucABCD,为典型的碳青霉烯耐药高毒力肺炎克雷伯菌(CR-hvKP)。该菌在大蜡螟感染模型中也显示了较强的毒力表型,感染肺炎克雷伯菌C35菌株48 h后大蜡螟幼虫存活率仅为16.7%,明显低于无毒力对照菌株的80.0%。
本研究在1株肠道定植CR-hvKP中检测到多个可接合性耐药质粒,包括同时携带blaCTX-M-199和mcr-1基因的IncI2质粒,需引起警惕,并对此类菌株进行主动监测。

碳青霉烯类抗生素是临床上治疗多重耐药革兰阴性菌感染的重要药物,碳青霉烯耐药菌株的出现和传播给全球公共卫生带来严峻挑战[1],其中碳青霉烯耐药肺炎克雷伯菌(CRKP)的耐药情况尤为严重。CHINET细菌耐药监测网历年来的数据显示,三级医院分离的肺炎克雷伯菌对碳青霉烯的耐药率从2005年的3%左右急剧上升至2018年的接近30%[2]。更令人担心的是,近年来高毒力肺炎克雷伯菌(hvKP)的报道不断增多,其所致感染往往后果严重甚至死亡[3]。多黏菌素是目前治疗CRKP感染为数不多的选择之一,其耐药性因此备受关注。2016年发现于中国的质粒介导多黏菌素耐药基因mcr-1因其水平传播能力而被格外重视[4]。2017年本课题组在3株分离自粪便标本的大肠埃希菌中发现1种新型的可接合IncI2质粒,除了mcr-1基因,该质粒还同时携带1个新基因,即blaCTX-M-199,该基因由blaCTX-M-64在109位(丙氨酸被替换为苏氨酸,即A109T)和130位(丝氨酸被替换为苏氨酸,即S130T)发生突变而形成,实验证实S130T突变导致了CTX-M-199对舒巴坦和他唑巴坦两种β内酰胺酶抑制剂耐药[5]。2018年本课题组在对住院患者粪便标本进行CRKP常规筛查时发现1株多黏菌素耐药的CRKP,该菌株blaCTX-M-199和mcr-1基因均为阳性,提示IncI2质粒已经传播到CRKP中,本研究对该菌株的耐药及毒力特征进行分析。
1. 菌株来源:患者女,93岁,2018年2月因肥厚性梗阻性心肌病入住本院心血管内科,后因病情加重转入脑科重症监护病房。考虑到患者年龄大且病情危重,入院后即预防性给予头孢哌酮/舒巴坦,连续使用47 d后,于患者粪便中筛查到1株碳青霉烯耐药的肠杆菌目细菌(CRE),经基质辅助激光解吸/电离飞行时间质谱仪(MALDI-TOF MS,德国布鲁克公司)鉴定为肺炎克雷伯菌,Vitek 2 Compact全自动细菌鉴定和药敏分析仪(法国生物梅里埃公司)显示该菌对碳青霉烯类抗生素耐药,本研究将其命名为肺炎克雷伯菌C35。该菌菌落光滑、湿润,非黏液状,使用接种环挑取时亦未见拉丝现象。
2. 药物敏感性试验:采用微量肉汤稀释法测定抗菌药物对细菌的最小抑菌浓度(MIC),受试药物包括亚胺培南、美罗培南、厄他培南、头孢噻肟、头孢他啶、头孢吡肟、哌拉西林/他唑巴坦、头孢哌酮/舒巴坦、头孢他啶/阿维巴坦、氨曲南、头孢美唑、阿米卡星、环丙沙星、四环素、替加环素和黏菌素,药敏板为本实验室委托珠海迪尔生物工程有限公司定制。操作步骤如下:挑取过夜培养的新鲜菌落调成0.5麦氏单位的菌悬液,吸取50 μl菌液至10 ml Mueller-Hinton肉汤培养基中混匀,吸取100 μl稀释后的菌液至96孔药敏板中,37 ℃孵箱过夜培养,根据药敏板中细菌生长与否判定抗菌药物的MIC。此外,氯霉素MIC值的测定采用E-test方法(法国梅里埃公司),磷霉素MIC值的测定采用琼脂稀释法(琼脂中含终浓度为25 μg/ml的葡萄糖-6-磷酸),操作方法和结果判断参照美国临床和实验室标准委员会(CLSI)标准[6, 7]。质控菌株为大肠埃希菌ATCC25922。头孢哌酮/舒巴坦的判断标准参照头孢哌酮,替加环素的结果判断参照欧洲药敏试验委员会(EUCAST)标准。
3. 全基因组测序(WGS):提取肺炎克雷伯菌C35基因组DNA,经DNA文库构建后在Illumina novaseq6000平台上进行WGS,产生的短读长(reads)采用SPAdes软件(v3.11.1)进行拼接。组装后的全基因组序列在基因组流行病学中心(CGE)网站(http://www.genomicepidemiology.org/)进行多位点序列分型(MLST)、质粒Inc分型(PlasmidFinder 2.1)和耐药基因识别(ResFinder 4.1)等分析,使用Kleborate v0.3.0 28程序搜索肺炎克雷伯菌毒力相关基因(https://github.com/katholt/Kleborate/)。使用RAST服务器(https://rast.nmpdr.org/)对DNA序列进行基因注释。
4. 菌株同源性分析:为明确肺炎克雷伯菌C35与同一时期分离自本院的CRKP之间的同源性关系,将C35与本实验室于2018年2至5月期间分离且做过WGS的CRKP进行系统进化分析。采用Harvest suite软件提取菌株核心基因组,根据核心基因组之间的单核苷酸多态性(SNP)使用Parsnp软件构建系统发育树,采用iTOL软件对生成的进化树进行编辑和可视化处理。
5. 接合试验:以利福平耐药的大肠埃希菌EC600作为受体菌,采用滤膜法进行接合试验。供体菌和受体菌分别接种于Luria-Bertani肉汤培养基,37 ℃摇床培养至对数生长期时,分别吸取20 μl菌液至1 cm2大小的滤纸上,37 ℃孵箱静置过夜。将滤膜上的混合培养物用Luria-Bertani肉汤培养基洗脱后,吸取一定量菌液均匀涂布于筛选平板,37 ℃孵箱过夜培养。不同耐药基因的接合试验选用不同的筛选平板。携带mcr-1基因的转移接合子采用500 μg/ml利福平和1 μg/ml黏菌素平板进行筛选,blaKPC-2基因的筛选采用500 μg/ml利福平和0.5 μg/ml美罗培南平板,tet(A)基因的筛选采用500 μg/ml利福平和20 μg/ml四环素平板。用MALDI-TOF MS对筛选平板上的菌落进行鉴定,结果为大肠埃希菌者转划筛选平板,PCR扩增和测序验证是否存在相应耐药基因。以转移接合子与受体菌数量的比值计算接合效率。
6. 毒力检测试验:建立大蜡螟感染模型检测肺炎克雷伯菌的毒力。根据文献报道[8],随机选取体长2~3 cm、体重250~300 mg、活动度好的大蜡螟幼虫作为实验对象。以本实验室先前报道的已知hvKP(K. pneumoniae 4)作为阳性对照株,以不含毒力基因的ST11型肺炎克雷伯菌FJ8作为阴性对照株[3]。简要操作步骤如下:将肺炎克雷伯菌接种Luria-Bertani肉汤培养基,培养至对数生长期,用磷酸盐缓冲液调整至1.0×108 CFU/ml,用微量注射器抽取10 μl菌液,经右后足注射入大蜡螟幼虫体腔,每株试验菌株测试8条随机分配的幼虫。为了保证肺炎克雷伯菌有最适生长温度,将幼虫置于无菌培养皿中放置37 ℃孵箱中,分别记录18、24、42、48 h时大蜡螟幼虫的存活情况,重复试验3次,绘制大蜡螟生存曲线。
1. 药物敏感性试验结果:肺炎克雷伯菌C35对包括碳青霉烯类在内的所有β内酰胺类、四环素、环丙沙星、氯霉素和磷霉素均呈高水平耐药,黏菌素的MIC为4 μg/ml,达到耐药水平。该菌株仅对阿米卡星、替加环素和头孢他啶/阿维巴坦敏感(表1)。
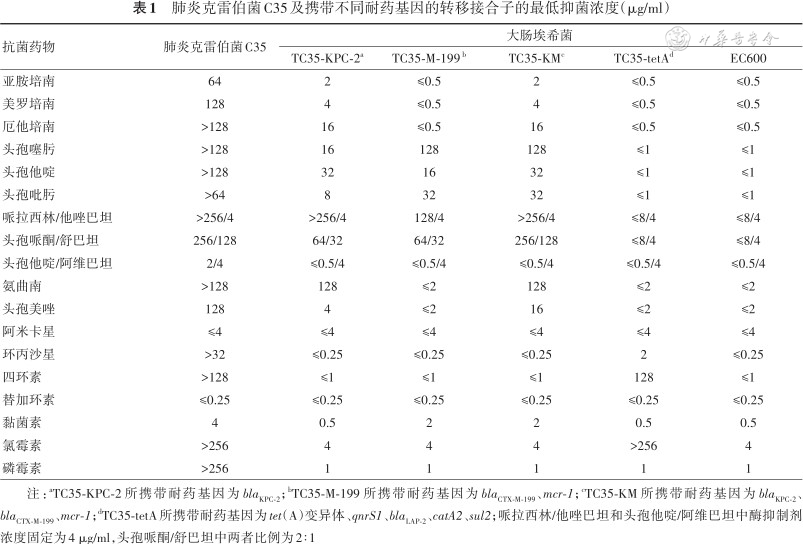
肺炎克雷伯菌C35及携带不同耐药基因的转移接合子的最低抑菌浓度(μg/ml)
肺炎克雷伯菌C35及携带不同耐药基因的转移接合子的最低抑菌浓度(μg/ml)
| 抗菌药物 | 肺炎克雷伯菌C35 | 大肠埃希菌 | ||||
|---|---|---|---|---|---|---|
| TC35-KPC-2a | TC35-M-199b | TC35-KMc | TC35-tetAd | EC600 | ||
| 亚胺培南 | 64 | 2 | ≤0.5 | 2 | ≤0.5 | ≤0.5 |
| 美罗培南 | 128 | 4 | ≤0.5 | 4 | ≤0.5 | ≤0.5 |
| 厄他培南 | >128 | 16 | ≤0.5 | 16 | ≤0.5 | ≤0.5 |
| 头孢噻肟 | >128 | 16 | 128 | 128 | ≤1 | ≤1 |
| 头孢他啶 | >128 | 32 | 16 | 32 | ≤1 | ≤1 |
| 头孢吡肟 | >64 | 8 | 32 | 32 | ≤1 | ≤1 |
| 哌拉西林/他唑巴坦 | >256/4 | >256/4 | 128/4 | >256/4 | ≤8/4 | ≤8/4 |
| 头孢哌酮/舒巴坦 | 256/128 | 64/32 | 64/32 | 256/128 | ≤8/4 | ≤8/4 |
| 头孢他啶/阿维巴坦 | 2/4 | ≤0.5/4 | ≤0.5/4 | ≤0.5/4 | ≤0.5/4 | ≤0.5/4 |
| 氨曲南 | >128 | 128 | ≤2 | 128 | ≤2 | ≤2 |
| 头孢美唑 | 128 | 4 | ≤2 | 16 | ≤2 | ≤2 |
| 阿米卡星 | ≤4 | ≤4 | ≤4 | ≤4 | ≤4 | ≤4 |
| 环丙沙星 | >32 | ≤0.25 | ≤0.25 | ≤0.25 | 2 | ≤0.25 |
| 四环素 | >128 | ≤1 | ≤1 | ≤1 | 128 | ≤1 |
| 替加环素 | ≤0.25 | ≤0.25 | ≤0.25 | ≤0.25 | ≤0.25 | ≤0.25 |
| 黏菌素 | 4 | 0.5 | 2 | 2 | 0.5 | 0.5 |
| 氯霉素 | >256 | 4 | 4 | 4 | >256 | 4 |
| 磷霉素 | >256 | 1 | 1 | 1 | 1 | 1 |
注:aTC35-KPC-2所携带耐药基因为blaKPC-2;bTC35-M-199 所携带耐药基因为blaCTX-M-199、mcr-1;cTC35-KM所携带耐药基因为blaKPC-2、blaCTX-M-199、mcr-1;dTC35-tetA所携带耐药基因为tet(A)变异体、qnrS1、blaLAP-2、catA2、sul2;哌拉西林/他唑巴坦和头孢他啶/阿维巴坦中酶抑制剂浓度固定为4 μg/ml,头孢哌酮/舒巴坦中两者比例为2∶1
2. WGS和生物信息学分析:肺炎克雷伯菌C35经二代测序后获得约5.8 Mbp的基因组序列。MLST显示该菌株为ST11型,属于国内分离CRKP中常见的型别[9]。耐药基因分析发现肺炎克雷伯菌C35携带13种基因,可介导β内酰胺类(blaKPC-2、blaCTX-M-199、blaSHV-11和blaLAP-2),多黏菌素类(mcr-1),磷霉素(fosA),喹诺酮类(qnrS1),四环素类[tet(A)],氯霉素类(catA2),甲氧苄啶(dfrA14),磺胺类(sul1、sul2)和氨基糖苷类(aadA2)等9个不同种类的抗菌药物耐药。其中以blaKPC-2、blaCTX-M-199和mcr-1基因最受关注。此外,经过与野生型tet(A)基因(GenBank序列号X00006)进行序列比对发现,本研究中的tet(A)基因存在7处点突变(I5R、V55M、I75V、T84A、S201A、F202S和V203F),是一种常见的可导致替加环素MIC上升的变异体,有学者将其命名为type 1变异体[10]。
序列分析发现IncI2型质粒片段(pC35-M-199,64 847 bp)同时携带blaCTX-M-199和mcr-1基因,前者位于ISEcp1介导的转座单元中,后者上游未见可移动元件,如常见的ISApl1[11]。对比发现该质粒序列与本实验室前期发现的pZE36质粒序列长度相当,且相似性为99.9%(图1A)[5],提示相同质粒在大肠埃希菌和肺炎克雷伯菌之间的传播。blaKPC-2基因位于国内常见的ISKpn27-blaKPC-2-ISKpn6核心结构中[9],该结构所在质粒片段(pC35-KPC-2,69 877 bp)为IncFⅡ型,其序列与分离自江苏省的肺炎克雷伯菌质粒pA1750-KPC相似性最高,主要差别是缺失了1个TnAs1所携带的blaSHV-12基因(图1B)。tet(A)变异体、qnrS1、blaLAP-2、catA2和sul2基因共同位于1个83 628 bp的不可分型质粒上(pC35-tetA),其序列与分离自杭州的肺炎克雷伯菌质粒pKP58-3高度相似,仅缺失1个TnAs3所携带的drfA14基因(图1C)。
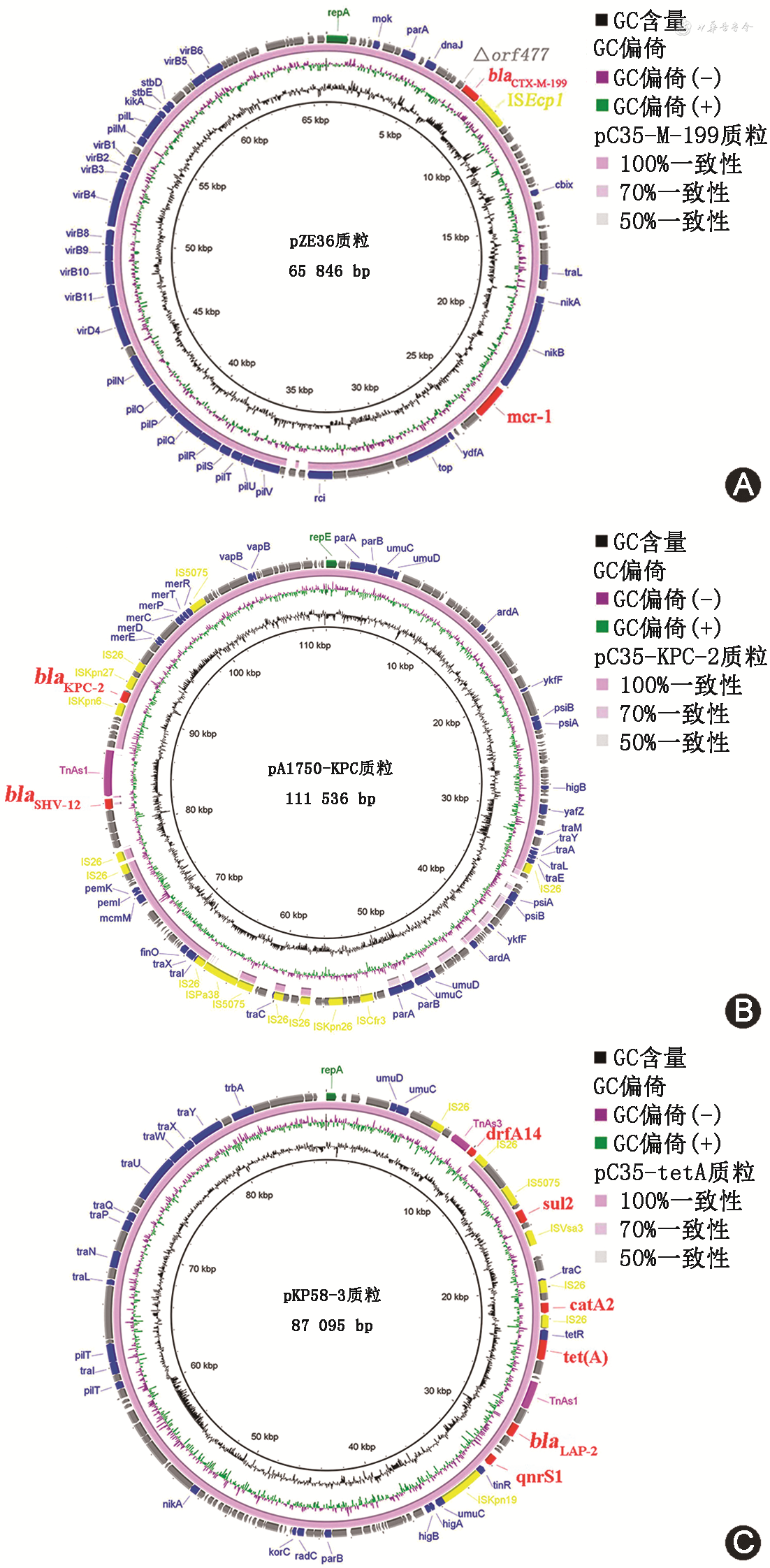

3. 菌株同源性分析结果:肺炎克雷伯菌C35与多株分离自不同病房、不同标本的产KPC-2酶CRKP具有很高的同源性,尤其与分离自同病房(脑科重症监护室)的0207和0221菌株、分离自胸外科病房的0426-1和0426-2菌株以及分离自肝胆胰外科病房的0505-1菌株仅有约50个SNP的差异,提示C35菌株可能是本院CRKP的优势克隆株。
4. 接合试验:编码blaCTX-M-199和mcr-1基因的pC35-M-199质粒、编码blaKPC-2基因的pC35-KPC-2质粒以及编码tet(A)变异体、qnrS1、blaLAP-2、catA2和sul2基因的pC35-tetA质粒均可通过接合试验传递给大肠埃希菌EC600,接合频率分别为2.5×10-4/受体菌、3.0×10-8/受体菌和5.0×10-5/受体菌,对应的大肠埃希菌转移接合子分别命名为TC35-M-199、TC35-KPC-2和TC35-tetA。药敏试验结果显示,大肠埃希菌TC35-KPC-2对碳青霉烯类、头孢菌素类和氨曲南等抗生素耐药,但对头霉素类和头孢他啶/阿维巴坦敏感。大肠埃希菌TC35-M-199对黏菌素的MIC值为2 μg/ml,虽处于敏感水平,但与接合前MIC值(0.5 μg/ml)相比,明显上升;对头孢菌素类耐药,但对氨曲南敏感;对哌拉西林/他唑巴坦和头孢哌酮/舒巴坦耐药,但对头孢他啶/阿维巴坦敏感,为典型的产CTX-M-199菌株的耐药谱。因为窄谱β内酰胺酶LAP-2不具备水解头孢菌素的能力,大肠埃希菌TC35-tetA仅表现出对四环素[tet(A)变异体]、环丙沙星(qnrS1)和氯霉素(catA2)耐药,但对替加环素敏感。本研究还筛选到1株同时携带pTC35-M-199和pTC35-KPC-2质粒的转移接合子(大肠埃希菌TC35-KM),除了具有两种质粒的耐药表型外,该菌株的头孢哌酮/舒巴坦的MIC值达到256/128 μg/ml,远高于携带单一质粒的菌株(64/32 μg/ml)(表1)。
5. 毒力基因及毒力表型检测:毒力基因分析发现肺炎克雷伯菌C35携带rmpA和rmpA2毒力基因以及气杆菌素生物合成相关基因iucABCD,前两者为荚膜多糖的调节基因,后者为铁载体相关因子。荚膜和气杆菌素的过量产生与肺炎克雷伯菌的高致病性表型密切相关,被认为是hvKP的特异性毒力因子[12],因此肺炎克雷伯菌C35属于CR-hvKP。毒力试验显示,感染hvKP阳性对照菌株(接种量1×106 CFU)18 h后,大蜡螟幼虫全部死亡;感染肺炎克雷伯菌C35 18 h后,大蜡螟幼虫存活率仅为25.0%(6/24),48 h后进一步下降到16.7%(4/24),低于无毒力的对照菌株80.0%的存活率,表现出了较强毒力。
KPC-2型碳青霉烯酶在世界范围内传播迅速[13, 14],2008年NDM-1型金属β内酰胺酶的出现进一步推高了CRKP的发生率[15]。2005年至今,国内三级医院分离的肺炎克雷伯菌对碳青霉烯的耐药率上升了近10倍[2],而高毒力菌株的出现使CRKP的防控更加严峻。本研究从1株国内常见的ST11型hvKP中检测到了多个重要的耐药基因,即blaKPC-2、blaCTX-M-199、mcr-1和tet(A)变异体,虽然这些基因所在可接合性质粒均为国内常见,但同时出现在1株hvKP中却十分罕见,尤其携带blaCTX-M-199和mcr-1基因的IncI2质粒发现于肺炎克雷伯菌中。另外,虽然肺炎克雷伯菌C35所携带rmpA和rmpA2基因以及气杆菌素相关基因在WGS测序结果中位于不同的DNA片段中,但有研究表明它们常位于同一个质粒上,且具有水平转移的能力[3, 12],提示该菌株具有很强的获得外源性基因的能力,需引起高度重视。系统发育分析表明肺炎克雷伯菌C35与多株同一时期分离自不同病房的CRKP具有很高的同源性。因此可以推测,C35作为本院CRKP的优势克隆,在抗生素压力及其他耐药菌株存在的情况下,通过水平转移获得此前仅存在于大肠埃希菌中的编码blaCTX-M-199和mcr-1基因的IncI2质粒以及常见于肺炎克雷伯菌中的编码tet(A)变异体的质粒和毒力质粒。C35菌株分离自本院住院患者粪便标本的CRE筛查,当时并未关注该患者粪便标本中是否存在碳青霉烯敏感、其他抗菌药物(如头孢哌酮/舒巴坦和四环素)耐药的菌株,因此缺乏同一患者体内不同菌株之间耐药基因转移的直接证据。
作为目前治疗CRKP感染的最后一道防线,多黏菌素耐药性一直是人们关注的焦点。肺炎克雷伯菌对黏菌素耐药主要是由于脂多糖修饰相关基因(即双组分系统)的突变,携带mcr-1基因的菌株比较少见[16]。本课题组此前的一项调查显示,国内健康成人粪便分离的肺炎克雷伯菌mcr-1基因阳性率仅为0.11%(8/7401),基因所在质粒的类型包括IncHI2、IncX4、IncHI2/IncN和IncFIB[17],未发现IncI2型质粒,本研究表明该质粒的跨种属传播已经发生。肺炎克雷伯菌的泛耐药率远高于大肠埃希菌,因此mcr-1基因在CRKP中将产生更大危害。虽然舒巴坦和他唑巴坦也不能很好地抑制KPC-2酶,但从转移接合子的药敏结果可以看出,KPC-2和CTX-M-199同时存在对头孢哌酮/舒巴坦的耐药性表现出了叠加效果,所幸这两种β内酰胺酶均可被阿维巴坦所抑制。与肺炎克雷伯菌C35相似,携带tet(A) type 1变异体的菌株也可表现为对替加环素敏感[18],然而有研究表明,与野生型tet(A)基因相比,该变异体可导致替加环素MIC值上升[19],在此变异体基础上诱导产生的某些突变(如G300E、S251A、I235F和I248L)则可导致MIC值进一步增加[20]。可以预见不久的将来,在抗生素的选择压力下,发生替加环素耐药突变的tet(A)变异体将会被筛选出来。
肠道定植CRE是临床感染重要的危险因素,在一项1 806例CRE定植患者的荟萃分析中,系统性CRE感染的总风险达16.5%[21]。本研究团队也曾报道过1例白血病患者由于肠道定植的CRKP异位引起的肝脓肿[18]。研究表明,导致肝脓肿的肺炎克雷伯菌大多属于高毒力菌株[22]。因此,如果CRKP进一步获得毒力质粒,可能增加其引起肝脓肿的风险。此外CRKP还可作为耐药基因的储存库和中转站,例如本研究中的肺炎克雷伯菌C35携带了至少3个可接合性耐药质粒,而其中的某些质粒(如编码blaCTX-M-199和mcr-1基因的IncI2质粒)极有可能来自于其他菌种,如大肠埃希菌。因毒力质粒缺乏抗性标记,本研究未证实肺炎克雷伯菌C35所携带毒力基因是否具备水平转移能力,hvKP毒力质粒在肠道细菌中的传播风险和传播能力有待进一步研究。
本研究所采用的大蜡螟毒力实验虽然不是评估细菌毒力最理想的方法,无法像小鼠模型一样准确区分高毒力和无毒力的肺炎克雷伯菌,细菌的接种量和大蜡螟幼虫的孵育时间等也会对结果判断产生影响[23],但由于大蜡螟幼虫毒力实验具有实验操作简单、成本低、结果易于观察等优点,目前仍在众多研究中被采用[3, 24, 25]。
综上所述,本研究报道的肠道定植hvKP所携带的耐药基因blaKPC-2、blaCTX-M-199、mcr-1和tet(A) type 1变异体可介导碳青霉烯类、β内酰胺酶抑制剂、多黏菌素和替加环素等多种临床重要抗菌药物耐药或敏感性降低。微生物实验室需加强CRKP的筛查和重要耐药基因和毒力基因的监测。
所有作者均声明不存在利益冲突



























