
调查我国实验室开展血液标本微生物细胞游离DNA(mcfDNA)宏基因组高通量测序(mNGS)的检测方法及质量保证情况。
2020年10月向来自全国的80家实验室发放了mNGS检测血液标本中mcfDNA的调查问卷。问卷内容包括mNGS分析前、分析中、分析后及性能确认开展情况4个部分。(1)分析前:对标本质量的要求,如对标本的采集、储存及运输条件等;(2)分析中:mcfDNA的提取流程、文库的质量要求、测序平台的使用及生物信息学分析软件等;(3)分析后:对mNGS的结果解释标准;(4)性能确认开展情况:对各类病原体的最低检出限。要求各实验室依据实际情况填写调查问卷。对上述调查问卷的回报结果进行统计分析。
80家实验室包括20家医疗单位和60家独立医学实验室。80.0%(64/80)的mNGS实验室检测血液中的mcfDNA时表示血浆及血清标本均可使用,其余实验室(16/80,20.0%)只采用血浆标本。参与调查的mNGS实验室所用的测序平台包括illumina 49家(61.3%),华大基因16家(20.0%),Ion Torrent 13家(16.3%),纳米孔测序2家(2.5%)。87.5%(70/80)的实验室使用第三方实验室搭建的集成分析工具,其他实验室(12.5%,10/80)使用开源软件自主搭建了分析平台。实验室间对mNGS结果的解释标准不一,其中标准化后病原体特异性序列数、相对丰度、基因组覆盖率及阴性对照中该微生物的检出情况是实验室考虑的主要因素。大部分实验室(76.3%,61/80)做过mcfDNA测序流程的性能确认实验,各实验室自建mNGS检测流程对革兰阳性菌、革兰阴性菌、真菌、寄生虫及其他病原体的最低检出限主要分布在10~100 拷贝/ml,对DNA病毒的检出限主要分布在500~1 000拷贝/ml。
各实验室之间的检测流程具有很大差异,为了确保检测结果的及时准确,需要各实验室积极优化mNGS检测流程,完善质量保证措施,应用于临床前规范开展性能确认工作。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
游离细胞DNA(cell-free DNA,cfDNA)指广泛存在于血液和各种体液中的DNA片段。一般认为cfDNA来源于死亡后的人体细胞和微生物被分解时释放到血液或体液中的小DNA片段[1]。cfDNA的高通量测序在胎儿异常的无创诊断、癌症监测和移植排斥反应的预测等方面具有广泛的应用[2, 3, 4]。通过宏基因组高通量测序(metagenomic next-generation sequencing,mNGS)检测微生物cfDNA(microbial cfDNA,mcfDNA)来诊断感染性疾病是cfDNA应用的另一重要领域。mNGS检测mcfDNA不依赖微生物培养,不易受前期使用抗生素的影响,无需假设即可在一次测试中检测多种病原体[5]。血液中的mcfDNA分析不仅可用于诊断血流感染[6],因血液流经全身,即使是深部感染或难以取样的部位感染时(如侵袭性真菌感染、肺炎或骨髓炎),也可将足量的病原体核酸释放入血液中用于感染性疾病的检测[7, 8, 9]。
尽管mNGS检测mcfDNA在感染性疾病诊断中前景广阔,但仍存在明显的局限性。最重要的瓶颈之一是此方法无法检测RNA病毒[10]。另外,如何确定检测到的mcfDNA是来源于病原体还是样本污染或定植菌群的一过性菌血症,仍然是困扰mNGS检测的难题[10]。目前,国内外广泛建立了mNGS检测平台并探究其在感染性疾病中的诊断价值。然而mNGS检测流程十分复杂[11],各实验室自建的mNGS检测流程存在很大差异[12],mNGS检测标准化进程缓慢。为全面了解国内实验室开展血液中mcfDNA mNGS检测的现状,本研究通过发放调查问卷的形式对其进行了调查研究,旨在促进我国mNGS检测的规范化与标准化。
来自19个省(自治区、直辖市)的80家开展mNGS检测的实验室填写了《血液(全血/血浆/血清)标本微生物cfDNA mNGS检测》调查问卷,其中有20家医疗单位和60家独立医学实验室。各实验室均自愿参加本次调查活动。
1.调查问卷:2020年10月向上述实验室发放电子调查问卷。问卷内容可分为mNGS分析前、分析中、分析后及性能确认开展情况4个部分。(1)分析前:主要包括对标本质量的要求,如对标本的采集、储存及运输条件等;(2)分析中:主要包括mcfDNA的提取流程,文库的质量要求,测序平台的使用及生物信息学分析软件等;(3)分析后:主要包括对mcfDNA mNGS的结果解释标准;(4)性能确认开展情况:调查实验室对各类病原体的最低检出限。实验室需依据实际情况填写调查问卷。
2. 统计学分析:对实验室回报结果进行整理,采用GraphPad Prism 8.0统计学软件进行数据的分析,计数资料以频数和百分比进行统计学描述。
参与调查的80家mNGS实验室主要分布在我国的中东部地区,以来自广东省的实验室最多(16家),其次是上海市(10家)、北京市(8家)、浙江省(7家)和湖北省(6家),其余各省份参加本次调查的实验室数量均不足6家。
80.0%(64/80)的mNGS实验室检测血液标本中mcfDNA时表示血浆及血清标本均可使用,其余实验室(16/80,20.0%)只采用血浆标本。各实验室对标本量的需求1~10 ml不等,多数实验室要求达3 ml及以上。游离DNA样本保存管是最常用的采血管(52/80,65.0%),一般在常温(15~25 ℃)下储存不超过7 d。此外,常规乙二胺四乙酸(EDTA)抗凝管也较常用(37/80,46.2%),使用常规EDTA 抗凝管采集的血液标本在4 ℃下储存,且不超过72 h。
48.8%(39/80)的实验室表示对提取的cfDNA浓度、体积及纯度无要求。其余41家实验室对cfDNA的质量要求不一,cfDNA浓度>0.1 ng/μl,体积>20 μl,吸光度值(A260/280)介于1.8~20.0之间是36.3%(29/80)的实验室提出的基本要求。
目前,基于边合成边测序原理的二代测序技术占据了mNGS的主要市场,在参与调查的mNGS实验室中,以illumina测序平台应用最为广泛(61.3%,49/80),另有16家(20%)实验室使用华大基因测序平台,13家(16.3%)实验室使用Ion Torrent半导体测序平台,2家(2.5%)实验室使用了纳米孔测序。66.7%(52/78)的基于二代测序原理的mNGS检测实验室要求插入文库片段大小在100~300 bp。74.4%(58/78)的实验室要求指出每份样本的常规测序量不低于20 M序列数。生物信息学分析的关键环节主要包括识别并去除标签序列、低质量及低复杂性序列的过滤、去除人源序列、序列比对及微生物序列注释等。各实验室采用的生物信息学分析流程存在较大差异,87.5%(70/80)的实验室使用了第三方实验室搭建的集成分析工具,如PMseq(10家)、Genseq PM(10家)及MAPMI(8家);12.5%(10/80)的其他实验室使用开源软件(如Kraken2、BWA等)自主搭建了分析平台。
各实验室对mNGS结果的解释有不同的标准,但mNGS检出的标准化后病原体特异性序列数、相对丰度、基因组覆盖率及阴性对照中该微生物的检出情况等是各家实验室考虑的主要因素。32家实验室表示能在24 h内完成从标本接收到出具最终报告;29家实验室表示需要24~48 h,19家实验室表示需要48 h以上。
61家实验室做过mcfDNA测序流程的性能确认实验,其中51家曾测试过本实验室mNGS检测流程对各类病原体的检出限(表1)。各实验室自建mNGS检测流程对革兰阳性菌、革兰阴性菌、真菌、寄生虫及其他病原体的最低检出限主要分布在10~100 拷贝/ml,对DNA病毒的检出限主要分布在500~1 000 拷贝/ml。
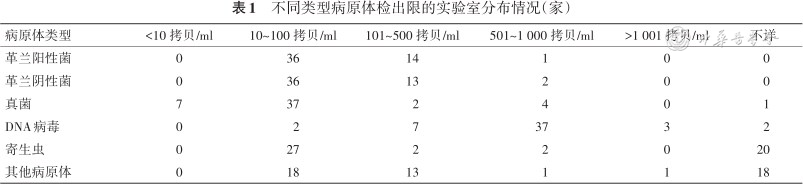
不同类型病原体检出限的实验室分布情况(家)
不同类型病原体检出限的实验室分布情况(家)
| 病原体类型 | <10 拷贝/ml | 10~100 拷贝/ml | 101~500 拷贝/ml | 501~1 000 拷贝/ml | >1 001 拷贝/ml | 不详 |
|---|---|---|---|---|---|---|
| 革兰阳性菌 | 0 | 36 | 14 | 1 | 0 | 0 |
| 革兰阴性菌 | 0 | 36 | 13 | 2 | 0 | 0 |
| 真菌 | 7 | 37 | 2 | 4 | 0 | 1 |
| DNA病毒 | 0 | 2 | 7 | 37 | 3 | 2 |
| 寄生虫 | 0 | 27 | 2 | 2 | 0 | 20 |
| 其他病原体 | 0 | 18 | 13 | 1 | 1 | 18 |
目前mNGS作为一种新的病原体感染检测技术在国内颇受关注,也逐步在向临床应用发展。然而,mNGS检测极为复杂,要保证mNGS检测的准确可靠,首先要建立一个准确的检测方法,确定质量控制关键点,实际检测中有严格的质量保证措施。从本调查问卷研究的结果看,我国实验室的血液mcfDNA mNGS检测流程存在很大差异,质量保证措施尚需全面完善。
质量保证可分为分析前、分析中和分析后3个阶段。分析前的质量保证包括标本的采集、运输及储存,血液标本等无菌标本在采集时应严格无菌操作,避免污染;有定植菌部位的标本采集时应尽量减少污染[13]。在本研究中,大部分实验室在检测mcfDNA时表示血浆和血清标本均可采用。目前尚无比较血浆和血清标本对mcfDNA影响的研究,但有文献指出血清中内源性核酸酶活性比血浆高14.9倍[14],血清中来自肿瘤和胎儿的cfDNA的含量及比例均低于血浆标本[15, 16]。
mNGS不适用于体外诊断产品,应按照实验室自建检测(laboratory developed tests,LDTs)的要求进行管理。美国《临床实验室改进修正案 1988》提出对于LDTs,在开展临床服务之前应对所建立的检测流程进行充分的性能确认工作,必须明确的检测性能包括准确性、精密度、可报告范围、参考区间、分析灵敏度和分析特异性等[17]。本研究发现参与调查的80家mNGS实验室中,有61家实验室做过mcfDNA测序流程的性能确认。用于性能确认准确性评估的样本应首选阳性临床血液标本,若数量不足可向健康人血液标本中添加纯化的微生物片段化核酸。将mNGS检测结果与已知结果比对,对于不一致的病原体判定结果采用经临床验证或美国食品和药物管理局批准的试验进行验证[18]。精密度包括批内精密度和批间精密度,欧洲临床病毒协会(European Society for Clinical Virology,ESCV)推荐从不低于20次运行中评估精密度的变化[19]。若mNGS报告定量结果,应在多个浓度下评估批内和批间精密度[20]。实验室应明确本mNGS检测流程可报告的病原体谱,mNGS临床报告应包括已知或怀疑有致病性的微生物,而不应报告非致病性微生物。检出限是指在95%置信区间下可检测到的最低微生物的核酸浓度[8]。此外,血液标本中存在的人源核酸或近缘菌株等会干扰mNGS检测的准确性,在开展性能确认时要评估这些关键因素对mcfDNA mNGS检测的影响[8]。在mNGS分析过程中,通过上述性能确认形成的质量控制关键点的使用是至关重要的。本调查研究发现近一半的实验室对提取的cfDNA的浓度、体积及纯度均无要求,然而,不合格的输入DNA将影响最终文库的质量及产出。各实验室应按照试剂的说明书或自行设计梯度实验摸索各检查点应设置的参数数值,不符合要求的实验材料不应进入下一环节。
在分析后的质量保证中,最重要的是如何从mNGS得到的数据中准确判断出病原体。血液mcfDNA mNGS检测易受采样污染、实验室环境背景微生物等干扰,影响真正病原体的判断。可以通过建立本实验室背景微生物数据库,开发新的统计模型等提高病原体判断的能力[21]。及时准确的mNGS检测报告的发布需要多学科的知识,应建立一个由临床实验室专家、临床医师、生物信息学专家等组成的报告解读团队,明确血流感染常见的病原体,在临床背景下共同讨论mNGS的报告结果[22, 23]。
本研究局限性:未调查核酸提取试剂盒、阴阳性对照、生物信息学具体分析流程、数据库使用、专家解读团队组成及性能确认的其他性能指标(如准确性、精密度、干扰)等情况。接下来将在全国开展血液标本mcfDNA mNGS检测的室间质量评价,来评价全国mNGS实验室的检测能力,将设计更加完善的调查问卷及质量评价方案,以此识别检测流程中存在的问题,并提出相应的改进措施。
所有作者均声明不存在利益冲突



























