
恶性肿瘤是一类死亡率较高的疾病,其发生、发展机制较为复杂,基因组测序技术对揭示肿瘤作用机制有一定意义。肿瘤组织中的细胞具有异质性,而细胞的空间位置分布对其异质性及组织生物功能有一定的影响。近几年,空间转录组测序技术发展迅速,可以对组织中细胞进行高通量、高分辨率的空间转录分析,从空间层面上展示转录组信息,揭示生物组织的细胞构成以及细胞间的相互作用关系。本文将对空间转录组测序技术在肿瘤中的应用进行阐述、前景进行展望。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
恶性肿瘤是一类发病率和死亡率均较高的疾病,大多数肿瘤患者的临床表现缺乏特异性,确诊时往往已经发展至晚期,预后较差[1]。尽管目前针对恶性肿瘤的治疗模式有所突破,但是肿瘤的晚期转移、化疗耐药等问题依旧棘手[2]。因此,进一步了解恶性肿瘤发生发展的分子机制极为重要。转录组测序(RNA sequencing,RNA-seq)技术通过高通量测序平台对基因组cDNA进行测序,同时计算不同信使RNA的表达量,分析转录本的结构与表达水平,还能发现未知转录本或稀有转录本,从而提供全面的转录组信息[3]。目前,多细胞水平的肿瘤转录组学研究得出的结果是此类群体细胞基因表达的平均值,数量较少的细胞亚群的表达信息会被该平均值所掩盖。单细胞转录组测序(single cell RNA sequencing,scRNA-seq)的研究结果虽然能够深入分析细胞之间的异质性,解析不同类型细胞的生理特点,但是难以还原细胞的原始位置信息[4]。而空间转录组测序技术(spatial transcriptome sequencing)恰好可以同时解决肿瘤异质性和空间定位问题。本文将综述空间转录组测序技术的原理以及其在肿瘤异质性、治疗等方面的应用。
空间转录组测序相关的技术最早可以追溯到20世纪60年代,在2016年瑞典卡罗琳斯卡研究所提出并详细介绍了空间转录组测序技术,随后该技术广泛发展并应用(图1)[5]。目前,用于空间转录组测序的现有技术主要分为四类:原位分子杂交技术(in situ hybridization,ISH)、激光捕获显微切割技术(laser capture microdissection,LCM)、原位测序技术(in situ sequencing,ISS)、基于分子矩阵空间转录组技术(spatial transcriptomics,ST)。
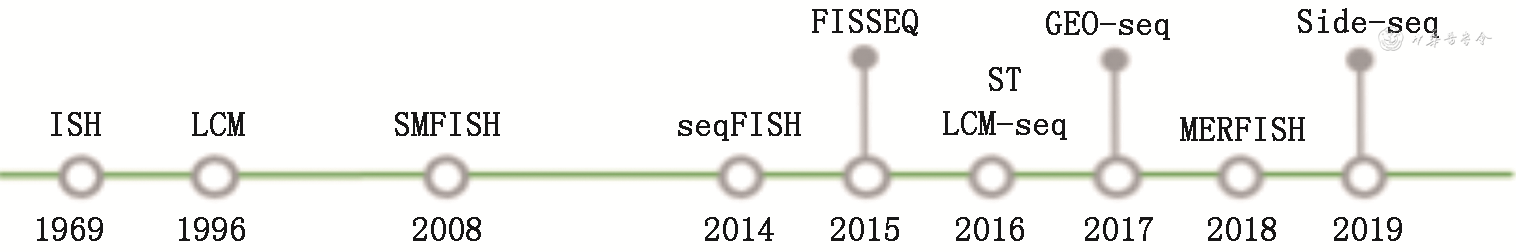

1. ISH:ISH是最为经典的一类空间转录组测序技术,通过将细胞或组织中目的RNA和其对应的寡核苷酸探针靶向互补、杂交后,在显微镜下成像分析[6]。该技术最初利用放射性同位素进行标记,后来改用荧光标记来减少环境污染、提高灵敏度[7]。随着RNA-seq技术的发展,Raj 等[8]提出了单分子荧光原位杂交技术(single-molecule FISH,SMFISH),该技术实现了单个细胞RNA定量和亚细胞分辨率下RNA定位。在此基础上,Lubeck等[9]提出了顺序荧光原位杂交技术(sequential FISH,seqFISH),通过单分子成像和连续条形码杂交,实现了在固定细胞中检测、定位12个基因。之后Eng等[10]改进了此项技术,提出seqFISH+,实现了单个细胞中10 000个基因的超分辨率成像。同时,一项多重容错的荧光原位分子杂交技术(multiplexed error-robust FISH,MERFISH)也迅速发展,该技术可描绘出100多万个细胞,靶向上万个基因[11, 12]。
2. LCM:LCM是一种以显微镜引导的切割技术,激光通过显微镜从异种样本中分离特定的细胞,以确保捕获的细胞和其周围组织形态完整[13, 14]。随着空间转录组学的迅速发展,基于LCM的技术也发展迅速。Nichterwitz等[15]提出的LCM-seq将LCM与RNA-seq结合,减少所需捕获细胞的数量,从而分析稀少的组织或细胞群。Chen等[16]提出了地域位置测序(geographical position sequencing,GEO-seq),将LCM与scRNA-seq结合,对少量细胞的转录组信息进行分析并保留它们的原始空间信息。
3. ISS:ISS较前两种技术新颖。它通过一种条形码锁式探针,与目标序列的任一侧杂交形成环状模板,之后通过连续滚环扩增,实现原位测序,从而发现测序信息与其位置之间的关联[17]。由于ISS探测的目标数量有限,Lee等[18]提出了荧光原位测序技术(fluorescent in situ sequencing,FISSEQ),将RNA转换为交联cDNA扩增子,同时联合共聚焦显微镜测序,对单细胞中全转录组进行分析。但是,该技术不易应用于组织切片。于是,Gyllborg等[19]提出基于杂交的原位测序技术,通过改进探针,以研究整个组织切片的大基因面板的空间构成,改善转录组的空间可视化,提高高通量成像的效率。
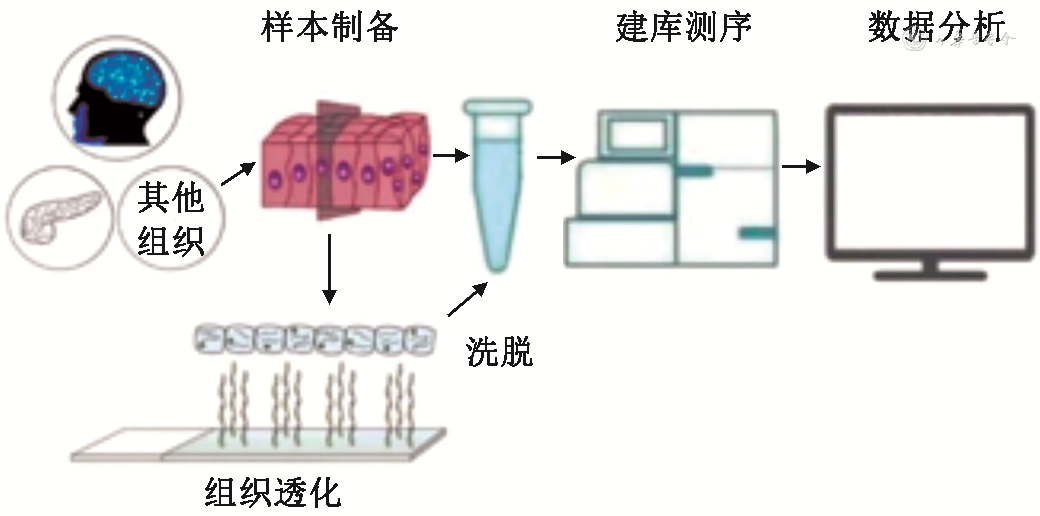
4. ST:ST是目前最新的一项技术,它通过定制的载玻片来捕获信使RNA。该载玻片有1 007个直径100 μm的微孔,每个微孔的寡核苷酸中都引入了位置条形码,组织透化后微孔捕获信使RNA,通过位置条形码将转录组测序数据分类到其相应的阵列特征,从而能够在单个组织切片中对转录组进行可视化和定量分析(图2)[5]。随着空间转录组学的发展,Vickovic等[20]提出了高清晰度空间转录组学,将载玻片中微孔直径缩短为2 μm,微孔内以百万珠阵列布局,从而实现对组织切片转录组的高分辨率分析。Rodriques等[21]提出的Slide-seq,则是将10 μm的带有条形码的微珠置于载玻片表面,并将组织切片中的RNA信息转移至该微珠上,从而获得单细胞分辨率的转录组空间信息。

肿瘤疾病一直以来都是人们想要攻克的一类疾病,随着测序技术的进步,一、二代测序技术已经在肿瘤学中广泛应用[22, 23, 24],scRNA-seq也已经在胆囊癌、结直肠癌等一系列恶性肿瘤中得以应用[25, 26]。空间转录组测序技术虽不完善,但随着人们研究的不断深入,其在肿瘤领域正在得到了越来越广泛的应用。人们可以通过空间转录组测序技术探索细胞的空间分布,从而对细胞间的相互作用有更加全面的理解。
1. 在肿瘤空间异质性研究中的应用:肿瘤组织内的细胞具有极大的异质性,同一肿瘤常常包含不同的克隆亚型,成纤维细胞、免疫细胞等所构成的肿瘤微环境也可在同一肿瘤的不同部位出现差异,肿瘤异质性的产生一定程度上增加了治疗难度。识别肿瘤组织内部各细胞亚群的空间分布位置或许对患者的诊疗有更大的帮助。空间转录组测序技术为描述肿瘤微环境、肿瘤异质性、疾病进展、识别肿瘤亚型提供了强有力的新工具。在临床中,胰腺癌的生存率很低,其微环境中细胞相互作用的机制尚需探索。Moncada等[27]采用ST与scRNA-seq相结合方法绘制了胰腺导管癌的空间图谱,发现了导管细胞、巨噬细胞、树突状细胞和肿瘤细胞亚群的富集程度受到了空间限制,其通过scRNA-seq识别的细胞类型标记将空间转录组表达谱反褶积为细胞类型比例,从而更好地探索亚群空间和功能之间的关系,这也首次分析了胰腺导管癌中细胞亚群的空间位置信息。前列腺癌在全球老年男性中的发病率居高不下,而寻找前列腺癌的新的分类方法迫在眉睫。Berglund等[28]对多灶性前列腺癌进行了空间转录组测序,使用ST方法绘制了基因表达谱,进一步展现其基因表达的异质性,发现得到的肿瘤表达区域要大于注释肿瘤区域,更准确地呈现了肿瘤病灶的范围,这也许会成为一种新的前列腺癌的分类方法,同时也对以后预测潜在癌症、前列腺上皮内瘤等一系列前列腺癌的发生发展提供重要的参考意义。有作者[29]通过FISH与LCM方法结合,研究浸润性乳腺癌前体肿瘤的基因和转录水平变化,发现导管腺癌在人表皮生长因子受体-2(human epidemal grouth factor receptor2,HER2)扩增之前存在转录组学和基因拷贝数变异(copy number varlations,CNV)的变化,这意味着癌前病变中存在一种细胞状态可为HER2扩增提供帮助。未来可利用此技术继续深入探索肿瘤进展的变化趋势。而胆囊癌作为一种新的癌中之王,其死亡率居高不下[30, 31],作者团队针对胆囊癌的肝侵犯及淋巴转移正在开展空间转录组测序工作。
2. 在肿瘤免疫研究中的应用:目前,空间转录组测序技术在肿瘤免疫学中初有进展,其对解释肿瘤免疫逃逸、免疫编辑等有一定的意义。随着肿瘤免疫学研究的不断深入,空间转录组测序技术是不二之选,这也很可能会影响肿瘤疾病未来治疗模式的变革。Salmén等[32]对乳腺癌进行空间转录组测序,利用ST方法分析了10例确诊为HER2+乳腺癌的患者的肿瘤组织切片,绘制了乳腺癌微环境中免疫细胞的三维图像。该研究发现肿瘤组织中侵袭性区域缺乏免疫细胞浸润,预后和总生存率较低,今后可以继续研究各种类型的肿瘤细胞与免疫细胞的相互作用,这可能是肿瘤治疗的关键。空间转录组测序技术一旦能够揭示肿瘤发生的免疫机制,这将大大推动肿瘤免疫治疗模式的发展。作者团队也正在利用空间转录组测序技术进行胆囊癌、胰腺癌的相关免疫机制的研究。
目前,空间转录组测序除了在肿瘤学中应用广泛外,还在发育生育学以及神经系统学等领域有所应用。在发育生育学领域,知道胚胎发育阶段细胞的空间分布位置和组织生长发育状况至关重要,空间转录组测序技术则能够很好地解决这类问题,该技术可以通过绘制空间基因表达图谱,很好地展现组织中全部细胞的异质性,还能为组织器官生长发育的机制探索提供基础。Asp等[33]对胚胎心脏的3个发育阶段进行了空间转录组测序,结合了ST、scRNA-seq和ISS 3种方法,绘制了心脏的空间基因表达谱,这将有利于进一步推进人类心脏发生发展机制的研究。在神经科学领域,下丘脑控制着基本的社会行为和体内平衡功能,其功能与空间结构密切相关,而绘制出下丘脑的空间基因表达图谱将会为神经系统疾病的致病机制研究提供坚实的基础。有作者[11]用scRNA-seq进行细胞亚群分类标记,又利用MERFISH对标记亚群进行成像,从而构建了小鼠下丘脑视前区的细胞空间图谱。此外,他们利用MERFISH和诱导即刻早期基因揭示不同社会行为激活的细胞亚群及其在不同行为中的功能。因此,我们可以利用空间转录组测序技术深入了解不同类型的细胞如何在不同神经系统疾病中发挥功能。空间转录组学有望将分子细胞类型同生理学、功能学、形态学联系在一起,从而克服了神经科学的挑战。
目前,四类空间转录组测序技术均发展迅速,但各自均有其局限性。比如,ST技术是现在的主流,它仍存在分辨率未达到单细胞水平以及成本昂贵等今后亟需解决的问题。近两年,很多研究团队通过将scRNA-seq和空间转录组测序结合来进行肿瘤研究,综合基因表达数据特征和病理学家注释的结果进一步阐明肿瘤的发生发展过程,以实现早期诊断。消化道肿瘤症状往往隐匿,发现时多半已经晚期,及早发现可以大大提高治愈率。不同技术之间的相互结合,分析方法的不断更新,在肿瘤中应用已越来越广泛。例如,将免疫荧光技术引入空间转录组学中实现了转录组学与蛋白质组学的结合,研究结果从空间位置角度揭示了肿瘤转录水平与翻译水平之间的联系。未来可以进一步探索多种技术的结合,向多组学分析发展;同时,构建时间与空间相结合的动态转录组测序数据库,从而清楚认识肿瘤的发展,真正克服肿瘤疾病。
所有作者均声明不存在利益冲突



























