
评价宏基因组高通量测序(mNGS)在免疫缺陷患者肺部感染诊断与治疗中的应用价值。
回顾性选取2018年11月至2022年5月在解放军总医院呼吸与危重症医学部第一医学中心重症监护室的78例免疫缺陷肺部感染患者[男55例,女23例,年龄(50.3±16.9)岁]和61例非免疫缺陷肺部感染患者[男42例,女19例,年龄(63.6±15.9)岁]。两组患者在临床诊断为肺部感染的同时,均行支气管肺泡灌洗液(BALF)的mNGS及常规微生物学检测(CMTs)。比较两种检测方法的诊断阳性率、病原体检出率以及与临床符合率的差异。同时比较两组患者根据mNGS检测结果抗感染治疗策略调整率的差异。
免疫缺陷组与非免疫缺陷组肺部感染患者mNGS诊断阳性率分别为94.9%(74/78)和82.0%(50/61),CMTs诊断阳性率分别为64.1%(50/78)和75.4%(46/61),免疫缺陷组mNGS与CMTs诊断阳性率差异有统计学意义(P<0.001)。mNGS在免疫缺陷组中耶氏肺孢子菌、巨细胞病毒的检出率分别为41.0%(32/78)、37.2%(29/78),在非免疫缺陷组中肺炎克雷伯菌、鹦鹉热衣原体、嗜肺军团菌的检出率分别为16.4%(10/61)、9.8%(6/61)、8.2%(5/61),均高于CMTs的1.3%(1/78)、7.7%(6/78)、4.9%(3/61)、0、0(均P<0.05)。免疫缺陷组mNGS、CMTs与临床符合率分别为89.7%(70/78)、43.6%(34/78),差异有统计学意义(P<0.001)。非免疫缺陷组mNGS、CMTs与临床符合率分别为83.6%(51/61)、62.3%(38/61),差异有统计学意义(P=0.008)。免疫缺陷组根据mNGS病原学结果,抗感染治疗策略调整率为87.2%(68/78),非免疫缺陷组抗感染治疗策略调整率为60.7%(37/61),差异有统计学意义(P<0.001)。
在免疫缺陷肺部感染患者中,与CMTs相比,mNGS在诊断阳性率、混合型感染诊断率、病原体检出率及指导抗感染治疗策略的调整方面更具优势,值得临床推广应用。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
近年来,免疫缺陷患者危重症比例已升至重症监护室(intensive care unit,ICU)入院患者总数1/3,其主要病因是肺部感染[1, 2]。肺部感染是造成免疫缺陷患者死亡的主要原因之一[3],亦成为困扰ICU医师的棘手问题。免疫缺陷患者易感染常见病原体及多种条件致病菌,且多为混合型感染,病情进展迅速,常发展为重症肺炎。致病病原体如不能及时准确鉴定,将导致过度诊治和产生不必要的医疗费用及不良预后[4]。早期准确识别致病病原体是实施精准抗感染治疗和改善临床结局的关键问题[1, 2, 3, 4, 5]。然而,传统病原学检测流程复杂、检测效率低、耗时长[6],不能完全满足免疫缺陷肺部感染患者及时诊治的需要。
近年来,宏基因组高通量测序(metagenomics next-generation sequencing,mNGS)不依赖培养,直接对标本中的核酸无偏倚检测病原微生物序列,且不易受前期抗生素治疗的影响,对常规病原微生物检测阴性、经验治疗失败、不明原因的危重感染以及新发突发传染病的病原体发现具有独特价值[7, 8],在感染领域备受关注。对于重症肺部感染患者,mNGS检测病原体的阳性率为80.0%~90.3%,而常规微生物学检测(conventional microbiological tests,CMTs)如微生物学培养、抗原抗体检测等总的阳性率仅为30.0%~40.0%[4,9]。本研究旨在评价mNGS在免疫缺陷患者肺部感染中进行病原学诊断和抗感染治疗策略调整的应用价值。
本研究为横断面研究,通过解放军总医院伦理委员会审核(伦理号:S2019-266-02)。选取2018年11月至2022年5月解放军总医院第一医学中心呼吸与危重症医学科重症监护室(respiratory intensive care unit,RICU)经临床诊断为肺部感染的患者139例,包括免疫缺陷患者78例,非免疫缺陷患者61例。所有患者均同时行支气管肺泡灌洗液(bronchoalveolar lavage fluid,BALF)mNGS及CMTs。免疫缺陷定义:原发免疫缺陷疾病,活动性恶性肿瘤,癌症化疗,实体器官移植,造血干细胞移植,接受皮质类固醇治疗(相当于泼尼松≥20 mg/d、超过14 d,或累积剂量>600 mg),接受生物免疫调节剂治疗,接受免疫抑制药物(如环磷酰胺、环孢素、他克莫司等)治疗[2]。
1. 社区获得性肺炎(community-acquired pneumonia,CAP)临床诊断标准:(1)社区发病。(2)肺炎相关临床表现:①新近出现的咳嗽、咳痰或原有呼吸道疾病症状加重,伴或不伴脓痰、胸痛、呼吸困难及咯血;②发热;③肺实变体征和(或)闻及湿啰音;④外周血白细胞>10×109/L或<4×109/L,伴或不伴细胞核左移。(3)胸部影像学检查:显示新出现的斑片状浸润影、叶或段实变影、磨玻璃影或间质性改变,伴或不伴胸腔积液。符合(1)与(3),以及(2)中的任何1项,并除外肺结核、肺部肿瘤、非感染性肺间质性疾病、肺水肿、肺不张、肺栓塞、肺嗜酸粒细胞浸润症及肺血管炎等,可建立临床诊断[10]。
2. 医院获得性肺炎(hospital-acquired pneumonia,HAP)/呼吸机相关性肺炎(ventilator-associated pneumonia,VAP)临床诊断标准:HAP 是指患者住院期间没有接受有创机械通气、未处于病原感染的潜伏期,而于入院48 h后新发生的肺炎。VAP是指气管插管或气管切开患者接受机械通气 48 h后发生的肺炎,机械通气撤机、拔管后48 h内出现的肺炎也属于VAP范畴。HAP/VAP临床诊断标准[11]:胸部X线或CT显示新出现或进展性的浸润影、实变影或磨玻璃影,同时具有下列3种临床症候中的2种或以上,可建立临床诊断:(1)体温>38 ℃;(2)脓性气道分泌物;(3)外周血白细胞计数>10×109/L或<4×109/L。
3. 资料收集:收集患者的人口学资料、基础疾病、急性生理及慢性健康评价(acute physiology and chronic health evaluation,APACHE)-Ⅱ评分、氧分压/吸入氧浓度比值(ratio of partial pressure of arterial oxygen to fraction of inspired oxygen,P/F)、临床表现、实验室检查、mNGS及CMTs结果、影像学检查、ICU入院时的初始抗感染方案以及根据mNGS病原学结果进行调整的情况、住院时间、RICU住院时间及转归等。
4. BALF提取:所有患者入RICU后24~48 h内均行支气管镜检查及肺泡灌洗。对于无人工气道的患者,经鼻或经口进入气管;有人工气道的患者,直接经人工气道进入气管。首先清理上气道分泌物,并应用碘伏棉签消毒鼻腔或人工气道开口。其次,根据影像学检查结果,若为局限性病变,选取靶病灶部位进行灌洗;若为弥漫性病变,选取右肺中叶或左肺舌叶进行灌洗。每次灌洗温生理盐水20 ml,进行回吸收。将BALF标本分份,进行CMTs和mNGS检测。第一瓶标本通常送病原体抗原检测,第二瓶标本送细菌、真菌涂片和培养以及抗酸染色,第三瓶标本送mNGS检测,其余标本进一步分份进行微生物学分析。
5. mNGS操作流程:参照文献进行核酸提取、文库构建、高通量测序、生物信息学分析和病原体数据解释[4]。具体操作流程:(1)标本处理和DNA提取:取BALF标本600 μl,使用预设的破壁仪程序进行破壁处理,然后根据试剂盒说明书提取DNA。提取的DNA经转座酶建库给待测核酸加上标签序列,制备好的文库经过纯化、扩增、再纯化后,使用Qsep1和Qubit分别对文库片段大小和文库浓度进行定量(微远建库试剂盒)。然后依据预设的上机数据量取定量文库混合后,使用Nextseq 550Dx测序仪进行测序。(2)数据分析:测序数据下机后,去除低质量和长度<35 bp的数据,以获得高质量数据。通过BWA(BWA:http://bio-bwa.sourceforge.net/)网站比对,将高质量数据中与人参考基因组序列相同的数据去除。剩余数据在去除低复杂度短序列(reads)后,与专用的微生物大数据库比对,并将比对后的数据按照病毒、细菌、真菌和寄生虫等进行分类和排列。
6. mNGS阳性判断标准[7,12, 13, 14]:细菌(除外结核及非结核分支杆菌)和病毒覆盖率高于其他微生物10倍,或真菌属(除外霉菌)覆盖率高于其他微生物5倍,或该微生物在种属水平相对丰度>30%且有文献支持其肺部致病性;对于结核分枝杆菌、鼠疫耶尔森菌、布鲁菌,在排除污染的前提下,检出1条特异性序列;对于非结核分枝杆菌,其reads数在细菌列表中位于前10位。符合以上任意1项即可判断为mNGS阳性。
7. CMTs阳性判断标准[10,11]:(1)血培养、合格下呼吸道标本(涂片镜检,鳞状上皮细胞<10个/低倍镜视野和多核白细胞>25个/低倍镜视野,或二者比值<1∶2.5)培养阳性,或胸腔积液培养阳性;(2)鼻咽或口咽拭子、下呼吸道标本实时聚合酶链反应(real time-polymerase chain reaction,RT-PCR)检测病毒阳性;(3)军团菌尿抗原或肺炎链球菌尿抗原检测阳性;(4)肺组织标本病理学、细胞病理学或直接镜检到真菌,并有组织损害的证据;(5)非典型病原体或病毒的血清IgM抗体由阴转阳,或急性期和恢复期双份血清特异性IgG抗体滴度呈4倍或4倍以上变化。符合以上任意1项即可判断为CMTs阳性。
8. 符合率评估:分别评估mNGS、CMTs诊断结果与临床的符合率,以及二者之间的符合率。mNGS、CMTs与临床的符合率是指二者检测结果与胸部影像、抗生素应用及临床结局的符合率。如免疫缺陷患者mNGS诊断为肺孢子菌肺炎,胸部影像需表现为弥漫性磨玻璃样改变,抗生素应用需是复方磺胺甲 唑,经治疗后胸部影像磨玻璃影应吸收,临床症状应好转,如mNGS、CMTs的检测结果与此类情况完全符合称为与临床符合。mNGS与CMTs检测结果的符合率是指两种检测方法检测的结果完全匹配。
唑,经治疗后胸部影像磨玻璃影应吸收,临床症状应好转,如mNGS、CMTs的检测结果与此类情况完全符合称为与临床符合。mNGS与CMTs检测结果的符合率是指两种检测方法检测的结果完全匹配。
9. 抗感染治疗策略的调整:根据mNGS病原学检测结果,进一步调整两组患者抗感染治疗策略。
使用SPSS 23.0统计软件进行数据处理。正态分布的计量资料以表示,组间比较采用独立样本t检验。计数资料以例数及百分比表示,组间比较采用χ2检验。双侧检验,检验水准α=0.05。
78例免疫缺陷患者中,77例(98.7%)接受长期激素、免疫抑制药物治疗或者化疗。78例患者基础疾病分别为血液系统恶性肿瘤35例(44.9%),其中合并造血干细胞移植后11例(14.1%),自身免疫性疾病17例(21.8%),实体器官肿瘤放化疗后8例(10.3%),肾脏系统疾病7例(9.0%),实体器官移植术后5例(6.4%),神经系统疾病4例(5.1%),肺部疾病(肺间质纤维化)及先天性免疫缺陷各1例。与非免疫缺陷患者组比较,免疫缺陷患者组发病年龄较低,病变呈双肺分布以及磨玻璃影所占比例较高(均P<0.001)。结果见表1。
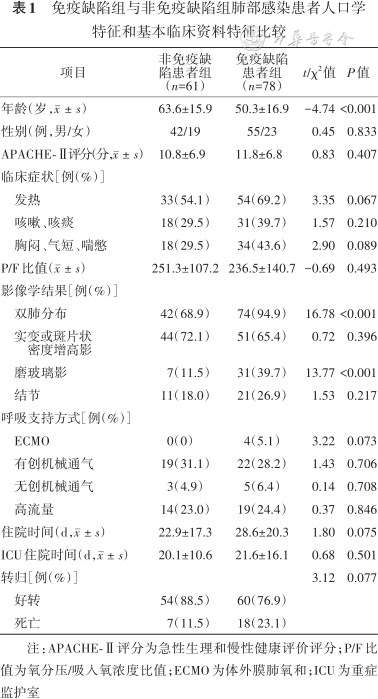
免疫缺陷组与非免疫缺陷组肺部感染患者人口学特征和基本临床资料特征比较
免疫缺陷组与非免疫缺陷组肺部感染患者人口学特征和基本临床资料特征比较
| 项目 | 非免疫缺陷患者组(n=61) | 免疫缺陷患者组(n=78) | t/χ2值 | P值 |
|---|---|---|---|---|
| 年龄(岁,) | 63.6±15.9 | 50.3±16.9 | -4.74 | <0.001 |
| 性别(例,男/女) | 42/19 | 55/23 | 0.45 | 0.833 |
| APACHE-Ⅱ评分(分,) | 10.8±6.9 | 11.8±6.8 | 0.83 | 0.407 |
| 临床症状[例(%)] | ||||
| 发热 | 33(54.1) | 54(69.2) | 3.35 | 0.067 |
| 咳嗽、咳痰 | 18(29.5) | 31(39.7) | 1.57 | 0.210 |
| 胸闷、气短、喘憋 | 18(29.5) | 34(43.6) | 2.90 | 0.089 |
| P/F比值() | 251.3±107.2 | 236.5±140.7 | -0.69 | 0.493 |
| 影像学结果[例(%)] | ||||
| 双肺分布 | 42(68.9) | 74(94.9) | 16.78 | <0.001 |
| 实变或斑片状密度增高影 | 44(72.1) | 51(65.4) | 0.72 | 0.396 |
| 磨玻璃影 | 7(11.5) | 31(39.7) | 13.77 | <0.001 |
| 结节 | 11(18.0) | 21(26.9) | 1.53 | 0.217 |
| 呼吸支持方式[例(%)] | ||||
| ECMO | 0(0) | 4(5.1) | 3.22 | 0.073 |
| 有创机械通气 | 19(31.1) | 22(28.2) | 1.43 | 0.706 |
| 无创机械通气 | 3(4.9) | 5(6.4) | 0.14 | 0.708 |
| 高流量 | 14(23.0) | 19(24.4) | 0.37 | 0.846 |
| 住院时间(d,) | 22.9±17.3 | 28.6±20.3 | 1.80 | 0.075 |
| ICU住院时间(d,) | 20.1±10.6 | 21.6±16.1 | 0.68 | 0.501 |
| 转归[例(%)] | 3.12 | 0.077 | ||
| 好转 | 54(88.5) | 60(76.9) | ||
| 死亡 | 7(11.5) | 18(23.1) | ||
注:APACHE-Ⅱ评分为急性生理和慢性健康评价评分;P/F比值为氧分压/吸入氧浓度比值;ECMO为体外膜肺氧和;ICU为重症监护室
免疫缺陷组肺部感染患者中,mNGS与CMTs检测阳性率差异有统计学意义(χ2=22.65,P<0.001)。非免疫缺陷组肺部感染患者中,mNGS与CMTs检测阳性率差异无统计学意义(χ2=0.78,P=0.377)。与非免疫缺陷组患者相比,免疫缺陷组患者mNGS检测混合型感染比例更高,结果见表2。
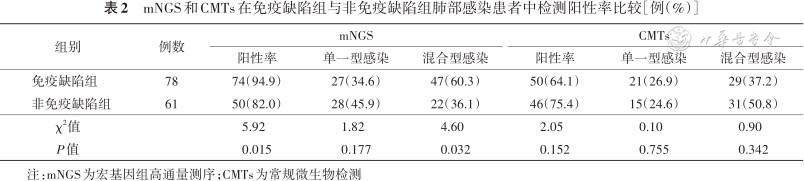
mNGS和CMTs在免疫缺陷组与非免疫缺陷组肺部感染患者中检测阳性率比较[例(%)]
mNGS和CMTs在免疫缺陷组与非免疫缺陷组肺部感染患者中检测阳性率比较[例(%)]
| 组别 | 例数 | mNGS | CMTs | ||||
|---|---|---|---|---|---|---|---|
| 阳性率 | 单一型感染 | 混合型感染 | 阳性率 | 单一型感染 | 混合型感染 | ||
| 免疫缺陷组 | 78 | 74(94.9) | 27(34.6) | 47(60.3) | 50(64.1) | 21(26.9) | 29(37.2) |
| 非免疫缺陷组 | 61 | 50(82.0) | 28(45.9) | 22(36.1) | 46(75.4) | 15(24.6) | 31(50.8) |
| χ2值 | 5.92 | 1.82 | 4.60 | 2.05 | 0.10 | 0.90 | |
| P值 | 0.015 | 0.177 | 0.032 | 0.152 | 0.755 | 0.342 | |
注:mNGS为宏基因组高通量测序;CMTs为常规微生物检测
免疫缺陷组肺部感染患者中,最常见的病原体为耶氏肺孢子菌、巨细胞病毒(cytomegalovirus,CMV),其次为鲍曼不动杆菌、铜绿假单胞菌、肺炎克雷伯杆菌。非免疫缺陷组肺部感染患者中,最常见的病原体是鲍曼不动杆菌、肺炎克雷伯菌、铜绿假单胞菌、鹦鹉热衣原体、嗜肺军团菌等。mNGS在免疫缺陷组患者中检测耶氏肺孢子菌、巨细胞病毒及在非免疫缺陷患者中检测肺炎克雷伯菌、鹦鹉热衣原体、嗜肺军团菌等病原体检出率高于CMTs(均P<0.05)。结果见表3。
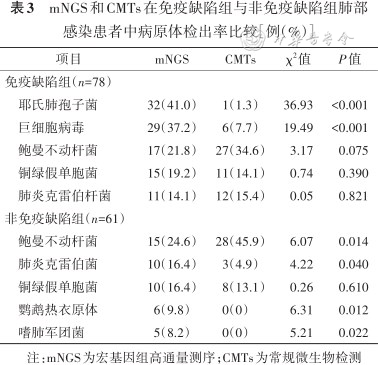
mNGS和CMTs在免疫缺陷组与非免疫缺陷组肺部感染患者中病原体检出率比较[例(%)]
mNGS和CMTs在免疫缺陷组与非免疫缺陷组肺部感染患者中病原体检出率比较[例(%)]
| 项目 | mNGS | CMTs | χ2值 | P值 |
|---|---|---|---|---|
| 免疫缺陷组(n=78) | ||||
| 耶氏肺孢子菌 | 32(41.0) | 1(1.3) | 36.93 | <0.001 |
| 巨细胞病毒 | 29(37.2) | 6(7.7) | 19.49 | <0.001 |
| 鲍曼不动杆菌 | 17(21.8) | 27(34.6) | 3.17 | 0.075 |
| 铜绿假单胞菌 | 15(19.2) | 11(14.1) | 0.74 | 0.390 |
| 肺炎克雷伯杆菌 | 11(14.1) | 12(15.4) | 0.05 | 0.821 |
| 非免疫缺陷组(n=61) | ||||
| 鲍曼不动杆菌 | 15(24.6) | 28(45.9) | 6.07 | 0.014 |
| 肺炎克雷伯菌 | 10(16.4) | 3(4.9) | 4.22 | 0.040 |
| 铜绿假单胞菌 | 10(16.4) | 8(13.1) | 0.26 | 0.610 |
| 鹦鹉热衣原体 | 6(9.8) | 0(0) | 6.31 | 0.012 |
| 嗜肺军团菌 | 5(8.2) | 0(0) | 5.21 | 0.022 |
注:mNGS为宏基因组高通量测序;CMTs为常规微生物检测
两组患者中,mNGS与临床符合率均高于CMTs与临床符合率(χ2=37.39、7.020,均P<0.01)。两组患者间比较,CMTs与临床符合率、mNGS与CMTs符合率差异均有统计学意义(均P<0.05)。结果见表4。
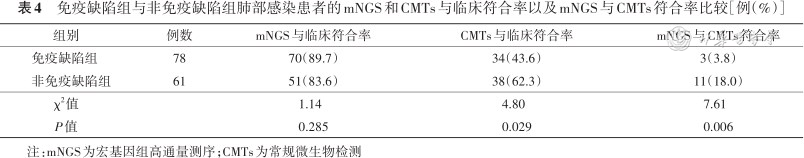
免疫缺陷组与非免疫缺陷组肺部感染患者的mNGS和CMTs与临床符合率以及mNGS与CMTs符合率比较[例(%)]
免疫缺陷组与非免疫缺陷组肺部感染患者的mNGS和CMTs与临床符合率以及mNGS与CMTs符合率比较[例(%)]
| 组别 | 例数 | mNGS与临床符合率 | CMTs与临床符合率 | mNGS与CMTs符合率 |
|---|---|---|---|---|
| 免疫缺陷组 | 78 | 70(89.7) | 34(43.6) | 3(3.8) |
| 非免疫缺陷组 | 61 | 51(83.6) | 38(62.3) | 11(18.0) |
| χ2值 | 1.14 | 4.80 | 7.61 | |
| P值 | 0.285 | 0.029 | 0.006 |
注:mNGS为宏基因组高通量测序;CMTs为常规微生物检测
78例免疫缺陷组患者中,根据mNGS病原学结果,26例增加了抗感染药物的种类,42例减少了抗感染药物的种类或给予降阶梯治疗,抗感染治疗策略调整率为87.2%(68/78)。61例非免疫缺陷组患者中,根据mNGS病原学结果,14例增加了抗感染药物的种类,23例减少了抗感染药物的种类或给予降阶梯治疗,抗感染治疗策略调整率为60.7%(37/61)。两组患者抗感染治疗策略调整率差异有统计学意义(χ2=13.03,P<0.001)。
本研究以BALF的mNGS为基础,比较免疫缺陷组与非免疫缺陷组肺部感染患者病原学诊断的有效性,以及根据mNGS结果调整抗感染治疗策略的可行性,mNGS在免疫缺陷组肺部感染患者中对确定感染类型及其个体化抗感染治疗策略调整方面是一个非常有效的工具。
研究结果显示,与CMTs相比,mNGS检测在两组患者中的阳性率都更高,与之前的研究结果一致[4,15, 16, 17]。同时本研究中两组患者CMTs阳性率较既往研究结果明显升高[4,9],考虑与本课题组和微生物科进行多学科病例讨论、及时有效沟通及实施BALF的标准化留取流程相关。免疫缺陷组与非免疫缺陷组患者mNGS与CMTs的符合率均较低。缺乏一致性的原因是mNGS结果中检测到的病原学谱更广。其次,mNGS检测提示免疫缺陷组肺部感染患者混合型感染多于非免疫缺陷组患者。近年来,临床医师也逐渐意识到免疫缺陷肺部感染患者以混合型感染为主,而病原体的明确诊断却异常困难[18]。mNGS对肺部混合型感染病原体检测的敏感性优于CMTs,与文献报道结果一致[19]。免疫缺陷组与非免疫缺陷组患者的肺部感染病原菌谱不同。在免疫缺陷组患者中,病原菌检出最多的是耶氏肺孢子菌和CMV,且二者同时感染率较高,mNGS对二者的检测敏感性明显优于CMTs。再次,mNGS对于鹦鹉热衣原体等生物安全性要求高的病原菌检测有显著优势[20]。因此,mNGS是一种有效的微生物学检测方法,特别是对免疫缺陷肺部感染患者的混合型感染的检测[9,21]。
本研究同时对免疫缺陷组与非免疫缺陷组肺部感染患者进行了基于mNGS病原学结果的抗感染管理策略的比较研究。与非免疫缺陷组患者相比,免疫缺陷组肺部感染患者的抗感染治疗调整次数更多。在本研究中,78例免疫缺陷患者中,有42例患者根据mNGS病原学结果减少了抗生素使用种类或给予降阶梯治疗。虽然临床医师已经逐渐认识到免疫缺陷肺部感染患者的特殊性,但针对此类患者的最佳抗感染治疗策略仍不确定。过度抗感染治疗可能导致耐药性增加和医疗资源浪费[2,22, 23]。文献报道,在免疫缺陷的重症肺炎所致急性呼吸窘迫综合征患者中,mNGS检测可显著缩短ICU住院时间、机械通气时间,降低ICU费用[24]。mNGS的独特优势可能有助于临床医师更全面地评估经验性抗感染治疗策略,并对免疫缺陷肺部感染患者做出有效的调整。
综上所述,mNGS在免疫缺陷肺部感染患者中获得了较高的微生物检出率。在免疫缺陷组与非免疫缺陷组患者中,肺部感染最常见的病原体谱不同。mNGS检测可能是早期识别肺部感染潜在病原体的有力工具,同时在实施经验性抗感染治疗的靶向调整方面具有优势。
段智梅, 沈志勇, 胡晔, 等. 宏基因组高通量测序在免疫缺陷患者肺部感染诊断与治疗中的应用价值[J]. 中华医学杂志, 2023, 103(25): 1885-1891. DOI: 10.3760/cma.j.cn112137-20221226-02703.
所有作者均声明不存在利益冲突



























