
传统的生存分析方法虽在生物医学领域已有广泛应用,但需满足一些前提假设。随机生存森林方法可克服这一弱点。本文以美国梅奥诊所的肝脏原发性胆汁肝硬化的数据为例,从随机生存森林的原理、建模步骤、实例演示和适用性讨论等方面进行阐述,以期为读者进行生存分析提供新的思路和方法。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
经全国继续医学教育委员会批准,本刊开设继教栏目,每年从第1 期至第10期共刊发10篇继教相关文章,文后附5道单选题,读者阅读后可扫描标签二维码答题,每篇可免费获得Ⅱ类继教学分0.5分,全年最多可获5.0分。
生存分析是一种常用的分析研究对象结局事件发生时间的分析方法,广泛地应用于临床医学、公共卫生和生物学等不同研究领域,解释在时间的影响下各因素与发生事件的关系。在比较各组之间的生存差异时,由于运用了假设检验的思想,部分常用的传统生存分析方法对于数据的分布特点有一定的要求,如Cox比例风险回归需要资料满足比例风险假设这一前提条件[1]。当资料不服从前提条件要求时,需要通过分层或者数据转换等使资料满足其假设后再进行分析,实质上大大限制了其应用范围,但很多研究者并未对这些前提条件给予足够的重视,从而导致了生存分析方法的误用。2008年,Ishwaran等[2]将随机森林(Random Forest)法和传统生存分析相结合,构建了随机生存森林(Random Survival Forest)模型,可克服传统生存分析方法的弱点,有着更广的应用范围。该模型本质上与随机森林法相似,均使用Bootstrap法,以有放回的形式随机抽取样本并形成多个二元决策树,进而组成随机生存森林。构建随机生存森林模型不需要满足特定的前提性假设,对于原始数据无特定要求。本研究以梅奥诊所的肝脏原发性胆汁肝硬化的数据为例,从随机生存森林模型的原理、建模方法、案例演示和适用性讨论四个方面展开阐述。
随机生存森林的基本单位是二元生存树,即传统的二元决策树(Binary Decision Tree)的拓展。二元决策树是一种树形数据结构,输入数据在经过二元决策树的节点时,会被节点的判断条件分裂成两组新的数据,直到输入数据均为同一类别为止,而数据最终存在于的节点即为其所属类别[3]。二元生存树的数据结构与二元决策树基本一致,仅在节点分裂的标准上有区别。数据经过生存树的节点时,节点会以最大化生存差异的标准分裂数据,直到输入数据的事件数不小于特定阈值,而此节点就为终端节点。常用的节点分裂标准为log-rank分数[2]。
随机生存森林通过训练大量生存树,以表决的形式,从个体树之中加权选举出最终的预测结果。构建随机生存森林的一般流程为:首先,模型通过“自助法”(Bootstrap)将原始数据以有放回的形式随机抽取样本,建立样本子集,并将每个样本中37%的数据作为袋外数据(Out-of-Bag Data)排除在外。其次,对每一个样本随机选择特征构建其对应的生存树。再次,利用Nelson-Aalen法估计随机生存森林模型的总累积风险。最后,使用袋外数据计算模型准确度。
定义(T(1, h),S(1, h)),……,(T(n, h),S(n, h))为输入的删失数据。其中,H为终端节点且h∈H。Ti表示第i个个体在节点h的生存时间,Si表示此个体的状态(0为删失,1为事件)。定义D(i, h)和Y(i,h)分别为在此终端节点时的事件(死亡)数和总个体数。根据Nelson-Aalen估计量,可以得到单个终端节点的累积风险函数[2]:
假设每个样本有维度为d的协变量X,那么由于生存树的二分性,Xi会只存在于单个终端节点h中。因此,终端节点在Xi的条件下的累积风险函数为:
由于原始数据被分为袋内数据和袋外数据,在估算模型总体的累积风险时,要分别计算。当个体属于袋外数据时,定义I(i, b)=1;除此之外,I值为0。使为第b个袋外样本,那么其总累积风险为:
对于袋内样本,其总累计风险为:
随机生存森林使用了Harrell的一致性指数(concordance index)来计算其准确度[2]。一致性指数是评估生存分析模型的常用指标,一致性指数的值与模型的优劣呈正相关。首先,将任意的袋内数据和预测数据两两分组,如果较短的生存时间对应了较差的预测结果,此组的一致性计为1;若较短的生存时间对应了较好的预测结果,此组的一致性则计为0;其他情况为0.5。生存时间相同且没有事件发生的数据组不被包含在内。因此,模型的指数(C)为:
袋外数据的预测错误率则为1-C。
由于随机生存森林模型选取输入数据的大量特征作为其分裂节点来构建模型,该模型保留了冗杂的变量。然而,并不是所有的变量在机器建立模型时都有积极意义,因此,通过变量筛选,我们可以了解各变量在建立模型时的作用。评判变量重要性的方法有两种,分别是VIMP(variable importance)法和最小深度(minimal depth)法。VIMP法的计算方法的原理是将袋外数据放入生存树中,令其随机分配到任一子节点;计算新的总累积风险;VIMP为原始错误率和新错误率的差。因此,VIMP越大就意味着此变量对于模型的准确度影响越大,此变量的重要性就越高。不同于VIMP,最小深度法则认为,需要保留的变量应该为能够区分最多数据的变量,即最靠近根节点的节点。因此,最小深度法认为值越小的变量则对模型越重要。
Ishwaran等在2020年1月20日开发了专门用于随机生存森林的R语言库“randomForestSRC”,截至2020年9月,最新的版本为2.9.3。不同于先前的随机生存森林库“randomSurvivalForest”“randomForestSRC”的开发基于OpenMP,可以进行多线程并行的计算,进而提高模型的构建效率。本次更新还提供了解决多变量数据缺失值和非均衡数据等问题的函数。为了能更灵活地可视化随机生存森林模型,John开发了“ggRandomForests”库,截至2020年9月,最新版本为2.0.1。用户仅需在R软件使用如下命令即可安装此库:
>install.packages(“randomForestSRC”)
>install.packages(“ggRandomForests”)
以美国梅奥诊所在1974—1984年间收集的原发性胆汁性胆管炎(primary biliary cholangitis,PBC)数据为例,通过构建随机生存森林模型来探究D-青霉胺(DPCA)治疗对于原发性胆汁性胆管炎生存的影响[4]。同时,也探讨其他主要临床指标是否也对PBC的生存有影响。上述数据均被收录在R“survival”库中,具体的R语句见附件1。
原始数据共有总计418例患有PBC的研究对象,包含20个特征变量。为使原始数据满足分析函数的要求,我们对变量类型进行了简单处理,结果如表1所示。逻辑型数据是R语言里的基本数据类型之一,用来储存二元名义变量,只有表示“真”(true)和“假”(false)两种数值。因子型(factor)则是R语言中用来储存分类变量的数据类型。其中,时间(time)的单位为生存天数,在此换算为生存年数。由于只考虑单次复发的情况,原始数据“status”变量中的事件重新分组为“0”(删失)和“1”(事件)。

1974—1984年美国梅奥诊所原发性胆汁性胆管炎数据的变量特征
1974—1984年美国梅奥诊所原发性胆汁性胆管炎数据的变量特征
| 变量 | 赋值情况 | 数据类型 |
|---|---|---|
| id | 样本编号 | 数值型(numeric) |
| treatment | 是否应用D-青霉胺 (D-青霉胺;安慰剂) | 因子型(factor) |
| years | 生存时长(年数) | 数值型(numeric) |
| status | 删失状态(0=删失;1=死亡) | 数值型(numeric) |
| age | 年龄(年) | 数值型(numeric) |
| sex | 性别 (f =女;m=男) | 逻辑型(logic) |
| ascites | 腹水(1=有;0=无) | 逻辑型(logic) |
| hepato | 肝癌(1=有;0=无) | 逻辑型(logic) |
| spiders | 蜘蛛痣(1=有;0=无) | 逻辑型(logic) |
| edema | 浮肿程度(1.0、0.5、0.0) | 数值型(numeric) |
| bili | 胆红素含量(mg/dl) | 数值型(numeric) |
| chol | 胆固醇含量(mg/dl) | 数值型(numeric) |
| albumin | 白蛋白含量(gm/dl) | 数值型(numeric) |
| copper | 铜含量(μg/day) | 数值型(numeric) |
| alk.phos | 碱性磷酸酶含量(U/L) | 数值型(numeric) |
| ast | 天冬氨酸转氨酶量(U/ml) | 数值型(numeric) |
| trig | 甘油三酯含量(mg/dl) | 数值型(numeric) |
| platelet | 血小板(ml/1 000) | 数值型(numeric) |
| protime | 凝血酶原时间(s) | 数值型(numeric) |
| stage | 肿瘤阶段(1~4) | 因子型(factor) |
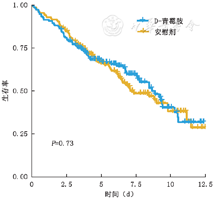
用乘积极限法来评估主要暴露因素(DPCA治疗)对结局事件(死亡)的影响。首先使用filter函数筛选出部分临床数据作为训练集,筛选比例视样本量和具体情况而定,并将剩余数据作为测试集,然后使用gg_survival函数建立“生存对象”(survival object)。gg_survival函数包含以下几个参数:interval,censor,by和data。Interval为生存时间变量,censor为生存事件变量,by为分层变量,data为输入数据。建立生存对象之后,使用ggsurvplot绘制其生存函数图。从图1的P值可知,D-青霉胺的使用对PBC患者的生存时间并无显著影响。

通过rfsrc函数构建随机生存森林模型,以构建各因素对PBC生存影响的预测模型。rfsrc是“randomForestSRC”库中最核心的函数,在filter函数筛选的测试集数据基础上,可通过调整多个参数以及袋内数据和袋外数据的构成比例,对模型进行精细的调整。例如,此函数可以用来进行多种随机森林模型的构建,也可以通过参数再次提高函数的运行速度。运行完毕后,可以通过rfsrc.print来查看模型构建情况。如表2所示,模型共生成了1 000个二元生存树,平均每个生存树有15个终端节点,模型基于袋外数据验证预测模型生存结局的错误率为18.2%。

rfsrc.print程序的输出结果
rfsrc.print程序的输出结果
| 变量 | 输出值 |
|---|---|
| Sample size | 312 |
| Number of deaths | 144 |
| Was data imputed | yes |
| Number of trees | 1 000 |
| Forest terminal node size | 15 |
| Average no. of terminal nodes | 15.768 |
| No. of variables tried at each split | 5 |
| Total no. of variables | 18 |
| Resampling used to grow trees | swor |
| Resample size used to grow trees | 197 |
| Analysis | RSF |
| Family | surv |
| Splitting rule | logrank*random* |
| Number of random split points | 10 |
| Error rate | 18.2% |
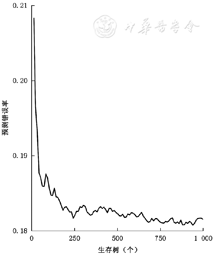
可视化袋外数据预测错误率的变化情况可以提供更直观的信息,有助于对模型进行调试。图2说明随着生存树数量的增加,其预测错误率明显降低;当生存树增加到一定数量后,错误率曲线趋于平稳(18.2%)。

进一步将上文构建的随机生存森林模型应用于filter函数生成的测试集,运用predict.rfsrc函数进行对测试集生存结局的预测,错误率为19.5% (表3)。而如若将该模型对训练集数据进行生存结局的预测,错误率为12.3%(表4)。

随机生存森林模型对测试集的预测结果
随机生存森林模型对测试集的预测结果
| 变量 | 预测值 |
|---|---|
| Sample size of test (predict) data | 106 |
| Number of deaths in test data | 42 |
| Was test data imputed | yes |
| Number of grow trees | 1 000 |
| Average no. of terminal nodes | 15.768 |
| Total no. of grow variables | 18 |
| Resampling used to grow trees | swor |
| Resample size used to grow trees | 67 |
| Analysis | RSF |
| Family | surv |
| Test set error rate | 19.51% |
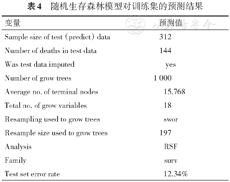
随机生存森林模型对训练集的预测结果
随机生存森林模型对训练集的预测结果
| 变量 | 预测值 |
|---|---|
| Sample size of test (predict) data | 312 |
| Number of deaths in test data | 144 |
| Was test data imputed | yes |
| Number of grow trees | 1 000 |
| Average no. of terminal nodes | 15.768 |
| Total no. of grow variables | 18 |
| Resampling used to grow trees | swor |
| Resample size used to grow trees | 197 |
| Analysis | RSF |
| Family | surv |
| Test set error rate | 12.34% |
在机器学习基础上进行变量筛选,可更好地理解变量重要性,而VIMP法和最小深度法是随机生存森林模型中常用的变量筛选方法。变量VIMP值小于0说明该变量降低了预测的准确性,而当VIMP值大于0时说明该变量提高了预测的准确性。最小深度法则通过计算运行到最终节点时的最小深度来给出各变量对于结局事件的重要性。
图3为综合两种方法的散点图,其中,蓝色点代表VIMP值大于0,红色则代表VIMP值小于0;在红色对角虚线上的点代表两种方法对该变量的排名相同,高于对角虚线的点代表其VIMP排名更高,低于对角虚线的点则代表其最小深度排名更高。
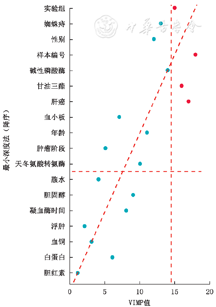

注:蓝色点代表VIMP值大于0,红色则代表VIMP值小于0
可以观察到,两种方法在最重要变量的选择结果基本相同,均为胆红素含量(bili),虽然对其他变量的排名不同,但是差异较小。综合两种方法变量选择结果,胆红素含量、铜含量、白蛋白含量、腹水情况、凝血酶原时间、胆固醇含量和浮肿情况为同时满足两种方法条件的重要变量,对于PBC患者的生存结局有着重要影响。无论采用何种方法,DPCA治疗均不是决定PBC患者生存结局的重要变量。
自20世纪80年代以来,以Cox比例风险回归模型为代表的传统生存分析模型为临床试验等医学研究做出了巨大的贡献[5, 6]。相较于Cox比例风险回归模型等传统生存分析方法,随机生存森林模型的预测准确度至少等同或优于传统生存分析方法。随机生存森林模型的优势体现在它不受比例风险假定、对数线性假定等条件的约束[2]。同时,随机生存森林具备一般随机森林的优点,能够通过两个随机采样的过程来防止其算法的过度拟合问题[7]。除此之外,随机生存森林还能够对高维数据进行生存分析和变量筛选,也能够应用于对竞争风险(competing risk)的分析[8, 9]。因而,随机生存森林模型有着更为广泛的研究空间。
“randomForestSRC”库功能十分强大,该库的官方操作指南也十分详细,并提供了相应的应用实例。本文以该库自带的临床数据为例介绍了随机生存森林的数学原理并运用随机生存森林进行生存分析的基本流程。需要强调指出的是,尽管多篇文献表明随机生存森林模型优于或至少等同于传统生存模型的准确度,但是在数据符合传统生存分析要求时,传统生存分析方法仍不可或缺。作为新兴方法,随机生存森林也存在缺陷:易受离群值的影响。在分析具有离群值数据时,预测准确度会稍劣于传统生存分析方法[10]。Cox比例风险回归模型对于生存数据的分析不仅仅用于预测,还可以较为便捷地给出各变量与生存结局的关系,所以应该和传统生存分析相结合应用,随机生存森林模型并不能完全替代传统生存分析模型。
对随机生存森林模型的应用,应注意以下几点:第一,在构建随机生存森林模型时,需要格外注意原始数据的数据类型,必要时提前对原始数据进行处理,替换成因子(factor)等类型。例如,示例中是否应用DPCA这一变量(treatment)即需转换为因子型,否则无法运行。第二,rfsrc函数包含了许多参数,使用时提前注意其参数的默认选项,避免错误。第三,本研究中使用的“na.impute”选项是使用函数默认的填充方法对缺失值进行填充,具体方法为利用随机森林模型寻找缺失值的邻近值。若对缺失值的填补有特殊要求,可提前进行相应的填补。需要注意的是,大量存在的缺失值会降低模型的准确度。第四,虽然大多数情况下,VIMP法和最小深度法所筛选出的变量类似,仍建议结合两种方法来进行变量的选择。第五,与传统的Cox比例风险回归模型不同,随机生存森林模型在建立的时候已经自动地对各变量进行调整,所以不需要再调整协变量带来的影响。


1. 随机生存森林模型的基本单位是:
A. 二元生存树
B. 二元森林
C. 二元决策树
D. 决策森林
2. 下列对随机生存森林描述正确的是:
A. 构建随机生存森林模型时,应将原始数据进行处理,不需要将离散变量转换成因子型数据。
B. 随机生存森林可以通过选择不同的变量组合来提高其预测准确率。
C. 随机生存森林模型通过使用“自助法”(Bootstrap)将原始数据以有放回的形式随机抽取样本,建立样本子集。
D. 随机生存森林模型可以完全替代其他传统生存分析模型。
3. 下列对VIMP值的理解正确的是:
A. VIMP的绝对值越大,其变量重要程度越高。
B. VIMP值不能为负。
C. VIMP越大就意味着此变量对于模型的准确度影响越大,此变量的重要性就越高。
D. 可以通过VIMP筛选变量来提高模型的预测准确率。
4. 下列对最小深度法描述正确的是:
A. 可以通过最小深度法筛选出的变量来提高模型的预测准确率。
B. 因为最小深度发得出的变量比VIMP更有参考价值,所以可以仅参考通过最小深度法筛选出的变量。
C. 最小深度值可以为负数。
D. 最小深度值越小,其变量重要程度越高。
5. 随机生存森林的局限性是:
A. 不能描述重要变量与结局变量之间的准确关系。
B. 模型预测准确度经常劣于传统生存分析模型。
C. 和其他机器学习算法一样,需要特别预防过拟合问题。
D. 使用该模型时,需要考虑数据是否满足比例风险等假设。



























