人冠状病毒OC43(human coronavirus OC43,HCoV-OC43)与SARS-CoV-2(severe acute respiratory syndrome coronavirus 2)同属于β冠状病毒属,自1967年发现以来在人群中持续流行,现已成为常见的季节性呼吸道病毒之一。致病率和致死率更高的SARS-CoV-2在2019年底出现,后又伴随多种变异株的出现,其传播和感染能力不断增强。HCoV-OC43与SARS-CoV-2在基因组结构和功能、物种进化、流行特点及临床表现上可能具有一定的相似性。本综述对HCoV-OC43和SARS-CoV-2的流行病学、基因组学、种系进化等方面进行分析,有助于了解这两种病毒的关联与差异,为认识HCoV-OC43存在的潜在威胁提供参考。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
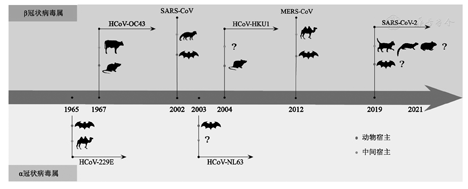
自2019年12月底SARS-CoV-2疫情出现,截至2021年6月,全球累计感染人数已经达到1.7亿,死亡病例数达到389万[1],SARS-CoV-2成为继1918年西班牙H1N1大流行之后对人类社会影响最严重的传染病[2]。SARS-CoV-2是一种冠状病毒,冠状病毒根据其基因组序列和血清学特征的不同,可分为α、β、γ、δ 4个种属[3]。目前包括SARS-CoV-2在内已经发现的7种人冠状病毒(human coronavirus, HCoVs)中,除HCoV-229E和HCoV-NL63属于α冠状病毒属外,HCoV-OC43、HCoV-HKU1、严重急性呼吸综合征冠状病毒(severe acute respiratory syndrome coronavirus, SARS-CoV)、中东呼吸综合征冠状病毒(Middle East respiratory syndrome coronavirus, MERS-CoV)和SARS-CoV-2均属于β冠状病毒属(图1)[4]。其中HCoV-OC43于1967年首次从患者的鼻咽分泌物中被分离[5],是现今常见的季节性冠状病毒之一[6]。由于HCoV-OC43在已知HCoVs中的人群检出率高但致病力较弱,过去常作为高致病性冠状病毒如SARS-CoV和MERS-CoV相关研究的模式病毒[7]。但伴随HCoV-OC43感染引发致命性肺炎和脑炎相关病例的出现,具有新基因组的HCoV-OC43临床分离株不断被鉴定[8],提示HCoV-OC43也可能存在潜在威胁。此外,高致病性HCoVs在近年来频繁出现,其中最新出现的SARS-CoV-2与以往SARS-CoV和MERS-CoV的短期暴发流行不同,多项研究认为SARS-CoV-2可能在大流行后演变为新的季节性冠状病毒[9]。因此,本文从流行病学、基因组学、种系进化、临床诊断与治疗等方面对HCoV-OC43与SARS-CoV-2的相似性进行综述,旨在了解两种病毒的关联与差异,为HCoV-OC43及其他动物源性冠状病毒存在的潜在威胁提供启示。
HCoV-OC43自1967年被发现以来在世界范围内广泛存在,现已成为常见的季节性冠状病毒之一,多在冬季、早春迎来流行高峰[11]。在各年龄组中由季节性冠状病毒引起的轻度自限性上呼吸道感染以HCoV-OC43检出率最高,特别是在儿童群体中[12]。虽然HCoV-OC43本身致病力较弱,但较HCoV-229E、HCoV-HKU1、HCoV-NL63更容易引起下呼吸道感染[13],常与其他呼吸道病毒如腺病毒、流感病毒等发生合并感染,特别是其神经侵袭性对免疫缺陷人群可造成致命性脑炎[14],在临床上仍具有一定的威胁性。有研究发现感染HCoV-OC43后血清中特异性抗体对同亚群的HCoV-HKU1感染具有一定的保护作用,抗体水平在一年后下降至感染前水平[15]。而首次感染HCoV-OC43多发生在两岁前[16],因此感染后产生的抗体使儿童对冠状病毒感染具有一定的免疫力,但由于抗体滴度下降和病毒变异等原因,个体可能会反复感染HCoV-OC43,并且再感染通常发生在上次感染后超过一年的时间[17],再感染时病毒载量多增高,导致其具有潜在暴发的威胁[18,19]。
相比之下,SARS-CoV-2疫情在冬季暴发后,迅速引起全球大流行,季节因素似乎对SARS-CoV-2的影响不大[20]。此外,SARS-CoV-2不仅具有较强的致病能力更容易引发下呼吸道感染,还表现出极强的传播性,感染者往往在症状出现之前就具有传染能力[21],常以嗅觉失灵作为COVID-19 (coronavirus disease 2019)的特征性表现之一[22]。研究发现新发HCoVs感染与较轻的COVID-19相关[23],患者中很少有幼儿、青少年或是症状较轻,感染后无症状患者的中位年龄是14岁,几乎一半以上病例发生在60岁以上的成年人中[24],HCoVs和SARS-CoV-2可能存在普遍的交叉反应性CD4+T细胞,表现出一种保护作用[25]。但与此同时,COVID-19患者的严重程度与血清中抗HCoV-OC43的S-IgG抗体水平呈正相关[26]。推测随着年龄的增加,成人体内针对HCoV-OC43 S蛋白的预存抗体增加,COVID-19患者血清中针对这两种病毒的低亲和力交叉反应性抗体可能形成免疫复合物,参与类似抗体依赖性增强反应[26]。个体在感染SARS-CoV-2后获得的保护力持续时间则与HCoV-OC43类似,抗体水平均可维持一年左右。但由于HCoV-OC43在人群中检出率高,血清抗体阳转率显著高于其他HCoVs,新生儿普遍能够携带针对HCoV-OC43的母体抗体[27],而孕期感染SARS-CoV-2的患者经胎盘转移特异性SARS-CoV-2抗体的效率则明显低于HCoV-OC43,新生儿通过母体获得针对SARS-CoV-2的免疫转移机制尚未建立[28,29]。并且多种变异毒株如Delta变异株的出现也导致SARS-CoV-2重复感染比例显著升高[30],说明两种病毒在保护性免疫的效率上可能存在差异。
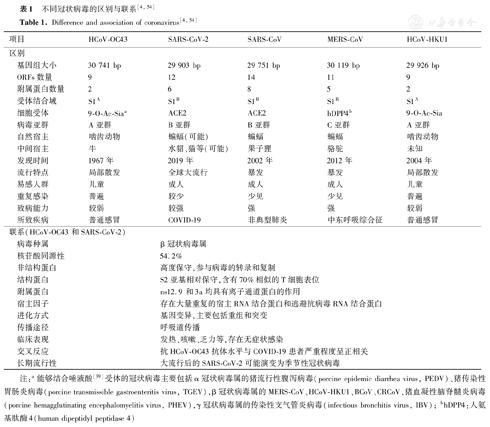
HCoV-OC43和SARS-CoV-2均属于β冠状病毒属,基因组RNA具有54.2%的核苷酸同源性[31],包含多个开放阅读框(open reading frames, ORFs),靠近5′端的ORF1a和ORF1b在两种冠状病毒中高度保守,占整个基因组大小约2/3,两ORF编码的多聚蛋白可以剪切为16个非结构蛋白,参与病毒的转录和复制(图2)。靠近3′端的ORFs在两者中则分别编码变异程度较大结构蛋白和附属蛋白,其中4种共有的结构蛋白包括刺突蛋白(spike protein, S)[32]、包膜蛋白(envelope protein, E)[33]、膜糖蛋白(membrane glycoprotein, M)[34]和核蛋白(nucleocapsid, N)[35],均在病毒复制过程中发挥关键作用。不参与复制的附属蛋白,在感染宿主的过程中发挥着特定的调控作用[36]。
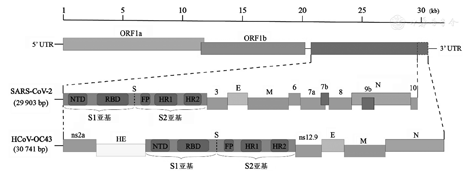
两种病毒在结构蛋白和附属蛋白上的差异较为显著。首先,HCoV-OC43含有额外的血凝素酯酶(hemagglutinin-esterase, HE),HE来源于C型流感病毒的HE融合蛋白与S蛋白共同位于病毒包膜表面形成大小不一的两种突起[37]。在适应人体组织的过程中HE发挥受体破坏酶的作用,介导S蛋白与受体决定因子结合缺失,从感染细胞表面释放病毒,扩大组织趋向性[38]。其次,两种病毒的S蛋白均可被弗林蛋白酶切割为S1和S2两种亚基,其中N端的S1亚基包括A、B、C、D四个结构域,HCoV-OC43和SARS-CoV-2的受体结合域分别位于结构域A(S1A)和B(S1B)[39,40]。HCoV-OC43利用S1A上的受体结合位点,在HE蛋白的辅助下结合宿主细胞表面唾液酸(9-O-acetylated sialic acid, 9-O-Ac-Sia)受体介导病毒黏附和进入[41,42],SARS-CoV-2则利用S1B上的受体结合域(receptor-binding domain, RBD)结合细胞受体血管紧张素转化酶2(angiotensin-converting enzyme 2, ACE2)黏附在感染细胞表面后,在S2亚基与跨膜蛋白酶丝氨酸2(transmembrane protease serine 2, TMPRSS2)的相互作用下进入胞内[43]。TMPRSS2与受体ACE2相关,在人呼吸道上皮细胞表面高表达,由于SARS-CoV-2对ACE2有更高的亲和力,因此人类呼吸道上皮细胞和病毒的质膜融合是感染的首选途径,以肺泡巨噬细胞、血管内皮细胞为主要靶细胞[44]。HCoV-OC43虽然也对TMPRSS2具有一定的敏感性,但对胞内组织蛋白酶不敏感,已经证明HCoV-OC43趋向感染表达N-乙酰基-9-O-乙酰神经氨酸(N-acetyl-9-O-acetylneuraminic acid,Neu5,9Ac2)的纤毛细胞[45]。其次虽然多种病毒均可结合唾液酸如牛冠状病毒(bovine coronavirus,BCoV),但由于HCoV-OC43对人类呼吸道α-2,6-糖苷键连接的唾液酸受体(α-2,6-sialic acids,α-2,6-SAs)的结合嗜性,HCoV-OC43对α-2,6-SAs的亲和力高于BCoV[39,46],该特性不仅增加病毒的存活机会还扩大了感染范围。除结构蛋白,两种病毒附属蛋白的数量、大小、位置也各不相同,HCoV-OC43主要表达的两种附属蛋白ns2a和ns12.9能够抑制宿主抗病毒反应元件,促进病毒复制[47,48]。相比之下,SARS-CoV-2编码的9种附属蛋白(3a, 3b, 6, 7a, 7b, 8, 9b, 9c, 10)则能够发挥更多的调控作用影响病毒的毒力和复制能力等[49]。
尽管HCoV-OC43和SARS-CoV-2在整体结构上存在较大差异,但仍具有一定的相似性。两种病毒的S和E基因间存在保守的附属蛋白基因,在HCoV-OC43中编码ns12.9,而在SARS-CoV-2中编码3a,均具有离子通道蛋白的作用,促进病毒粒子的产生和释放[48]。在易发生变异的结构蛋白中,S2亚基相对保守。HCoV-OC43 S2亚基上的T细胞表位与SARS-CoV-2最高相似性为70%[50],其S蛋白能够激活SARS-CoV-2感染后的记忆T细胞应答[51]。感染SARS-CoV-2的恢复期患者血清中记忆B细胞产生的IgG抗体能够与HCoV-OC43的S2亚基结合[52],反映两种病毒S2亚基免疫原性相近,具有一定的交叉反应性。而造成病毒持续感染的关键环节在于病毒与宿主因子的相互作用,包括利用宿主RNA结合蛋白和逃避抗病毒RNA结合蛋白的能力,在已经鉴定出的109个直接与SARS-CoV-2的RNA结合的宿主因子中,发现有107个能够在HCoV-OC43相应的宿主因子中发现,说明两者的RNA与宿主因子间的相互作用在进化上可能存在相似性[31]。
通过冠状病毒的系统发生学(phylogenetics)发现β冠状病毒属可能源自共同的祖先,早期经历趋异进化形成5种亚群[4],包括Embecovirus(A)、Sarbecovirus(B)、Merbecovirus(C)、Nobecovirus(D)和Hibecovirus(E),其中HCoV-OC43和SARS-CoV-2分别来自A亚群和B亚群,基因组RNA较大(约30 kb),易发生高频重组和缺失,具有遗传多样性。
现有研究普遍认为HCoV-OC43来源于啮齿动物,于19世纪末跨越物种屏障在人类宿主中流行[10]。进化过程中,HCoV-OC43的HE为适应人类宿主自发丧失其凝集素活性,通过HE和S蛋白的趋同进化,使两者在受体破坏和受体结合功能上相互依赖维持平衡,确保病毒的有效传播[55]。因此经过长期的趋同进化,HCoV-OC43形成与流感病毒相似的病毒-唾液酸多糖受体使用偏性[37]。2016年野生大猩猩中HCoV-OC43的暴发也说明其存在和流感病毒相似的宿主转换[56]。除宿主转移的广泛性外,基因型转移也是HCoV-OC43维持流行的另一种适应机制[57],目前至少存在8种重组基因型(A, B, C, D, E, F, G, H),以2004年出现的D型为优势流行株[58]。对当前处于大流行的SARS-CoV-2开展溯源研究,发现在已有的蝙蝠冠状病毒序列信息中的RaTG13(horse-shoe bat coronavirus)与SARS-CoV-2有96%的同源性,整体密码子使用性相似[59],是已知与SARS-CoV-2基因组序列最接近的蝙蝠冠状病毒,因此推断SARS-CoV-2可能来源于蝙蝠[60]。此外,猫、雪貂、仓鼠、水貂、浣熊、猪等动物均能够感染SARS-CoV-2[61],甚至在水貂和人之间存在互相传播感染的病例[62]。但与SARS-CoV-2不同,牛是HCoV-OC43而非SARS-CoV-2的中间宿主,SARS-CoV-2对牛自然感染少见,乳牛暴露于SARS-CoV-2后病毒复制有限且被感染的和同养的犊牛中没有观察到病毒传播[63]。
宿主和病毒相互作用下的自然选择是变异株出现和在世界范围内传播的源动力,HCoV-OC43在自然选择作用下,始终维持S蛋白N端结构域关键糖基结合位点的保守性,借助其周围的点突变不断适应宿主维持流行,但至今尚未发现HCoV-OC43的感染能力发生明显改变。而当前相继出现的多种SARS-CoV-2变异株,如Alpha变异株(B.1.1.7)、Beta变异株(B.1.351)、Gamma变异株(P.1)、Delta变异株(B.1.617.2)等,病毒的感染和传播能力却明显增强。研究推测与HCoV-OC43结合位点的相对保守不同,在SARS-CoV-2进化过程中,其S蛋白的突变可能驱动RBD与ACE2结合能力增加,从而导致SARS-CoV-2变异株的复制能力和传染性增强[64]。
HCoV-OC43感染与SARS-CoV-2感染初期的症状相似,主要均表现为发热、咳嗽、乏力等感冒症状,无症状感染者多见于儿童和青少年,及时控制疫情并采取有效的治疗措施需要对两种病毒进行鉴别诊断,建立完善的临床检验策略。首先,病原学检查是诊断金标准,当前SARS-CoV-2的临床筛查和确诊广泛采用基于实时荧光定量RT-PCR核酸检测,核酸检测试剂盒中的引物和探针大多针对ORF1ab基因、包膜蛋白基因及核衣壳蛋白基因中的2种或3种所设计[65]。尤其是近年来以各HCoVs保守区N为靶基因设计特异性引物而建立的多重RT-PCR方法,其可行性也得到验证,该检测方法能够实现对同一标本进行多种人冠状病毒的同时检测[66],方便快捷更适用于临床实验室。但核酸检测较血清学检测对标本、操作条件等要求高,虽然发病初期体内抗体阳性率较低,但针对恢复期患者或核酸检测结果阴性的疑似病例,血清中抗病毒IgM/IgG抗体滴度仍具有一定的诊断意义[67]。其次,临床上综合诊断还应辅以影像学检查等手段,胸片显示广泛或多病灶磨玻璃浑浊伴肺实质性病变常提示高致病性冠状病毒感染,而HCoV-OC43一般表现为多个结节性和楔形浑浊[68]。
明确感染的冠状病毒类型后应采取相应的治疗措施,但由于冠状病毒的高度变异性,尚无特异性或广谱抗HCoVs药物可供临床使用,因此有必要基于现有HCoVs寻找共同或保守的治疗靶点。研究发现HCoV-OC43-HR2P对所有HCoVs的HR1Ps表现出广谱结合活性,并对HCoVs介导的细胞-细胞融合具有抑制活性[8]。此外以HCoV-OC43为研究模型,也发现激活干扰素基因刺激因子(stimulator of interferon genes,STING)信号能够有效阻断HCoV-OC43和SARS-CoV-2的感染,这些位点均是开发广谱冠状病毒抑制药物的潜在靶点。但目前临床上的抗病毒治疗仍主要依据临床表现采用相应的治疗措施以达到治疗或减缓病情的发展[69]。HCoV-OC43等季节性冠状病毒引起的自限性轻度感染和SARS-CoV-2轻症感染采用一般疗法即可痊愈。结合临床观察,COVID-19的轻症患者服用寒湿疫方、宣肺败毒方等中药也可起到一定的治疗效果。但当疾病发展至重型、危重型应在治疗基础疾病的同时积极防治并发症预防继发感染,临床上多给予广谱抗病毒药物进行抗病毒治疗、免疫治疗、有效氧疗等治疗措施,联合使用中药注射剂和中药汤剂也有积极疗效[70,71]。
从1965年第一个人冠状病毒HCoV-229E发现至今,包括HCoV-OC43、HCoV-NL63和HCoV-HKU1通常感染人类上呼吸道引起普通感冒,但在儿童、老年人和免疫功能低下的人群中也会导致严重甚至致命性疾病,表明这些常见HCoVs可能会对人类构成威胁。因此本文对HCoV-OC43和SARS-CoV-2这两种流行率较高的HCoVs在流行特点、基因组结构功能、种系进化、临床诊断与治疗方面进行比较分析(表1)。两种病毒均易发生重组变异,具有广泛的宿主转移性,并在宿主中表现出相似的免疫逃逸机制和感染初期的临床表现,感染后对肺组织、胃肠道表现出趋向性,诱导产生的抗体和T细胞应答具有交叉反应。尽管目前SARS-CoV-2的致病性显著高于HCoV-OC43,但两者感染后普遍存在无症状感染现象。研究认为HCoV-OC43由于复制过程相对缓慢难以触发固有免疫应答[45],SARS-CoV-2则可以逃避呼吸道上皮细胞分泌的干扰素,导致免疫应答延迟[72]。而在人支气管上皮细胞中,由于SARS-CoV-2与ACE2受体亲和力增强,SARS-CoV-2更易激活巨噬细胞等炎性细胞引发强烈的趋化和炎症反应[45]。这种抗病毒反应降低和促炎状态的升高被认为是导致COVID-19的原因之一。因此随着HCoV-OC43唾液酸受体在病毒刺突蛋白介导的细胞融合过程中,导致病毒传播感染中的作用被进一步揭示[40],在将来的进化过程中HCoV-OC43可能会增强其致病力进而引发与SARS-CoV-2相似的大流行。
| 项目 | HCoV-OC43 | SARS-CoV-2 | SARS-CoV | MERS-CoV | HCoV-HKU1 | |
|---|---|---|---|---|---|---|
| 区别 | ||||||
| 基因组大小 | 30 741 bp | 29 903 bp | 29 751 bp | 30 119 bp | 29 926 bp | |
| ORFs数量 | 9 | 12 | 14 | 11 | 9 | |
| 附属蛋白数量 | 2 | 6 | 8 | 5 | 2 | |
| 受体结合域 | S1A | S1B | S1B | S1B | S1A | |
| 细胞受体 | 9-O-Ac-Siaa | ACE2 | ACE2 | hDPP4b | 9-O-Ac-Sia | |
| 病毒亚群 | A亚群 | B亚群 | B亚群 | C亚群 | A亚群 | |
| 自然宿主 | 啮齿动物 | 蝙蝠(可能) | 蝙蝠 | 蝙蝠 | 啮齿动物 | |
| 中间宿主 | 牛 | 水貂、猫等(可能) | 果子狸 | 骆驼 | 未知 | |
| 发现时间 | 1967年 | 2019年 | 2002年 | 2012年 | 2004年 | |
| 流行特点 | 局部散发 | 全球大流行 | 暴发 | 暴发 | 局部散发 | |
| 易感人群 | 儿童 | 成人 | 成人 | 成人 | 儿童 | |
| 重复感染 | 普遍 | 较少 | 少见 | 少见 | 普遍 | |
| 致病能力 | 较弱 | 较强 | 强 | 强 | 较弱 | |
| 所致疾病 | 普通感冒 | COVID-19 | 非典型肺炎 | 中东呼吸综合征 | 普通感冒 | |
| 联系(HCoV-OC43和SARS-CoV-2) | ||||||
| 病毒种属 | β冠状病毒属 | |||||
| 核苷酸同源性 | 54.2% | |||||
| 非结构蛋白 | 高度保守,参与病毒的转录和复制 | |||||
| 结构蛋白 | S2亚基相对保守,含有70%相似的T细胞表位 | |||||
| 附属蛋白 | ns12.9和3a均具有离子通道蛋白的作用 | |||||
| 宿主因子 | 存在大量重复的宿主RNA结合蛋白和逃避抗病毒RNA结合蛋白 | |||||
| 进化方式 | 基因变异,主要包括重组和突变 | |||||
| 传播途径 | 呼吸道传播 | |||||
| 临床表现 | 发热、咳嗽、乏力等,存在无症状感染 | |||||
| 交叉反应 | 抗HCoV-OC43抗体水平与COVID-19患者严重程度呈正相关 | |||||
| 长期流行性 | 大流行后的SARS-CoV-2可能演变为季节性冠状病毒 | |||||
注:a能够结合唾液酸[39]受体的冠状病毒主要包括α冠状病毒属的猪流行性腹泻病毒(porcine epidemic diarrhea virus, PEDV)、猪传染性胃肠炎病毒(porcine transmissible gastroenteritis virus, TGEV),β冠状病毒属的MERS-CoV、HCoV-HKU1、BCoV、CRCoV、猪血凝性脑脊髓炎病毒(porcine hemagglutinating encephalomyelitis virus, PHEV),γ冠状病毒属的传染性支气管炎病毒(infectious bronchitis virus, IBV); bhDPP4:人氨基肽酶4(human dipeptidyl peptidase 4)
除已发现的HCoVs外,还需警惕动物源性冠状病毒的溢出,例如与HCoV-OC43受体类似的犬呼吸道冠状病毒(canine respiratory coronavirus, CRCoV)和牛冠状病毒,3种病毒间S蛋白同源性高于95%,均能够结合唾液酸可能具有相同的受体特异性[42,46]。研究指出BCoV与HCoV-OC43具有96%核苷酸同源性,BCoV在冬季犊牛中的血清流行率为43%~65%,与青少年冬季原发性HCoV-OC43感染率相当,推测两者可能并没有真正分离,BCoV在进化过程中可能发生宿主转移[73]。除β冠状病毒属外,当前α冠状病毒属中一种全新的犬冠状病毒(canine coronavirus, CCoV)也在有野生动物接触史的肺炎患者中被发现[74],再次表明人畜跨种传播在冠状病毒中并不罕见。因此,我们不仅要关注HCoVs,也要重视对其他动物源性冠状病毒的监测,以提高对新兴冠状病毒的预警性和防控能力。
所有作者均声明不存在利益冲突