
建立新型冠状病毒(2019 novel coronavirus,2019-nCoV)的细胞培养和全基因组测序方法,了解2019-nCoV基因变异情况。
选取部分2019-nCoV感染的肺炎确诊病例的鼻、咽拭子标本接种Vero-E6细胞培养管进行分离培养,逐日观察病变,对病变的细胞开展荧光定量RT-PCR检测确认病毒分离结果;选取部分分离到的新型冠状病毒进行10倍稀释后接种细胞测定病毒滴度。采用2019-nCoV全基因组捕获技术及二代测序方法对部分分离到的毒株进行全基因组测序,对获得的序列进行序列比对和进化分析,分析其变异位点。
在生物安全三级实验室建立2019-nCoV在Vero-E6细胞系中的病毒分离方法,共对150份2019-nCoV阳性病例鼻、咽拭子标本开展病毒分离,共分离到22株2019-nCoV毒株。对其中的18株2019-nCoV开展全基因组测序,获得的基因组序列全长约为29 600 bp,基因同源性在99.9%以上,共有43个核苷酸和24个氨基酸位点发生了变异。基因进化分析显示,分离到的病毒分为L型和S型分支,在L型分支中,又包含了欧洲家系I型和II.1型分支。
通过Vero-E6细胞成功分离培养了2019-nCoV,并分析了其基因变异规律,为病例的溯源、药物筛选、生物学特性研究等工作提供了基础。同时,持续开展2019-nCoV分离和全基因组测序分析对后续的新冠肺炎防控及变异毒株的监测也至关重要。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
新型冠状病毒肺炎是由新型冠状病毒(2019 novel coronavirus, 2019-nCoV)引起的一种新型急性呼吸道传染病[1,2]。疫情自2019年12月出现以来,目前仍处于大流行阶段[2]。截止到2021年4月20日,全球已经报告新型冠状病毒感染的肺炎病例(简称"新冠肺炎病例")1.4亿例,死亡300多万例[3]。
2019-nCoV属于β属冠状病毒,有包膜,颗粒呈圆形或椭圆形,常为多形性,直径60~140 nm,其基因特征与SARS-CoV和MERS-CoV有明显区别。病毒的基因组是一条单股多链RNA,长度为29 903个核苷酸,具备典型的冠状病毒属结构,包括5′端非编码区、复制酶复合体(open reading frame 1ab,ORF1ab)基因、刺突糖蛋白(spike,S)基因、包膜蛋白(envelope,E)基因、膜蛋白(Membrane,M)基因、核衣壳蛋白(Nucleocapsid,N)基因、含有poly(A)尾的3′UTR和几个未识别的非结构ORF[4]。
近年来,随着分子生物学技术的不断发展,二、三代基因组测序技术不断成熟,利用基因组学技术开展区域性或全球性流行病的研究已经迅速普及。同时,全基因组测序在病原体鉴定上具有重要的作用,科学家通过测得的全基因组序列,初步判定2019-nCoV来源于蝙蝠和穿山甲。同时,随着疫情的不断扩散,病例不断增多,测得的全基因组序列不断增加,通过对获得的基因组进行进化分析,根据基因组序列的单核苷酸多态性将2019-nCoV分成不同的基因型,如中国分型法、Pangolin分型法、GISAID分型法、Nextstrain分型法[5]。同时,科学家可根据全基因组序列找到相应的蛋白质变异位点,了解病毒变异位点对病毒传播、致病力的影响,如S蛋白的D614G变异位点,能增强病毒的传播能力[6]。
山东省自2020年1月21日报道首例新冠肺炎病例后,截至2021年4月20日共报告确诊病例876例,其中包括本土病例、境外输入性病例和青岛暴发疫情中的本土感染病例[7]。在本研究对150多例报告的新冠肺炎病例的鼻、咽拭子标本进行病毒分离,对分离到的病毒开展全基因组测序和基因进化分析,了解其变异情况,并为后续药物筛选、生物学特性研究等提供基础。
本研究所涉及到的标本均来自于各市送检的新冠肺炎确诊病例的鼻、咽拭子标本。
Vero-E6细胞为山东省疾病预防控制中心保存,MEM培养液购买日本日水制药株式会社,胎牛血清、谷氨酰胺以及双抗均购自GIBCO公司。
将新冠肺炎确诊病例的鼻、咽拭子标本用10%的双抗进行处理,在4 ℃冰箱静置4 h。在生物安全三级实验室(BSL-3)吸取处理好的200 μl鼻、咽拭子标本接种到PBS清洗2遍的单层Vero-E6细胞管内,在37 ℃、5% CO2培养箱中吸附1 h。吸附完成后,吸出吸附液,PBS清洗2遍,加入1 ml含2%胎牛血清的MEM病毒生长液,在37 ℃、5% CO2培养箱培养。同时设置阴性细胞对照管。每日在倒置显微镜下观察细胞病变(cytopathic effect,CPE)连续观察6 d,出现病变的细胞培养管采用Real-time RT-PCR进行鉴定。所有接种标本均盲传三代,盲传三代没有出现病变的标本判定为阴性。
鉴定核酸提取在BSL-3实验室开展。吸取200 μl细胞上清液加入到裂解液中,病毒RNA提取按照QIAGEN公司的RNeasy Mini Kit(货号:74104)说明书操作,real-time RT-PCR检测按照上海伯杰生物科技有限公司生产的新型冠状病毒荧光定量RT-PCR检测试剂盒说明书操作,主要检测其ORF1a/b基因和N基因。
挑取部分病毒的第二、第三代病毒液100 μl进行10倍系列稀释,直到106倍,分别记作10-1~10-6。稀释完成后依次接种到铺满单层Vero-E6细胞的96孔板内,每个稀释度接种8个复孔,每孔接种100 μl;另设2组正常细胞对照孔,置于37 ℃、5%CO2的培养箱中吸附1 h。吸附完成后,弃去上清液,加入200 μl含有2%胎牛血清的MEM病毒生长液继续培养,每日使用倒置显微镜下观察病变,连续观察4天,记录各孔的CPE情况,按照Reed-Muench法计算50%细胞的病变浓度(TCID50)。
选取部分鉴定为2019-nCoV的毒株,在BSL-3实验室提取RNA,按照北京微未来生物科技有限公司生产的ULSEN超灵敏度2019-nCoV全基因组捕获试剂盒(V-090418-1)说明书将提取的RNA进行全基因组特异性扩增。利用QIAquick PCR purification试剂盒(Qiagen,德国)对PCR产物进行纯化。按照NexteraXT DNA Sample Preparation Kit试剂盒(Illumina,美国)说明书构建DNA测序文库。文库构建完成后,采用Illumina公司的Miseq二代测序仪进行全基因组测序。测序完成后,使用北京微未来生物科技有限公司提供的序列拼接软件对获得的下机序列进行拼接。采用DNAMAN、MEGA 6.0等分子生物学软件对拼接的2019-nCoV全基因组序列进行序列比对和同源性分析,采用中国分型法、pangolin分型法、基于邻接法进行系统发育分析并构建进化树,bootstrap法检验重复值设置为1 000,以2019-nCoV/Wuhan/WIV04/2019基因组作为参考序列。根据全基因组序列数据,推导其编码的氨基酸序列,并比较氨基酸序列的变异情况。
共对150余例新冠肺炎病例的鼻、咽拭子标本进行病毒分离,通过病变观察和荧光定量RT-PCR验证,从Vero-E6细胞系上成功分离到2019-nCoV 22株,经过盲传三代,均能稳定的进行增值,挑选的3株病毒的TCID50为1×103.95/100 μl~1×104.75/100 μl。其CPE结果见图1。从标本采集时间来看,2020年1月份11株、2月份6株、3月份2株、10月份3株。从病例感染来源来看,早期的本土病例17株、境外输入性病例2例(分别来自于美国和意大利),2020年10月青岛暴发疫情的本土感染病例3例。


A.Vero-E6细胞对照;B.48小时的细胞病变;C.96小时的细胞病变;D.完全病变
A. Vero-E6 cell control;B.48 h CPE; C.96 h CPE; D.completely change of Vero-E6 cells
采用北京微未来生物科技有限公司的2019-nCoV全基因组捕获法试剂盒及Nextera XT DNA Sample Preparation Kit试剂盒构建文库并进行全基因组测序,对获得的下机数据进行拼接,共获得18株2019-nCoV的全基因组序列,全基因组全长约为29 600 bp左右,其同源性为99.9~100%。
与参考序列比较,分离到的2019-nCoV共有43个核苷酸发生了变异,分布于9个开放阅读框。按照核苷酸突变突变位点由多到少依次为:ORF1a区21个、S蛋白区7个、ORF1b区5个、N蛋白区3个、ORF3a区3个、M蛋白区1个、ORF8区1个、ORF7区1个和5′UTR区1个,关键位点变化见表1。
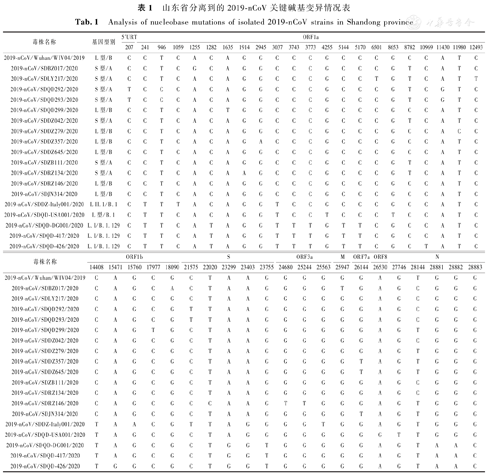
山东省分离到的2019-nCoV关键碱基变异情况表
Analysis of nucleobase mutations of isolated 2019-nCoV strains in Shandong province
山东省分离到的2019-nCoV关键碱基变异情况表
Analysis of nucleobase mutations of isolated 2019-nCoV strains in Shandong province
| 毒株名称 | 基因型别 | 5′URT | ORF1a | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 207 | 241 | 946 | 1059 | 1255 | 1282 | 1635 | 1914 | 2945 | 3037 | 3743 | 3773 | 4255 | 5144 | 5170 | 6501 | 8653 | 8782 | 10969 | 11430 | 11980 | 12493 | ||
| 2019-nCoV/Wuhan/WIV04/2019 | L型/B | C | C | T | C | A | C | A | G | G | C | C | C | G | C | C | C | G | C | C | A | T | C |
| 2019-nCoV/SDBZ017/2020 | S型/A | C | C | T | C | G | C | A | G | G | C | C | C | G | C | C | C | G | T | C | A | T | C |
| 2019-nCoV/SDLY217/2020 | S型/A | C | C | T | C | A | C | A | G | G | C | C | C | G | C | C | T | G | T | C | A | T | T |
| 2019-nCoV/SDQD292/2020 | S型/A | T | C | C | C | A | C | A | G | G | C | C | C | G | C | C | C | G | T | C | G | T | C |
| 2019-nCoV/SDQD293/2020 | S型/A | T | C | C | C | A | C | A | G | G | C | C | C | G | C | C | C | G | T | C | G | T | C |
| 2019-nCoV/SDQD299/2020 | L型/B | C | C | T | C | A | C | T | G | G | C | C | C | G | C | C | C | G | C | C | A | T | C |
| 2019-nCoV/SDDZ042/2020 | S型/A | C | C | T | C | A | C | A | G | G | C | C | C | G | C | C | C | G | T | C | A | T | C |
| 2019-nCoV/SDDZ279/2020 | L型/B | C | C | T | C | A | C | A | G | G | C | C | C | G | C | C | C | G | C | C | A | C | C |
| 2019-nCoV/SDDZ357/2020 | L型/B | C | C | T | C | A | C | A | G | A | C | C | C | G | C | C | C | G | C | C | A | T | C |
| 2019-nCoV/SDDZ645/2020 | L型/B | C | C | T | C | A | C | A | G | G | C | C | C | G | C | C | C | G | C | C | A | T | C |
| 2019-nCoV/SDZB111/2020 | S型/A | C | C | T | C | A | C | A | G | G | C | C | C | G | C | C | C | G | T | C | A | T | C |
| 2019-nCoV/SDRZ134/2020 | S型/A | C | C | T | C | A | C | A | A | G | C | C | C | G | C | C | C | G | T | C | A | T | C |
| 2019-nCoV/SDRZ146/2020 | L型/B | C | C | T | C | A | C | A | G | G | C | C | C | G | C | C | C | G | C | C | A | T | C |
| 2019-nCoV/SDJN314/2020 | L型/B | C | C | T | C | A | C | A | G | G | C | C | C | G | C | C | C | G | C | C | A | T | C |
| 2019-nCoV/SDDZ-Italy001/2020 | L II.1/B.1 | C | T | T | T | A | C | A | G | G | T | C | C | G | C | C | C | G | C | C | A | T | C |
| 2019-nCoV/SDQD-USA001/2020 | L型/B.1 | C | T | T | C | A | C | A | G | G | T | C | C | T | C | C | C | T | C | C | A | T | C |
| 2019-nCoV/SDQD-DG001/2020 | L.I/B.1.129 | C | T | T | C | A | T | A | G | G | T | T | T | G | T | T | C | G | C | C | A | T | C |
| 2019-nCoV/SDQD-417/2020 | L.I/B.1.129 | C | T | T | C | A | T | A | G | G | T | T | T | G | T | T | C | G | C | C | A | T | C |
| 2019-nCoV/SDQD-426/2020 | L.I/B.1.129 | C | T | T | C | A | T | A | G | G | T | T | T | G | T | T | C | G | C | T | A | T | C |
| 毒株名称 | ORF1b | S | ORF3a | M | ORF7a | ORF8 | N | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 14408 | 15471 | 15760 | 17977 | 18090 | 21575 | 22020 | 23299 | 23403 | 23755 | 24680 | 25244 | 25563 | 25947 | 26144 | 26530 | 27746 | 28144 | 28881 | 28882 | 28883 | |
| 2019-nCoV/Wuhan/WIV04/2019 | C | A | G | C | G | C | T | A | A | G | G | G | G | G | G | A | G | T | G | G | G |
| 2019-nCoV/SDBZ017/2020 | C | A | G | C | A | C | T | A | A | G | G | G | G | T | G | A | G | C | G | G | G |
| 2019-nCoV/SDLY217/2020 | C | A | G | C | G | C | T | A | A | G | G | G | G | G | G | A | G | C | G | G | G |
| 2019-nCoV/SDQD292/2020 | C | A | G | C | G | T | T | A | A | G | G | G | G | G | G | A | G | C | G | G | G |
| 2019-nCoV/SDQD293/2020 | C | A | G | C | G | T | T | A | A | G | G | G | G | G | G | A | G | C | G | G | G |
| 2019-nCoV/SDQD299/2020 | C | A | G | T | G | C | T | A | A | G | G | G | G | G | G | A | G | T | G | G | G |
| 2019-nCoV/SDDZ042/2020 | C | A | G | C | G | C | T | A | A | G | G | G | G | G | G | A | G | C | G | G | G |
| 2019-nCoV/SDDZ279/2020 | C | A | G | C | G | C | T | A | A | G | G | G | G | G | G | A | G | T | G | G | G |
| 2019-nCoV/SDDZ357/2020 | C | A | G | C | G | C | T | A | A | G | G | G | G | G | T | A | G | T | G | G | G |
| 2019-nCoV/SDDZ645/2020 | C | A | G | C | G | C | T | A | A | G | G | G | G | G | T | A | G | T | G | G | G |
| 2019-nCoV/SDZB111/2020 | C | A | G | C | G | C | T | A | A | G | G | G | G | G | G | A | G | C | G | G | G |
| 2019-nCoV/SDRZ134/2020 | C | A | G | C | G | C | T | A | A | G | G | G | G | G | G | A | G | C | G | G | G |
| 2019-nCoV/SDRZ146/2020 | C | A | G | C | G | C | C | A | A | G | T | T | G | G | G | A | G | T | G | G | G |
| 2019-nCoV/SDJN314/2020 | C | A | G | C | G | C | T | A | A | G | G | G | G | G | T | A | G | T | G | G | G |
| 2019-nCoV/SDDZ-Italy001/2020 | T | A | A | C | G | T | T | A | G | G | G | G | T | G | G | A | G | T | G | G | G |
| 2019-nCoV/SDQD-USA001/2020 | T | A | G | C | G | C | T | A | G | G | G | G | G | G | G | G | T | T | G | G | G |
| 2019-nCoV/SDQD-DG001/2020 | T | A | G | C | G | C | T | G | G | T | G | G | G | G | G | A | G | T | A | A | C |
| 2019-nCoV/SDQD-417/2020 | T | A | G | C | G | C | T | G | G | T | G | G | G | G | G | A | G | T | A | A | C |
| 2019-nCoV/SDQD-426/2020 | T | G | G | C | G | C | T | G | G | T | G | G | G | G | G | A | G | T | A | A | C |
8782和28144这两个核苷酸位点的突变为L型和S型的主要区别,其中L型第8782和28144位点分别为C和T,S型第8782和28144位点分别为T和C。2020年1~2月收集的病例基因序列中,有7份为S型,6份为L型。2020年3月以后收集的5份病例基因序列均为L型,其中包括2020年3月的2例输入病例和10月青岛疫情的3例病例。
C241T、C3037T、C14408T和A23403G变异是2019-nCoV L型欧洲家系的分支位点,存在于2020年3月以后收集的5份病例基因序列中,如同时存在G28 881A、G28 882A和G28883C这3个核苷酸突变位点则可划分为L型欧洲家系分支I。青岛疫情的3例病例的基因序列属于L型欧洲家系分支I。G25563T为L型欧洲家系分支II特征位点,而C1059T和C18877T可将L型欧洲家系分支II进一步划分为II.1和II.3。输入性病例分离的毒株2019-nCoV/SDDZ-Italy001/2020同时存在G25563T和C1059T突变位点,属于L型欧洲家系分支II.1。
18株新型冠状病毒共有24个氨基酸位点发生了变异,分布于4个阅读框,按氨基酸突变总数由多到少依次为:ORF1a区9个、S蛋白区7个、ORF1b区和ORF3a区均为3个及N蛋白区2个。从氨基酸位点变化来看,早期采集的13株毒株有11个位点发生了变异,2株输入病例分离到的病毒有6个氨基酸位点发生了变异,青岛疫情3例病例存在9个氨基酸位点的变异,关键氨基酸位点变化具体见表2。从氨基酸序列来看,2例输入性病例和3例青岛疫情感染病例所分离到的病毒均在S蛋白发生了D614G的变异,且从青岛疫情分离到的3株病毒在S蛋白的M731I和N蛋白的R203 K和G204R也发生了变异。
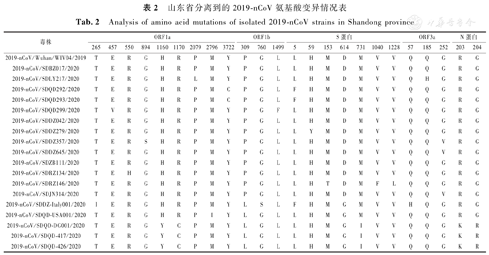
山东省分离到的2019-nCoV氨基酸变异情况表
Analysis of amino acid mutations of isolated 2019-nCoV strains in Shandong province
山东省分离到的2019-nCoV氨基酸变异情况表
Analysis of amino acid mutations of isolated 2019-nCoV strains in Shandong province
| 毒株 | ORF1a | ORF1b | S蛋白 | ORF3a | N蛋白 | |||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 265 | 457 | 550 | 894 | 1160 | 1170 | 2079 | 2796 | 3722 | 309 | 760 | 1499 | 5 | 59 | 153 | 614 | 731 | 1040 | 1228 | 57 | 185 | 252 | 203 | 204 | |
| 2019-nCoV/Wuhan/WIV04/2019 | T | E | R | G | H | R | P | M | Y | P | G | L | L | H | M | D | M | V | V | Q | Q | G | R | G |
| 2019-nCoV/SDBZ017/2020 | T | E | R | G | H | R | P | M | Y | P | G | L | L | H | M | D | M | V | V | Q | Q | G | R | G |
| 2019-nCoV/SDLY217/2020 | T | E | R | G | H | R | L | M | Y | P | G | L | L | H | M | D | M | V | V | Q | H | G | R | G |
| 2019-nCoV/SDQD292/2020 | T | E | R | G | H | R | P | M | C | P | G | L | F | H | M | D | M | V | V | Q | Q | G | R | G |
| 2019-nCoV/SDQD293/2020 | T | E | R | G | H | R | P | M | C | P | G | L | F | H | M | D | M | V | V | Q | Q | G | R | G |
| 2019-nCoV/SDQD299/2020 | T | V | R | G | H | R | P | M | Y | P | G | F | L | H | M | D | M | V | V | Q | Q | G | R | G |
| 2019-nCoV/SDDZ042/2020 | T | E | R | G | H | R | P | M | Y | P | G | L | L | H | M | D | M | V | V | Q | Q | G | R | G |
| 2019-nCoV/SDDZ279/2020 | T | E | R | G | H | R | P | M | Y | P | G | L | L | Y | M | D | M | V | V | Q | Q | G | R | G |
| 2019-nCoV/SDDZ357/2020 | T | E | R | S | H | R | P | M | Y | P | G | L | L | H | M | D | M | V | V | Q | Q | V | R | G |
| 2019-nCoV/SDDZ645/2020 | T | E | R | G | H | R | P | M | Y | P | G | L | L | H | M | D | M | V | V | Q | Q | V | R | G |
| 2019-nCoV/SDZB111/2020 | T | E | R | G | H | R | P | M | Y | P | G | L | L | H | M | D | M | V | V | Q | Q | G | R | G |
| 2019-nCoV/SDRZ134/2020 | T | E | H | G | H | R | P | M | Y | P | G | L | L | H | M | D | M | V | V | Q | Q | G | R | G |
| 2019-nCoV/SDRZ146/2020 | T | E | R | G | H | R | P | M | Y | P | G | L | L | H | T | D | M | F | L | Q | Q | G | R | G |
| 2019-nCoV/SDJN314/2020 | T | E | R | G | H | R | P | M | Y | P | G | L | L | H | M | D | M | V | V | Q | Q | V | R | G |
| 2019-nCoV/SDDZ-Italy001/2020 | I | E | R | G | H | R | P | M | Y | L | S | L | F | H | M | G | M | V | V | H | Q | G | R | G |
| 2019-nCoV/SDQD-USA001/2020 | T | E | R | G | H | R | P | I | Y | L | G | L | L | H | M | G | M | V | V | Q | Q | G | R | G |
| 2019-nCoV/SDQD-DG001/2020 | T | E | R | G | Y | C | P | M | Y | L | G | L | L | H | M | G | I | V | V | Q | Q | G | K | R |
| 2019-nCoV/SDQD-417/2020 | T | E | R | G | Y | C | P | M | Y | L | G | L | L | H | M | G | I | V | V | Q | Q | G | K | R |
| 2019-nCoV/SDQD-426/2020 | T | E | R | G | Y | C | P | M | Y | L | G | L | L | H | M | G | I | V | V | Q | Q | G | K | R |
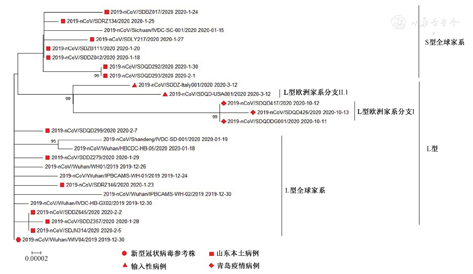
本研究测定的18株病毒全基因组和NCBI和GISAID数据库中早期的代表株序列进行系统进化分析。结果显示,18份全基因组序列分成2个分支,其中一支属于S型全球家系分支,另一支属于L型分支,在L型分支中,又分成了L型欧洲家系分支I、II.1和全球家系。具体见图2。

本研究利用Vero-E6细胞系成功分离到22株新型冠状病毒,病毒分离结果显示,分离到的病毒在Vero-E6细胞可稳定传代,且部分病毒的TCID50在103.95/100 μl到104.75/100 μl之间。
2019-nCoV属于冠状病毒科,为单股正链RNA病毒,由于RNA本身特性,新冠病毒在传播过程中存在着基因变异,变异速率约为1~2个核苷酸/月[8]。由于病毒基因变异携带有每个病毒可识别的独特信息,全基因组测序和进化分析能为病原特性评估和疫情溯源提供重要依据。本研究采用2019-nCoV全基因组捕获技术结合二代测序获得了18份全基因组序列,全基因组序列分析结果显示,新型冠状病毒的同源性较高,具有99.9%的同源性,进一步证实了2019-nCoV基因组变异性较低。基因进化分析结果显示不同时期的全基因组序列属于不同的基因型,提示不同时期的病毒来源不同。2020年1~2月山东省流行的毒株主要是L型和S型,13份全基因组序列中7份为S型,6份为L型,与早期武汉疫情的序列高度同源,说明1-2月的病例来源主要与武汉疫情有关;而2020年3月的输入病例的序列属于L分支欧洲家系,且具有C241T、C3037T、C14408T、A23403G四个共同的变异位点,说明病例感染来源与欧洲流行的毒株相关,而非本土感染;青岛暴发疫情获得的序列均属于L分支欧洲家系I.1,且共同具有G23755T、G28881A、G28882A和G28883C四个共同的变异位点,说明3个病例为同一感染来源,且与早期武汉疫情的序列处于不同的分支上,说明青岛疫情不是本土2019-nCoV的持续传播,也不是动物病毒的"溢出",而是冷链环境标本传染给人而导致的本土暴发[9]。
通过与参考序列比较,18份全基因组序列存在42个核苷酸变异,除10个分支特征性核苷酸突变位点外,其余位点多为随机突变位点,其中以ORF1a基因变异最多,从而导致了9个氨基酸发生了变异;其次为N基因发生了7个核苷酸变异,导致7个氨基酸的变异,且3月份后分离的5株病毒均发生了D614G的变异。
2019-nCoV的S蛋白为病毒表面的刺突蛋白,主要介导病毒与宿主细胞的融合,是治疗性抗体和药物筛选的关键靶点。研究表明,S蛋白的D614G突变可以改变S蛋白三聚体氢键的相互作用,从而增加与ACE2受体结合的能力,从而增强其感染性和病毒在细胞中的复制能力[10,11,12],但此突变不会削弱抗体对病毒的中和作用[13]。因此要加强对D614G变异位点相关病毒的监测,防止病毒的扩散、传播。同时,我们也在S蛋白的第731位发现了M-I的变异,可能为青岛疫情特有的特征性变异,此变异对病毒是否有影响仍需进一步观察。
2019-nCoV N蛋白的R203 K和G204R突变能提高病毒对人群的适应性、存活率和适应能力[14]。3株青岛暴发疫情病例的N蛋白均发生了R203 K和G204 R的变异。对于这些位点的变异及相关的作用,还需进一步验证。
2019-nCoV的成功分离和全基因组测序,为病毒的溯源、药物筛选和疫苗效果评价上提供了基础。随着疫情的进一步扩散,病毒仍在不断的发生变异,表现为流行株具备多种基因型别,且持续出现新的变异株。截止目前,已经发现Alpha、Beta、Gamma和Delta等四种高度关注的变异株,其传播力、致病力、受体亲和力等关键的特征已经发生了变异。因此,我们应进一步加强监测,持续开展病毒分离和基因变异监测,提高监测能力,为快速识别新的变异株、开展病例溯源、及时调整疫苗接种策略提供依据。
所有作者均声明不存在利益冲突



























