
基于生物信息学方法筛选肾透明细胞癌关键基因,识别可能的微RNA(miRNA)-mRNA作用轴,探讨相关基因在肾透明细胞癌组织和细胞中的表达。
选取基因表达综合(GEO)数据库基因表达谱GSE40435、GSE71302数据集,肾透明细胞癌TCGA-KIRC数据集来自癌症基因组图谱(TCGA)数据库。采用R软件筛选差异表达的mRNA和miRNA,并对mRNA进行功能富集分析。采用STRING数据库和Cytoscape软件进行蛋白质互作分析。采用Oncomir数据库筛选预后相关的差异表达miRNA。采用TargetScan和miRDB靶基因预测工具筛选miRNA可能调控的靶基因。收集2021年6月至12月山西医科大学第一医院收治的34例肾透明细胞癌患者的组织样本及临床资料,并选择正常肾细胞株293T和肾透明细胞癌细胞株786O。采用实时荧光定量聚合酶链反应检测基因的相对表达量;蛋白质印迹法、免疫组织化学染色法检测目的蛋白的表达水平;双荧光素酶报告基因实验验证基因间的靶向关系。
差异分析筛选得到1 351个差异表达mRNA和50个差异表达miRNA。功能富集分析提示肾透明细胞癌中脂肪酸代谢、异生物质代谢等途径被抑制,细胞凋亡、免疫应答途径被激活。蛋白质互作分析提示肾透明细胞癌中信号转导、蛋白泛素化等途径起到关键作用。筛选结果显示miRNA-224-5p(miR-224-5p)与肾透明细胞癌进展关系最紧密,且在肿瘤组织中高表达,其预后相关靶基因为NEDD4L。肾透明细胞癌组织与癌旁组织的NEDD4L mRNA相对表达量分别为0.138±0.103、1.000±0.026(t=46.23,P<0.05);miR-224-5p相对表达量分别为1.000±0.043、0.129±0.108(t=45.28,P<0.05)。不同分级及分期肾透明细胞癌组织NEDD4L mRNA、miR-224-5p的表达差异均有统计学意义(均P<0.05)。NEDD4L蛋白在肾透明细胞癌中表达降低。293T与786O细胞中NEDD4L基因相对表达量分别为1.000±0.125、0.210±0.044 (t=17.52,P<0.05);miR-224-5p基因相对表达量分别为0.209±0.049、1.000±0.234(t=10.609,P<0.05)。转染后的293T细胞样本中,miRNA模拟物组与阴性对照组的NEDD4L mRNA相对表达量分别为0.236±0.062、1.000±0.024,差异有统计学意义(t=43.56,P<0.05)。NEDD4L蛋白在miRNA模拟物组中表达降低。双荧光素酶报告基因实验提示NEDD4L是miR-224-5p的直接靶基因。
在肾透明细胞癌中,miR-224-5p靶向调节NEDD4L表达,这一机制可能与肾透明细胞癌的发生、发展有关。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
肾细胞癌是起源于肾小管上皮细胞的恶性肿瘤,占所有肿瘤的2%~3%,在泌尿系统肿瘤中致死率居第三,发病率和死亡率逐年增高[1,2,3]。肾透明细胞癌是肾细胞癌的亚型之一,恶性程度高[4]。手术是局限性肾透明细胞癌比较有效的治疗方式,但20%~40%的患者预后并不理想且容易复发、转移[5]。对于转移性肾透明细胞癌,目前可以采取化疗和免疫治疗,但治疗效果不理想。因此探索肾透明细胞癌潜在的分子生物学机制,找到新的诊断、预后预测及指导临床治疗的靶点十分重要。微RNA(miRNA)是非编码基因的重要组成部分,可以通过影响下游靶基因表达而影响肿瘤进展[6]。目前关于肾透明细胞癌发生、发展的基因调控机制仍不完全清楚。本研究基于生物信息学方法对高通量测序数据进行筛选,寻找肾透明细胞癌关键基因靶点,识别可能的miRNA-mRNA作用轴,探讨肾透明细胞癌发生、发展的潜在分子机制,为肾透明细胞癌患者的诊断、预后预测以及指导临床治疗提供理论依据。
纳入标准:术前未接受放疗、化疗和分子靶向治疗等抗肿瘤治疗,术后组织病理确诊为肾透明细胞癌,临床信息完善。收集2021年6月至12月于山西医科大学第一医院行根治性肾切除术的肾透明细胞癌患者组织样本及临床资料(包括年龄、性别、T分期、病理分级)。最终纳入34例患者,包括男性24例,女性10例;年龄33~83岁;T分期Ⅰ、Ⅱ期27例,Ⅲ、Ⅳ期7例;病理分级Ⅰ、Ⅱ级28例,Ⅲ、Ⅳ级6例。本研究经山西医科大学第一医院伦理委员会批准(批准文号:2021K034),所有患者均签署知情同意书。
正常肾细胞株293T和肾透明细胞癌细胞株786O购自中国科学院上海细胞库,均进行常规培养。293T细胞用DMEM完全培养液,786O细胞用RPMI 1640完全培养液,完全培养液中均加入10%胎牛血清及1%青链霉素双抗,于37 ℃、5% CO2细胞培养箱中培养,2~3 d换液或传代,取对数生长期细胞用于后续实验。
选取基因表达综合(GEO)数据库基因表达谱GSE40435、GSE71302数据集,分别包含101对肾透明细胞癌肿瘤组织及其配对癌旁组织的mRNA数据(基于GPL10558平台)和5对肾透明细胞癌肿瘤组织及其配对癌旁组织的miRNA数据(基于GPL10850平台)。肾透明细胞癌TCGA-KIRC数据来自癌症基因组图谱(TCGA)数据库,包含基因表达数据和对应患者的临床信息。其中mRNA表达数据来自539例肿瘤组织及72例癌旁组织,miRNA表达数据来自545例肿瘤组织及71例癌旁组织,临床数据包含530例患者的临床信息。
用R软件Limma包分析基因表达数据获取差异表达基因,阈值设置为|log2FC|>1(FC为差异倍数),P<0.05。探针ID转换基因名以特定平台的注释文件为依据。丢弃无基因匹配探针。若一个基因匹配多个探针,以探针检测的最大值作为基因最终表达值。
基因集富集分析(GSEA)是一种通过特定计算方式查看既定生物学途径是否在不同状态下存在显著差异的分析方法。使用R软件ClusterProfiler包对所有基因进行GSEA[7]。
STRING数据库收集和整合了多种生物中蛋白质的相互作用,用于展示已知蛋白质分子间的功能联系,预测潜在的分子联系[8]。使用Cytoscape 3.9.1软件的MCODE插件对PPI进行评估,筛选重要功能模块。
Oncomir数据库收集了大量miRNA与癌症临床参数相关性的数据,用于筛选、识别关键调控的miRNA[9]。TargetScan和miRDB是两个比较全面的miRNA靶基因预测工具,用于检索miRNA可能调控的靶基因[10,11]。用TCGA数据库中数据进行生存分析,分析肾透明细胞癌中筛选出的miRNA及其靶基因的表达情况,绘制受试者工作特征(ROC)曲线,验证二者对肾透明细胞癌的诊断效能。
利用TRIzol法提取组织和细胞株总RNA。用聚合酶链反应(PCR)试剂盒(AUQ01,北京全式金生物技术公司)对RNA模板进行反转录以检测NEDD4L和内参基因β-actin的表达水平。用加尾法PCR试剂盒(AT351,北京全式金生物技术公司)对RNA模板进行反转录并检测miR-224-5p和内参基因U6的表达水平。引物序列:NEDD4L正向引物5'-TCAGAGGAAG TGAATATCGCTGG-3',反向引物5'-ACCTTGAGGGT TCTCTTT-3';GAPDH正向引物5'-GTCCACCACCCTG TTGCTGTA-3',反向引物5'-ACCCACTCCTCCACCTT TGA-3';miR-224-5p引物:5'-AACAGTGTCAAGTCAC TAGTGGTT-3';U6正向引物5'-TCGCTTCGGCAGCA CATAT-3',反向引物5'-ATTTGCGTGTCATCCTTGC-3'。以2-△△Ct计算目的基因相对表达量。
提取组织和细胞总蛋白,应用BCA法进行蛋白定量。使用10%十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)分离蛋白,随即将蛋白电转至聚偏二氟乙烯(PVDF)膜,以质量分数5%脱脂奶粉的TBST进行封闭2 h后,分别加入NEDD4L抗体(ab46521,英国Abcam公司,1∶1 000)及内参抗体,4 ℃过夜。培养完成后将膜使用TBST洗涤3次后加入二抗(稀释比例1∶5 000),室温下培养1.5 h。使用化学发光系统检测信号并进行分析。每组实验重复4次,以β-actin为内参照。
临床组织样本经过石蜡包埋、组织切片、脱蜡水化、抗原修复和灭活内源性过氧化物酶后,使用NEDD4L抗体(A8085,武汉爱博泰克生物科技有限公司,1∶1 000)和二抗对组织切片进行IHC染色,二氨基联苯胺(DAB)显色,苏木精对比染色。
野生型NEDD4L(WT-NEDD4L)质粒、突变型NEDD4L(MUT-NEDD4L)质粒、miR-224-5p模拟物、miR-224-5p阴性对照(miR-NC)由上海汉恒生物科技公司合成。12孔板中培养293T细胞至融合度约60%,将细胞分别转染miR-224-5p模拟物、miR-NC,明确转染效率,转染24 h检测细胞NEDD4L基因表达情况,48 h检测细胞蛋白表达情况。另取细胞分别共转染WT-NEDD4L+miR-224-5p模拟物、WT-NEDD4L+ miR-NC、MUT-NEDD4L+miR-224-5p模拟物、MUT-NEDD4L+miR-NC,转染24 h后应用双荧光素酶检测试剂盒(上海汉恒生物科技公司)检测相对荧光素酶活性。以萤火虫荧光素酶荧光值为内参,以海肾荧光素酶荧光值与萤火虫荧光素酶荧光值的比值表示相对荧光素酶活性。
采用SPSS 26.0、R 4.0.1、GraphPad Prism 8.3软件进行数据分析。计量资料符合正态分布,以 ±s表示,组间比较采用t检验。相关性分析采用Pearson相关性分析法。生存分析采用Kaplan-Meier法,并行log-rank检验。以P<0.05为差异有统计学意义。
±s表示,组间比较采用t检验。相关性分析采用Pearson相关性分析法。生存分析采用Kaplan-Meier法,并行log-rank检验。以P<0.05为差异有统计学意义。
GSE40435数据集共筛选得到1 351个差异表达mRNA,其中表达上调609个,表达下调742个。GSE71302数据集共筛选得到50个差异表达miRNA,其中表达上调25个,表达下调25个。
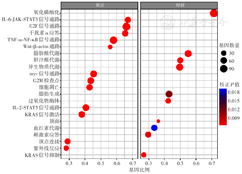
GSEA结果显示,肾透明细胞癌中氧化磷酸化、脂肪酸代谢、异生物质代谢、过氧化物酶体等途径被抑制;多种信号通路、免疫应答、细胞凋亡等途径被激活(图1)。

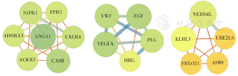
采用STRING数据库构建差异表达mRNA的PPI(PPI富集P<0.001)。选择结合置信度高(>0.9)的筛选条件,得到包含654个蛋白的PPI。用Cytoscape软件筛选得到重要功能模块,其中包括信号转导模块、生长因子模块、蛋白泛素化模块等(图2)。

用Oncomir数据库对显著差异表达的miRNA(|log2FC|>3,P<0.05)进行筛选,结果显示有3个关键miRNA与肾透明细胞癌的预后和进展显著相关,分别为miR-224-5p、miR-138-5p、miR-885-5p。其中miR-224-5p与肾透明细胞癌进展关系最紧密(t检验的P值最小),且在肿瘤组织中高表达。
将关键miRNA的潜在靶基因与差异表达mRNA合并,结合生存分析结果显示,miR-224-5p潜在调控的预后相关靶基因有1个;miR-138-5p潜在调控的预后相关靶基因有11个;miR-885-5p潜在调控的预后相关靶基因有11个(表1)。
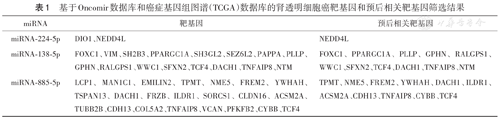
基于Oncomir数据库和癌症基因组图谱(TCGA)数据库的肾透明细胞癌靶基因和预后相关靶基因筛选结果
基于Oncomir数据库和癌症基因组图谱(TCGA)数据库的肾透明细胞癌靶基因和预后相关靶基因筛选结果
| miRNA | 靶基因 | 预后相关靶基因 |
|---|---|---|
| miRNA-224-5p | DIO1、NEDD4L | NEDD4L |
| miRNA-138-5p | FOXC1、VIM、SH2B3、PPARGC1A、SH3GL2、SEZ6L2、PAPPA、PLLP、GPHN、RALGPS1、WWC1、SFXN2、TCF4、DACH1、TNFAIP8、NTM | FOXC1、PPARGC1A、PLLP、GPHN、RALGPS1、WWC1、SFXN2、TCF4、DACH1、TNFAIP8、NTM |
| miRNA-885-5p | LCP1、MAN1C1、EMILIN2、TPMT、NME5、FREM2、YWHAH、TSPAN13、DACH1、FRZB、ILDR1、SORCS1、CLDN16、ACSM2A、TUBB2B、CDH13、COL5A2、TNFAIP8、VCAN、PFKFB2、CYBB、TCF4 | TPMT、NME5、FREM2、YWHAH、DACH1、ILDR1、ACSM2A、CDH13、TNFAIP8、CYBB、TCF4 |
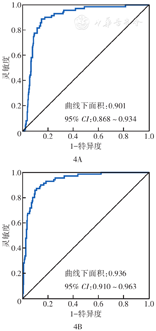
用TCGA数据库中数据对肾透明细胞癌NEDD4L、miR-224-5p的表达情况及诊断价值进行分析。配对差异分析结果显示,在肾透明细胞癌中,与癌旁组织相比较,肿瘤组织中NEDD4L表达下调,miR-224-5p表达上调(图3)。ROC曲线分析结果显示,NEDD4L和miR-224-5p均有较高诊断效能(曲线下面积分别为0.901和0.936,图4)。预后分析结果显示,NEDD4L高表达患者较低表达患者预后更好,而miR-224-5p低表达患者较高表达患者预后更好。
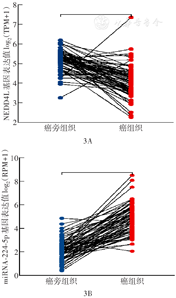


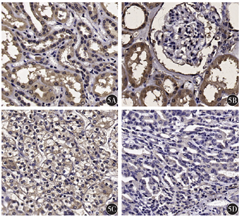
qRT-PCR结果显示,在肾透明细胞癌与配对癌旁组织中,NEDD4L mRNA相对表达量分别为0.138±0.103、1.000±0.026(t=46.23,P<0.05);miR-224-5p相对表达量分别为1.000±0.043、0.129±0.108(t= 45.28,P<0.05)。病理分级Ⅰ~Ⅱ级、Ⅲ~Ⅳ级患者NEDD4L mRNA相对表达量分别为0.033±0.017、0.161±0.100(t=3.07,P<0.05);miR-224-5p相对表达量分别为0.281±0.126、0.096±0.069,(t=5.14,P<0.05);T分期Ⅰ~Ⅱ期、Ⅲ~Ⅳ期患者NEDD4L mRNA相对表达量分别为0.027±0.012、0.167±0.097(t=3.75,P<0.05);miR-224-5p相对表达量分别为0.293±0.099、0.086±0.055(t=7.43,P<0.05)。不同年龄及性别间NEDD4L mRNA、miR-224-5p相对表达量差异均无统计学意义(均P>0.05)。蛋白质印迹法检测结果显示,与癌旁组织相比,NEDD4L蛋白在肾透明细胞癌组织中表达水平降低。IHC结果显示,与癌旁组织相比,NEDD4L蛋白在正常肾组织中表达水平升高,在肾透明细胞癌中表达水平降低;与病理分级Ⅰ级肾透明细胞癌组织相比,病理分级Ⅲ级肾透明细胞癌组织NEDD4L表达水平更低(图5)。

qRT-PCR结果显示,在正常肾细胞株293T与肾透明细胞癌细胞株786O中,NEDD4L mRNA相对表达量分别为1.000±0.125、0.210±0.044(t=17.52,P<0.05);miR-224-5p相对表达量分别为0.209±0.049、1.000±0.234(t=10.61,P<0.05)。蛋白质印迹法检测结果显示,NEDD4L蛋白在786O细胞株中表达水平较293T细胞株降低。转染效率显示,在miR-224-5p模拟物组及阴性对照组中,miR-224-5p相对表达量分别为0.223±0.034、1.000±0.032(t=39.17,P<0.05),提示转染效果好。293T细胞株中,miR-224-5p模拟物组及阴性对照组中NEDD4L mRNA相对表达量分别为0.236±0.062、1.00±0.024(t=43.56,P<0.05);蛋白质印迹法结果显示,NEDD4L蛋白在miR-224-5p模拟物组中表达降低。
双荧光素酶报告基因实验结果显示,WT-NEDD4L+ miR-224-5p模拟物组荧光素酶活性较WT-NEDD4L+miR-NC组降低(0.462±0.027比1.000±0.023),差异有统计学意义(t=55.80,P<0.05);MUT-NEDD4L+ miR-224-5p模拟物组荧光素酶活性与MUT-NEDD4L+ miR-NC组比较(0.996±0.067比1.000±0.015),差异无统计学意义(t=0.18,P=0.863)。miR-224-5p与NEDD4L的靶向结合模式见图6。
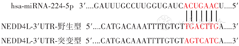
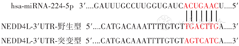
肾透明细胞癌是肾癌最常见的类型,与其他类型肾癌相比预后最差。尽管目前的影像学技术已经可以使部分患者在早期确诊,但是仍有部分患者在确诊时就处于晚期[12]。所以探索肾透明细胞癌潜在分子生物学机制,识别可能的诊断及预后预测靶点是必要的。
本研究通过生物信息学分析获得差异表达基因。对差异表达mRNA进行GSEA分析,结果显示脂肪酸代谢等代谢途径被抑制,这一结果与相关研究报道的肾透明细胞癌中存在异常的脂质堆积研究结果一致[13,14]。本研究发现细胞凋亡通路被激活,Ford等[15]研究发现肿瘤细胞可以通过细胞凋亡造成组织微环境的紊乱,进而促进癌症进展;Pavlyukov等[16]发现凋亡的肿瘤细胞会通过释放囊泡促进肿瘤增殖;Morana等[17]提出肿瘤细胞中存在细胞增殖与细胞死亡的平衡失调,其中异常活跃的细胞凋亡会刺激肿瘤微环境中的修复和再生反应,进而使肿瘤进展,这一观点为研究肾透明细胞癌的进展机制提供了新的见解和思路。PPI分析结果显示,信号转导模块、生长因子模块、蛋白泛素化模块可能在肾透明细胞癌调控中发挥着关键作用。
许多研究已经证实细胞中的miRNA可以通过调节下游靶基因的表达对细胞功能进行调控[6]。本研究筛选得到3个与肾透明细胞癌预后和进展相关的关键miRNA,靶基因筛选后得到23个关键靶基因。这些基因可能在肾透明细胞癌的发生、发展过程中起到重要调节作用,具体作用机制有待进一步研究。
NEDD4L是泛素连接酶NEDD4家族成员,参与蛋白泛素化,帮助蛋白内化和更新,可以维持细胞稳态[18]。NEDD4L与多种癌症的发生、发展密切相关[19]。如NEDD4L低表达有助于非小细胞肺癌的进展和转移[20];NEDD4L低表达有利于子宫内膜癌发生[21];与良性前列腺增生相比,NEDD4L在前列腺癌中低表达。此外,NEDD4L与患者预后存在一定的相关性,如NEDD4L低表达与胃癌患者的预后不良相关。同时miR-224-5p在多种癌症中发挥调节作用[22],miR-224-5p通过靶向PIK3R3/AKT3抑制葡萄膜黑色素瘤的恶性进展[23];miR-224-5p可能成为消化系统肿瘤的诊断和预后标志物[24];miR-224-5p通过靶向PTEN调控胰腺黏液性囊腺癌的增殖、迁移和侵袭[25];miR-224-5p通过靶向EGR2促进甲状腺乳头状癌的发展[26]。这些均提示NEDD4L和miR-224-5p可能在癌症中发挥着重要作用。本研究结果发现NEDD4L和miR-224-5p基因表达在肾透明细胞癌中降低,且与患者临床分期和病理分级相关。其中NEDD4L在肾透明细胞癌中低表达,miR-224-5p在肾透明细胞癌中高表达,体外实验证实了这一结论。预后分析提示NEDD4L高表达患者预后好,提示NEDD4L在肾透明细胞癌中可能通过介导蛋白泛素化发挥着保护作用。此外,我们通过转染及双荧光素酶报告基因实验证实,NEDD4L是miR-224-5p直接抑制的靶基因,miR-224-5p可结合在NEDD4L的3′-UTR区并直接抑制NEDD4L基因的表达,从而进一步对肾透明细胞癌的发生、发展产生影响。本研究存在一些不足之处:仅研究了miR‐224‐5p与NEDD4L的靶向调节关系,对于其具体的调节机制,如对肾透明细胞癌增殖、迁移和侵袭的影响,有待进一步研究。
所有作者均声明不存在利益冲突



























