
探讨脑表达X连锁(BEX)家族基因在泛癌中的差异性表达情况,及其在泛癌诊断和预后中的价值。
于2022年4月10日自加州大学圣克鲁斯分校(UCSC Xena)在线数据库下载来自癌症基因组图谱(TCGA)数据库中33种不同肿瘤的转录组测序(RNA-seq)数据、生存数据、免疫亚型、基于RNA和DNA甲基化的干细胞评分。利用R软件(V.4.2.0)limma包分析TCGA数据库中BEX家族基因的表达情况,通过Wilcox检验分析比较BEX家族基因在泛癌组织及正常组织中的差异性表达;根据BEX家族基因表达水平的中位值将泛癌患者分为高表达组和低表达组,采用Kaplan-Meier生存分析评估BEX家族基因表达与患者总生存(OS)的关系,采用Cox比例风险模型分析BEX家族基因表达对泛癌患者OS的影响并绘制森林图。根据相关指数Cor值分析BEX家族基因的表达与泛癌患者肿瘤微环境、肿瘤干细胞相关性,Spearman相关法分析胃癌组织的肿瘤微环境、肿瘤干细胞指数及BEX家族基因表达的相关性。从CellMiner数据库下载不同肿瘤细胞株RNA-seq及药物敏感性数据,分析BEX家族基因的表达与药物敏感性之间的相关性。使用Kruskal检验分析泛癌及胃癌免疫亚型与BEX家族基因表达间的相关性。
TCGA数据库中,BEX3在泛癌组织高表达,BEX2和BEX4在泛癌组织中度表达,BEX1和BEX5在泛癌组织相对低表达;BEX2、BEX3与BEX4在胆管癌中表达最高,BEX5在子宫内膜癌中表达最高,BEX1在浸润性乳腺癌中表达最高;相较于正常组织样本,BEX家族基因在多种肿瘤组织中的表达上调或下调(均P<0.05);生存分析表明BEX家族基因的表达与多种癌症OS相关,BEX1、BEX3、BEX4、BEX5高表达的部分肿瘤患者OS较低表达患者好,差异均有统计学意义(均P<0.05),其余BEX家族基因高表达患者OS较低表达患者差,差异均具有统计学意义(均P<0.05);Cox回归分析显示,BEX1高表达对于胃癌;BEX2高表达对于急性髓系白血病、胸腺癌、子宫内膜癌;BEX3高表达对于头颈鳞状细胞癌、肉瘤、胃癌、子宫内膜癌;BEX4高表达对于直肠腺癌、胃癌、子宫内膜癌;BEX5高表达对于肾嫌色细胞癌、胸腺癌,均为OS的危险因素(均P<0.05)。BEX家族基因表达与多数肿瘤的基质评分呈负相关(均P<0.05),与肿瘤干细胞评分呈正相关(均P<0.05)。BEX家族基因表达与胃癌肿瘤干细胞评分呈负相关(P<0.05),与基质评分、总评分呈正相关(均P<0.05)。CellMiner数据库不同肿瘤细胞株中,BEX家族基因可能与威罗菲尼(Cor=-0.368,P=0.004)、Kahalide f(Cor=-0.391,P=0.002)、O-6-苄基鸟嘌呤(Cor=-0.375,P=0.003)等药物的耐药性相关。BEX家族中各基因均与泛癌的免疫亚型有关,均在C5亚型中呈现高表达(均P<0.05);对于胃癌,除BEX5基因外(P=0.24),其余基因均在C3亚型中高表达(均P<0.05)。
BEX家族基因的表达与泛癌患者的预后密切相关,且对于肿瘤微环境、肿瘤干细胞以及药物敏感性存在影响。BEX家族基因可能是泛癌诊断及预后的潜在生物标志物。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
早期诊断对于癌症患者的预后至关重要,且患者预后与诊断时的年龄、病理分期、组织学类型、肿瘤标志物等密切相关[1,2]。目前肿瘤治疗以手术为主,但即使进行完全切除,患者总生存(OS)仍较差[3],且缺乏有效判断预后的指标。脑表达X连锁(BEX)家族基因包含BEX1、BEX2、BEX3、BEX4、BEX5、BEX6[4,5],其编码的蛋白质不仅参与神经发育、细胞周期调控以及细胞凋亡[6,7,8],而且其异常表达诱导肿瘤细胞的增殖及转移。BEX1已被证实与急性髓系白血病、食管鳞状细胞癌、小儿颅内室管膜瘤的发生有关[7,9,10];BEX2通过调节线粒体凋亡途径及G1细胞周期来促进乳腺癌细胞的增殖[8]。泛癌研究指同时对多种不同肿瘤的基因组进行分析,进而更好地对癌症进行诊断和治疗。本研究通过分析加州大学圣克鲁斯分校(UCSC Xena)在线数据库中的数据,探讨泛癌组织中BEX家族基因的差异表达情况及其与患者生存预后的相关性,同时探讨其与免疫亚型、肿瘤微环境、肿瘤干细胞以及药物敏感性的关系。
自UCSC Xena在线数据库(https://xena.ucsc.edu/)下载来自癌症基因组图谱(TCGA)数据库中33种不同肿瘤的转录组测序(RNA-seq)、生存数据、免疫亚型、基于RNA和DNA甲基化的干细胞评分,使用Perl编程语言整合所获数据,匹配患者的临床信息与基因表达信息,删除未知或不完整的信息。从CellMiner数据库(版本:2022.1,数据库:2.8.1,https://discover.nci. nih. gov/cellminer/home.do)下载不同肿瘤细胞株RNA-seq及药物敏感性数据(DTP NCI-60),删除临床试验及美国食品药品管理局(FDA)认证以外的数据。数据下载时间均为2022年4月10日。
利用R软件(V.4.2.0)limma包分析TCGA数据库中BEX家族基因的表达情况;利用ggpubr包、limma包进行Wilcox检验,分析比较BEX家族基因在泛癌组织及正常组织中的差异性表达;采用corrplot包分析BEX家族基因间的相关关系。
利用TCGA数据库中获取的生存数据分析BEX家族基因表达与患者预后的关系,根据BEX家族基因表达水平的中位值将泛癌患者分为高表达组和低表达组。利用survminer包及survival包绘制生存曲线,采用Kaplan-Meier法进行生存分析,并行log-rank检验。利用survival包进行Cox比例风险模型分析并绘制森林图,以确定BEX家族基因表达对泛癌患者OS的影响。
利用estimate包和limma包计算TCGA数据库中泛癌组织的基质细胞评分、免疫细胞评分及总评分,利用corrplot包绘制BEX家族基因表达与肿瘤微环境间相关性图表。使用肿瘤干细胞评分描述肿瘤细胞和肿瘤干细胞之间的相似性,利用limma包和corrplot包进行Spearman检验分析BEX家族基因的表达与肿瘤干细胞相关性,以相关系数Cor值表示BEX家族基因的表达与肿瘤微环境、肿瘤干细胞之间的潜在关联,绝对值越大代表相关性越强。利用reshape2包、ggplot2包、ggpubr包、limma包进行Spearman相关分析法分析胃癌组织的肿瘤微环境、肿瘤干细胞评分及BEX家族基因表达的相关性。
利用limma包、ggplot2包、reshape2包进行Kruskal检验,分析BEX家族基因与泛癌免疫亚型间的相关性,同样的方法得出BEX家族基因与胃癌免疫亚型间的相关性分析。
利用impute包、limma包、ggplot2包、ggpubr包分析,CellMiner数据库中不同肿瘤细胞株BEX家族基因的表达与药物敏感性之间的相关性,以Cor值表示BEX家族基因的表达与药物敏感性之间的潜在关联,绝对值越大代表相关性越强。
TCGA数据库中,BEX3在泛癌组织高表达,BEX2和BEX4在泛癌组织中度表达,BEX1和BEX5在泛癌组织相对低表达。进一步对比BEX基因在不同癌症组织中的表达水平发现,BEX2、BEX3与BEX4在胆管癌中表达最高,BEX5在子宫内膜癌中表达最高,BEX1在浸润性乳腺癌中表达最高(图1)。TCGA数据库中的RNA-seq数据分析表明,相较于正常组织样本,BEX家族基因在多种肿瘤组织中的表达上调或下调(均P<0.05)(表1)。
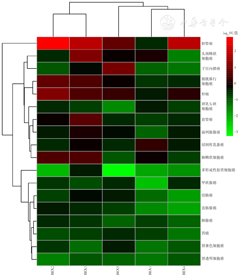

注:TCGA为癌症基因组图谱;BEX为脑表达X连锁;FC为差异倍数;每个小矩形中的颜色代表BEX家族基因在每种癌症中的表达情况;黑色代表非差异表达基因,红色代表基因表达上调,绿色代表基因表达下调,颜色越深表示基因的表达量越高
脑表达X连锁(BEX)家族基因在癌症基因组图谱(TCGA)数据库泛癌肿瘤组织中的表达情况
脑表达X连锁(BEX)家族基因在癌症基因组图谱(TCGA)数据库泛癌肿瘤组织中的表达情况
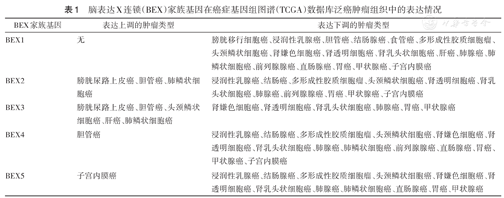
| BEX家族基因 | 表达上调的肿瘤类型 | 表达下调的肿瘤类型 |
|---|---|---|
| BEX1 | 无 | 膀胱移行细胞癌、浸润性乳腺癌、胆管癌、结肠腺癌、食管癌、多形成性胶质细胞瘤、头颈鳞状细胞癌、肾嫌色细胞癌、肾透明细胞癌、肾乳头状细胞癌、肝癌、肺腺癌、肺鳞状细胞癌、前列腺腺癌、直肠腺癌、胃癌、甲状腺癌、子宫内膜癌 |
| BEX2 | 膀胱尿路上皮癌、胆管癌、肺鳞状细胞癌 | 浸润性乳腺癌、结肠癌、多形成性胶质细胞瘤、头颈鳞状细胞癌、肾透明细胞癌、肾乳头状细胞癌、肺腺癌、前列腺腺癌、胃癌、甲状腺癌、子宫内膜癌 |
| BEX3 | 膀胱尿路上皮癌、胆管癌、头颈鳞状细胞癌、肝癌、肺鳞状细胞癌 | 肾嫌色细胞癌、肾透明细胞癌、肾乳头状细胞癌、肺腺癌、胃癌、甲状腺癌 |
| BEX4 | 胆管癌 | 浸润性乳腺癌、结肠腺癌、多形成性胶质细胞瘤、头颈鳞状细胞癌、肾嫌色细胞癌、肾透明细胞癌、肾乳头状细胞癌、肺腺癌、肺鳞状细胞癌、前列腺腺癌、直肠腺癌、胃癌、甲状腺癌、子宫内膜癌 |
| BEX5 | 子宫内膜癌 | 浸润性乳腺癌、结肠腺癌、多形成性胶质细胞瘤、头颈鳞状细胞癌、肾嫌色细胞癌、肾透明细胞癌、肾乳头状细胞癌、肺腺癌、肺鳞状细胞癌、直肠腺癌、胃癌、甲状腺癌 |
TCGA数据库中,根据不同癌症组织中BEX家族基因表达水平的中位值将患者分为高表达组和低表达组。Kaplan-Meier生存分析提示,BEX家族基因表达与多种癌症OS相关。其中BEX1高表达的肾嫌色细胞癌、肾透明细胞癌、脑低级别胶质瘤、葡萄膜黑色素瘤,BEX2高表达的肾透明细胞癌、脑低级别胶质瘤、间皮瘤患者较低表达患者OS好,差异均有统计学意义(均P<0.05);BEX3高表达的肾上腺皮质癌、弥漫大B细胞淋巴瘤、肾透明细胞癌、脑低级别胶质瘤、葡萄膜黑色素瘤患者较低表达患者OS好,差异均有统计学意义(均P<0.05);BEX4高表达的弥漫大B细胞淋巴瘤、肾嫌色细胞癌、肾透明细胞癌、脑低级别胶质瘤、肺腺癌、间皮瘤、胰腺癌、葡萄膜黑色素瘤患者较低表达患者OS好,差异均有统计学意义(均P<0.05);BEX5高表达的肾上腺皮质癌、浸润性乳腺癌、肾透明细胞癌、肺腺癌、皮肤黑色素瘤、葡萄膜黑色素瘤患者较低表达患者OS好,差异均有统计学意义(均P<0.05);其余BEX家族基因高表达患者OS较低表达患者的OS差,差异均具有统计学意义(均P<0.05)。
Cox回归分析进一步显示了BEX家族基因对泛癌OS的影响(图2,表2)。相较于BEX低表达的泛癌患者,BEX1高表达是肾上腺皮质癌、脑低级别胶质瘤、胰腺癌、葡萄膜黑色素瘤OS的保护因素(HR<1,P<0.05),是胃癌OS的危险因素(HR>1,P<0.05)。BEX2高表达是肾上腺皮质癌、肾透明细胞癌、肾乳头状细胞癌、脑低级别胶质瘤、间皮瘤、胰腺癌、嗜铬细胞瘤和副神经节瘤、皮肤黑色素瘤OS的保护因素(HR<1,P<0.05),而是急性髓系白血病、胸腺癌、子宫内膜癌患者OS的危险因素(HR>1,P<0.05)。BEX3高表达是弥漫大B细胞淋巴瘤、脑低级别胶质瘤、卵巢浆液性囊腺癌、胰腺癌、葡萄膜黑色素瘤患者OS的保护因素(HR<1,P<0.05),是头颈鳞状细胞癌、肉瘤、胃癌、子宫内膜癌患者OS的危险因素(HR>1,P<0.05)。BEX4高表达是肾上腺皮质癌、肾嫌色细胞癌、肾透明细胞癌、肾乳头状细胞癌、脑低级别胶质瘤、肺腺癌、间皮瘤、胰腺癌、甲状腺癌、葡萄膜黑色素瘤OS的保护因素(HR<1,P<0.05),是直肠腺癌、胃癌、子宫内膜癌OS的危险因素(HR>1,P<0.05)。BEX5高表达是肾透明细胞癌、肺腺癌、卵巢浆液性囊腺癌、胰腺癌、甲状腺癌、葡萄膜黑色素瘤OS的保护因素(HR<1,P<0.05),是肾嫌色细胞癌、胸腺癌OS的危险因素(HR>1,P<0.05)。
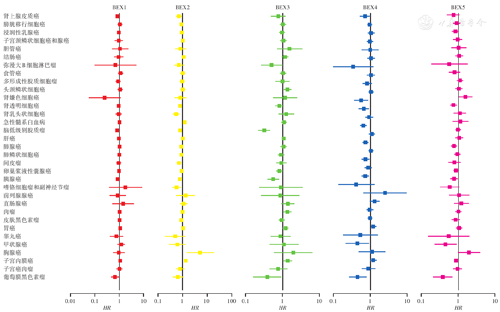

注:TCGA为癌症基因组图谱;BEX为脑表达X连锁;不同颜色的线表示不同基因在肿瘤中的风险比(HR)(基因高表达比低表达)
TCGA数据库泛癌中不同BEX家族基因表达水平的Cox比例风险模型生存分析结果
TCGA数据库泛癌中不同BEX家族基因表达水平的Cox比例风险模型生存分析结果
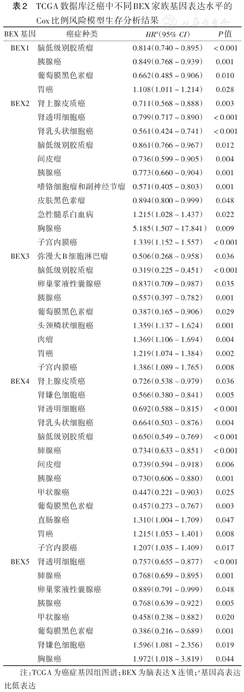
| BEX基因 | 癌症种类 | HRa(95% CI) | P值 |
|---|---|---|---|
| BEX1 | 脑低级别胶质瘤 | 0.814(0.740~0.895) | <0.001 |
| 胰腺癌 | 0.849(0.768~0.939) | 0.001 | |
| 葡萄膜黑色素瘤 | 0.662(0.485~0.906) | 0.010 | |
| 胃癌 | 1.108(1.011~1.214) | 0.028 | |
| BEX2 | 肾上腺皮质癌 | 0.711(0.568~0.888) | 0.003 |
| 肾透明细胞癌 | 0.799(0.717~0.890) | <0.001 | |
| 肾乳头状细胞癌 | 0.561(0.424~0.741) | <0.001 | |
| 脑低级别胶质瘤 | 0.861(0.766~0.967) | 0.012 | |
| 间皮瘤 | 0.736(0.599~0.905) | 0.004 | |
| 胰腺癌 | 0.773(0.660~0.904) | 0.001 | |
| 嗜铬细胞瘤和副神经节瘤 | 0.571(0.405~0.803) | 0.001 | |
| 皮肤黑色素瘤 | 0.894(0.800~0.999) | 0.048 | |
| 急性髓系白血病 | 1.215(1.028~1.437) | 0.022 | |
| 胸腺癌 | 5.185(1.507~17.841) | 0.009 | |
| 子宫内膜癌 | 1.339(1.152~1.557) | <0.001 | |
| BEX3 | 弥漫大B细胞淋巴瘤 | 0.506(0.268~0.958) | 0.036 |
| 脑低级别胶质瘤 | 0.319(0.225~0.451) | <0.001 | |
| 卵巢浆液性囊腺癌 | 0.837(0.709~0.987) | 0.035 | |
| 胰腺癌 | 0.557(0.397~0.782) | 0.001 | |
| 葡萄膜黑色素瘤 | 0.387(0.165~0.906) | 0.029 | |
| 头颈鳞状细胞癌 | 1.359(1.137~1.624) | 0.001 | |
| 肉瘤 | 1.369(1.106~1.694) | 0.004 | |
| 胃癌 | 1.219(1.074~1.384) | 0.002 | |
| 子宫内膜癌 | 1.386(1.089~1.765) | 0.008 | |
| BEX4 | 肾上腺皮质癌 | 0.726(0.538~0.979) | 0.036 |
| 肾嫌色细胞癌 | 0.566(0.380~0.841) | 0.005 | |
| 肾透明细胞癌 | 0.692(0.588~0.815) | <0.001 | |
| 肾乳头状细胞癌 | 0.664(0.503~0.876) | 0.004 | |
| 脑低级别胶质瘤 | 0.650(0.549~0.769) | <0.001 | |
| 肺腺癌 | 0.734(0.633~0.851) | <0.001 | |
| 间皮瘤 | 0.739(0.594~0.918) | 0.006 | |
| 胰腺癌 | 0.730(0.606~0.880) | 0.001 | |
| 甲状腺癌 | 0.447(0.221~0.903) | 0.025 | |
| 葡萄膜黑色素瘤 | 0.457(0.273~0.767) | 0.003 | |
| 直肠腺癌 | 1.310(1.004~1.709) | 0.047 | |
| 胃癌 | 1.215(1.053~1.401) | 0.008 | |
| 子宫内膜癌 | 1.207(1.035~1.409) | 0.017 | |
| BEX5 | 肾透明细胞癌 | 0.757(0.655~0.877) | <0.001 |
| 肺腺癌 | 0.768(0.659~0.895) | 0.001 | |
| 卵巢浆液性囊腺癌 | 0.889(0.791~0.999) | 0.048 | |
| 胰腺癌 | 0.768(0.639~0.922) | 0.005 | |
| 甲状腺癌 | 0.458(0.238~0.882) | 0.020 | |
| 葡萄膜黑色素瘤 | 0.386(0.216~0.689) | 0.001 | |
| 肾嫌色细胞癌 | 1.596(1.081~2.356) | 0.019 | |
| 胸腺癌 | 1.972(1.018~3.819) | 0.044 |
注:TCGA为癌症基因组图谱;BEX为脑表达X连锁;a基因高表达比低表达
利用ESTIMATE算法计算泛癌的基质评分、免疫评分及总评分,结果显示,BEX家族基因的表达与多数癌症的肿瘤微环境评分呈负相关(Cor<0),而与肿瘤干细胞评分呈正相关(Cor>0)。通过Spearman检验进一步对胃癌组织的肿瘤微环境、肿瘤干细胞评分及BEX家族基因的表达进行相关性分析,结果显示,BEX家族基因与RNA干性评分(RNAss)呈负相关(P<0.05);BEX3、BEX4的表达与DNA干性评分(DNAss)呈负相关(P<0.05);BEX1、BEX2、BEX3的表达与基质评分呈正相关(P<0.05);BEX2、BEX4的表达与免疫评分间呈正相关(P<0.05);BEX1、BEX2、BEX3、BEX4的表达与总评分之间呈正相关(P<0.05)(表3)。
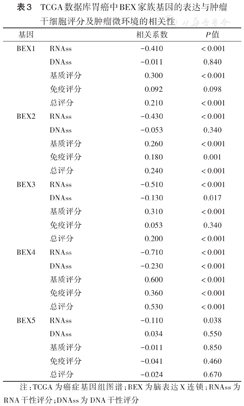
TCGA数据库胃癌中BEX家族基因的表达与肿瘤干细胞评分及肿瘤微环境的相关性
TCGA数据库胃癌中BEX家族基因的表达与肿瘤干细胞评分及肿瘤微环境的相关性
| 基因 | 相关系数 | P值 | |
|---|---|---|---|
| BEX1 | RNAss | -0.410 | <0.001 |
| DNAss | -0.011 | 0.840 | |
| 基质评分 | 0.300 | <0.001 | |
| 免疫评分 | 0.092 | 0.098 | |
| 总评分 | 0.210 | <0.001 | |
| BEX2 | RNAss | -0.430 | <0.001 |
| DNAss | -0.053 | 0.340 | |
| 基质评分 | 0.260 | <0.001 | |
| 免疫评分 | 0.180 | 0.001 | |
| 总评分 | 0.240 | <0.001 | |
| BEX3 | RNAss | -0.510 | <0.001 |
| DNAss | -0.130 | 0.017 | |
| 基质评分 | 0.310 | <0.001 | |
| 免疫评分 | 0.053 | 0.340 | |
| 总评分 | 0.200 | <0.001 | |
| BEX4 | RNAss | -0.710 | <0.001 |
| DNAss | -0.230 | <0.001 | |
| 基质评分 | 0.600 | <0.001 | |
| 免疫评分 | 0.360 | <0.001 | |
| 总评分 | 0.530 | <0.001 | |
| BEX5 | RNAss | -0.110 | 0.038 |
| DNAss | 0.034 | 0.550 | |
| 基质评分 | -0.011 | 0.850 | |
| 免疫评分 | -0.041 | 0.460 | |
| 总评分 | -0.024 | 0.670 |
注:TCGA为癌症基因组图谱;BEX为脑表达X连锁;RNAss为RNA干性评分;DNAss为DNA干性评分
根据TCGA数据库中33种不同癌症的免疫肿瘤微环境及RNA-seq数据,泛癌免疫亚型分为组织治愈型(C1)、干扰素γ主导型(C2)、炎症型(C3)、淋巴细胞耗竭型(C4)、免疫静默型(C5)以及转化生长因子β主导型(C6)。BEX家族中各基因表达在免疫亚型中差异有统计学意义(均P<0.05),均在C5亚型中呈现高表达,在C3、C4亚型中度表达,在其余亚型中呈现低度或者中度表达。对于胃癌,除BEX5基因外(P=0.240),其余基因的表达均与胃癌存在关联(均P<0.05):BEX1、BEX2、BEX3、BEX4均在C3亚型中呈现高表达,在C1、C2亚型中度表达。
CellMiner数据库不同肿瘤细胞株中,BEX家族基因的表达与芬维A胺、氟达拉滨、奈拉滨、迪皮质醇、三氧化二砷、白屈菜红碱、异环磷酰胺、PX-316(Akt抑制剂)、吡唑啉吖啶、氟奋乃静等多种化疗药物及肿瘤靶向药物的敏感性相关(表4)。
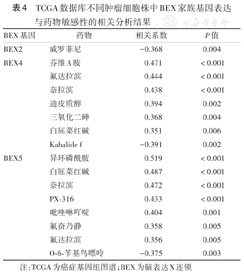
TCGA数据库不同肿瘤细胞株中BEX家族基因表达与药物敏感性的相关分析结果
TCGA数据库不同肿瘤细胞株中BEX家族基因表达与药物敏感性的相关分析结果
| BEX基因 | 药物 | 相关系数 | P值 |
|---|---|---|---|
| BEX2 | 威罗菲尼 | -0.368 | 0.004 |
| BEX4 | 芬维A胺 | 0.471 | <0.001 |
| 氟达拉滨 | 0.444 | <0.001 | |
| 奈拉滨 | 0.438 | <0.001 | |
| 迪皮质醇 | 0.394 | 0.002 | |
| 三氧化二砷 | 0.368 | 0.004 | |
| 白屈菜红碱 | 0.351 | 0.006 | |
| Kahalide f | -0.391 | 0.002 | |
| BEX5 | 异环磷酰胺 | 0.519 | <0.001 |
| 白屈菜红碱 | 0.487 | <0.001 | |
| 奈拉滨 | 0.472 | <0.001 | |
| PX-316 | 0.433 | <0.001 | |
| 吡唑啉吖啶 | 0.404 | 0.001 | |
| 氟奋乃静 | 0.358 | 0.005 | |
| 氟达拉滨 | 0.356 | 0.005 | |
| O-6-苄基鸟嘌呤 | -0.375 | 0.003 |
注:TCGA为癌症基因组图谱;BEX为脑表达X连锁
肿瘤是重要的公共卫生问题,尽管在诊断和治疗方面取得巨大突破,肿瘤患者的预后仍不容乐观[11]。早期诊断是提高肿瘤患者生存率以及改善预后的关键,生物标志物的提出为早期诊断指明了方向。研究表明,整合素αL表达的上调与患者较差的OS、预后不良和增强的免疫浸润相关[12];信使RNA的反向剪接产物环状RNA-8(8-circRNA)可作为早期检测胃癌的潜在生物标志物[13];转录抑制因子2(SNAI2)过表达可导致转录因子ELF3水平降低,从而负反馈持续激活SNAI2并促进胃癌细胞进一步增殖,引起不良预后[14]。以上研究表明特定的生物标志物对于早期诊断肿瘤、判断疾病进展及评估预后有重要作用。
本研究发现各型BEX家族基因在多种癌症中呈现不同程度的表达,与基因低表达组相比,较多高表达组癌症患者的OS更高。Cox回归分析结果显示,BEX家族基因的高表达可能是多种肿瘤患者(如胰腺癌、葡萄膜黑色素瘤等)OS保护因素。但是既往研究发现BEX家族基因成员的表达能够促进肿瘤细胞增殖,如Wang等[15]发现BEX1能够激活Wnt-β-catenin信号通路,从而维持肝癌干细胞的活性。Hu等[16]发现BEX2通过c-Jun氨基末端蛋白激酶(JNK-c-Jun)信号通路促进结直肠癌细胞的增殖;而该团队在另一项研究中发现BEX2可以沉默Hedgehog信号通路以抑制结直肠癌细胞的转移[17]。这些差异可能是由于肿瘤复杂的发病机制导致的,不同的信号通路可引起肿瘤细胞的差异变化。这些结果表明BEX家族基因的表达变化是癌症患者的重要预测因素,对于探究患者生存率、预后有着关键作用,有助于针对BEX家族基因靶向治疗的进一步研究。
本研究结果提示BEX家族基因的表达与肿瘤微环境和肿瘤干细胞相关。肿瘤微环境是肿瘤的重要组成部分,其改变可能通过复杂的信号网络调节促进肿瘤细胞的增殖和迁移[18,19]。ESTIMATE算法基于单样本基因集富集分析生成基质评分、免疫评分和总评分[20],对肿瘤微环境进行了评估。肿瘤干细胞评分反映肿瘤组织和正常组织之间的差异,较高的肿瘤干细胞评分与较差的预后和更大的致癌去分化相关,其中RNAss反映基因表达,DNAss反映表观遗传特征[21]。BEX家族基因的表达与多数癌症的基质评分呈负相关,与干细胞评分呈正相关,尤其是脑低级别胶质瘤、胃癌、甲状腺癌、睾丸癌。进一步行Spearman检验分析发现胃癌患者BEX家族基因的表达与基质评分、估计评分呈正相关;与肿瘤干细胞指数呈负相关,BEX4尤为明显。以上结果强调探索肿瘤干细胞与肿瘤微环境的必要性,通过调节BEX家族基因的表达或许能够重塑肿瘤微环境并影响肿瘤干细胞增殖,为未来多种癌症的治疗提供了新的方向;同时BEX家族基因可作为研究肿瘤干细胞增殖凋亡机制的靶点,通过靶向改变BEX的表达情况或可以抑制肿瘤干细胞的增殖。
免疫亚型分析发现BEX家族中各基因均在泛癌C5亚型中呈现高表达,此前已有研究表明C5亚型中免疫细胞浸润最低,患者的生存较差[22,23]。胃癌中除BEX5外,其余基因的表达均在C3亚型中呈现高表达,C3亚型患者的免疫细胞,如M1巨噬细胞、激活的自然杀伤细胞浸润增多,Toll样受体4(TLR4)、趋化因子CX3C的配体1(CX3CL1)等免疫调节基因增高,提示患者体内免疫功能的增强可能使预后良好[24];Foxp3阳性的调节性T淋巴细胞可以预防微生物相关炎症导致的癌变。Kim等[25]发现C3亚型胃癌患者组织内的Foxp3阳性肿瘤浸润淋巴细胞比例低于其他亚型,提示不良预后。这些结果表明BEX基因家族的表达可能与不同免疫亚型患者的预后相关,提示基因表达对于免疫信号具有预测作用。
目前已有多项研究表明基因的表达与肿瘤细胞的药物敏感性改变有关。Xiao等[26]发现bcl-2和BEX1相互作用后可抑制抗凋亡bcl-2/bcl-2相关X蛋白异二聚体的形成,进而促进伊马替尼诱导的细胞凋亡。Gao等[27]发现BEX3的表达在介导鼻咽癌细胞对顺铂的敏感性方面有独特作用。Lee等[28]表明抑制BEX4的表达可以提高长春新碱抑制肿瘤细胞增殖的疗效。本研究中BEX家族基因与药物敏感性密切相关,BEX2、BEX4的表达与威罗菲尼、Kahalide f的耐药相关;BEX2的表达与芬维A胺、氟达拉滨、奈拉滨、迪皮质醇、三氧化二砷、白屈菜红碱的敏感性增加有关;BEX5的表达与异环磷酰胺、白屈菜红碱、奈拉滨、PX-316、吡唑啉吖啶、氟奋乃静、氟达拉滨的敏感性增加相关。BEX家族基因可作为泛癌患者药物治疗效果的重要生物标志物,对其表达情况进行调整能够增加药物疗效,从而提高患者的生存率。
本研究发现BEX家族基因可作为泛癌诊断及预后的潜在生物标志物,其表达情况与患者的生存、预后相关,此外BEX家族基因的表达对于肿瘤微环境、肿瘤干细胞以及药物敏感性存在影响。但由于缺乏体外及体内实验数据,本研究的结果还需要进一步在动物或者细胞分子水平上进行证实,大样本、多中心的队列研究也需要未来探索。总之,泛癌组织中BEX家族基因的表达差异为未来的动物实验、细胞实验提供了研究方向,同时为患者的诊断、预后评估以及基因靶向治疗提供了重要理论依据,从而更好地预防并治疗癌症。
所有作者均声明不存在利益冲突



























