
长链非编码RNA(lncRNA)可在遗传水平、转录水平、转录后水平等多层面上通过多种机制调控基因表达,参与生理、病理过程,与多种人类重大疾病关系密切。lncRNA在造血细胞成熟、分化过程中也起重要的调控作用,深入探索其作用机制可为血液疾病的诊治提供新思路。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
长链非编码RNA(lncRNA)是一类长度超过200 nt、缺乏显著开放阅读框(OFR)的非编码RNA,定位于细胞核内或细胞质中[1]。lncRNA内部某些局部序列保守性高,剪切位点稳定,而且具有特定的二级结构和表达的时空特异性,可在多种层面调控基因表达,并参与基因组印记、X染色体沉默、染色质修饰、转录激活、转录干扰,以及转录后调控、核内运输、调节原癌基因活化等多种重要过程。在表观遗传学、转录及转录后多种层面发挥调控作用,是生命活动中重要的调控分子,广泛参与生物进化、胚胎发育、物质代谢等生理过程,与包括肿瘤在内的多种人类重大疾病密切相关。在造血系统中,血细胞是通过造血干细胞(HSC)增殖、分化、成熟而来的。通过对E14.5小鼠胎肝造血细胞lncRNA的表达分析发现其具有精细的谱系特异性[2],这对于确定lncRNA在调节血细胞分化中的作用十分有利,将进一步丰富我们对造血系统的认识,为疾病的诊治提供新的治疗靶点。需要指出的是,lncRNA作用复杂,范围广泛,有时即使单一的lncRNA在不同细胞内或同一细胞不同定位时发挥的作用也不同,与血细胞分化成熟及恶性血液病均密切相关,生理、病理作用相互交叉。文章按髓系和淋系两类,对参与血细胞成熟分化调控的lncRNA作一综述。
HOX基因座位功能复杂,通过基因测序发现HOXA和HOXB基因在正常HSC和原发急性髓系白血病(AML)中均有表达[3]。HOX基因座位能够转录数百种lncRNA,很多都与血细胞分化相关,其中由HOXC座位转录的lncRNA HOX转录反义RNA HOTAIR(HOX transcript antisense RNA)是第一个被发现具有反式转录调控作用的lncRNA,长度2.2 kb。Xing等[4]发现HOTAIR在白血病细胞株和原代白血病细胞中表达均明显增高,而且他们认为AML中HOTAIR表达增高与预后差相关。HOTAIR可以同时结合PRC2和LSD1,并介导这两种复合体结合到特异性的基因组位点,分别使染色体组蛋白H3第27位赖氨酸三甲基化和组蛋白H3第4位赖氨酸二甲基化,进而形成抑制性的染色体结构,导致基因沉默[5]。另一个与HOX基因有关的lncRNA是HOXA反义转录物髓系异构体(HOTAIRM1),位于HOX基因簇的3'端,从HOXA基因簇转录而来(HOXA1和HOXA2之间),与HOXA1协同表达,存在于HSC和胚胎干细胞中,被认为是骨髓细胞分化成熟所必需的调节因子。在全反式维甲酸诱导NB4细胞分化过程中HOTAIRM1表达上调,基因敲除NB4细胞HOTAIRM1可阻滞全反式维甲酸诱导粒细胞分化过程,使大量不成熟和增殖阶段细胞停滞在细胞周期的G1~S期。急性白血病细胞系中HOTAIRM1表达下调。目前研究认为HOTAIRM1可以通过调节整合素基因表达水平,从而调控骨髓成熟过程中细胞周期的进展[6]。
H19是最先被报道的lncRNA之一,位于染色体11p15.5,全长2.3 kb,主要存在细胞质内;该基因也是最早被发现的印记基因之一,父系基因印记,母系基因表达,主要通过外显子编码的miR-675和胰岛素样生长因子2(Igf2)发挥调节作用[7]。H19在长期HSC中较为活跃,而在短期HSC及多能干细胞中表达下调,通过调控Igf2-Igfr1通路在转录和转录后水平维持HSC静止[8]。Guo等[9]认为H19在bcr-abl阳性的慢性粒细胞白血病(CML)中表达上调,且受到癌基因c-myc正向调控。通过甲基化抑制剂5-硫唑嘌呤-2'-脱氧胞苷(5-Aza-2'-dC)能够抑制H19基因ICR区甲基化,下调H19基因表达[10]。
在嗜酸性粒细胞分化过程中,lncRNA eosinophil granule ontogeny(EGO)表达丰度增加,翻译调控二级颗粒蛋白,是嗜酸性粒细胞分化所必需的。在白细胞介素5(IL-5)刺激嗜酸性粒细胞分化过程中,沉默EGO后可下调CD34+ HsC及体外CD34+细胞系主要碱性蛋白(MBP)和嗜酸细胞源性神径毒素(EDN)mRNA表达丰度,但机制尚不明确[11,12]。在小鼠红系分化末期可检测到促进红细胞存活的长链基因间非编码RNA(lncRNA-EPS)通过抑制促凋亡基因Pycard发挥作用[13]。Hu等[14]在EPO孵化3 d鼠胎肝红系祖细胞成熟的过程中,通过shRNA下调40%~50% lncRNA-EPS,发现红细胞聚集受抑,大量凋亡,分化受阻;在生理水平异位表达lincRNA-EPS可阻止由于EPO缺失导致的红系祖细胞凋亡。有研究表明,鼠类红系中超过80%的lncRNA,甚至高表达的lncRNA在人类红系类似发育阶段均未发现,同时人类红系高表达的lncRNA在小鼠中也未发现,这可能是物种进化的结果,进一步提示lncRNA在高等物种中起重要调控作用[15]。
PML/RARα融合基因是急性早幼粒细胞白血病的特异性标志,PML/RARα可以抑制NEAT1(一个核限制型lncRNA,在NB4中高表达,与细胞分化有关)[16]。NEAT1和DBHS蛋白家族可构成染色体区间的亚细胞核结构体Paraspeckles,这种结构复杂的RNA-蛋白复合体在多种细胞生命活动过程如应激反应、细胞分化和病毒感染中调控基因表达,参与生理、病理过程[17]。lncRNA母素表达基因(MEG3)定位于染色体14q32.2,长度约1.6 kb,与多种肿瘤有关,通过p53直接或间接发挥抑癌作用[18]。Benetatos等[19]检测85例AML和骨髓增生异常综合征(MDS)患者lncRNA MEG3表达情况,发现近一半的AML患者MEG3启动子异常超甲基化,这与患者总生存率降低有关。有研究在CML细胞系K562中发现了与bcr-abl转录有关的lncRNA,定义为lncRNA beta globin locus 3(βGL3),它可以正向调控肿瘤抑制因子PTEN的作用,降低K562细胞的存活率,而且可以被酪氨酸激酶抑制剂(如伊马替尼)诱导凋亡[20]。lncRNA(UCA1)与AML CEBPA突变有关,下调UCA1可以影响AML细胞增殖[21]。另外,lncRNA还可调控与AML预后有关的WT1基因[22]。
在雌性哺乳动物胚胎发育早期,其中一条X染色体失活,X染色体失活中心(Xic)可转录多种非编码RNA,其中包括(Xist)。敲除雌性小鼠HSC上的Xist后导致较高丰度的X连锁基因,这些小鼠高风险转化为骨髓增殖性肿瘤(MPN)和MDS。野生型小鼠植入Xist-/-的骨髓可出现MPN/MDS,而Xist-/-小鼠植入野生型骨髓则不会出现,所以,Xist-/-提示HSC成熟受损以及缺少长期HSC。这些作用都依赖于广泛的X基因活化导致基因层面的改变,包括上调或下调与MPN和MDS有关的癌基因或抑癌基因,所以Xist功能丢失可能会导致血液肿瘤[23]。同时Xist在白血病中也有表达,但具体机制尚不清楚[24]。
lncRNA与机体免疫细胞成熟、分化也密切相关[25]。活化T细胞核因子(NFAT)最初作为在活化T细胞IL-2启动子上结合的转录因子而被发现,在免疫反应中对诱导基因转录起重要作用,在多种免疫细胞中表达。NFAT的一个关键调节子是NFAT的非编码阻抑物(NRON),长约0.8~3.7 kb[26]。RNAi T细胞系细胞株(Jurkat T细胞)中的NRON以及缺失支架蛋白IQGAP-1的小鼠T细胞均表现出NFAT依赖的细胞因子增加[27,28]。胸腺特异性非编码RNA-1(Thy-ncR1)也是与T细胞成熟有关的lncRNA,位于细胞质内,通过抑制微纤丝相关糖蛋白4(MFAP4)mRNA起作用[29]。在Jurkat细胞系中,MFAP4 mRNA的稳定性与hUPFI依赖通路有关。另一种与T细胞分化有关的lncRNA是NeST,位置紧邻编码可以促进T细胞分化干扰素γ(IFN-γ)基因IFNG,而且通过改变IFNG座位组蛋白甲基化来刺激IFNG翻译,实现对宿主Theiler病毒和沙门菌感染敏感性的调节[30]。另外,在幼稚CD8+T细胞分化为各种辅助性T细胞亚群的过程中,定义了一种通过GATA3活化的Th2细胞特异性基因的转录因子LncR-Ccr2-5'AS[31],但具体机制尚不清楚。
通过分析单核细胞分化为树突状细胞(DC)的过程发现,DC分化所必需的lnc-DC存在于细胞质中,通过与激活转录因子STAT3直接结合发挥作用[32]。另一种lncRNA-Cox2可以明显上调鼠骨髓来源DC和巨噬细胞Toll样受体(TLR)信号,在炎症信号中通过调控复合物的相互作用调节一些免疫应答基因表达,还可通过与hnRNP-A/B及hnRNP-A2/B1的相互作用下调不同肿瘤免疫基因表达[33]。
急性白血病INK4b-ARF-INK4α位点通常缺失或高度甲基化[34]。INK4位点反义非编码RNA NRIL全长34.8 kb,定位于人染色体9p21上。Yu等[35]研究显示急性淋巴细胞白血病(ALL)患者中ANRIL表达增高。Iacobucci等[36]研究美国费城人群中ANRIL基因多态性与ALL的相关性,认为CDKN2B-AS rs564398位点多态性有利于转录本的改变,使得CDKN2A/B表型发生变异,增加ALL发病风险。INK4b为Rb通路的一部分,通过表达细胞周期抑制因子致癌,还以表观遗传沉默方式调控肿瘤抑制基因p15,促进肿瘤发生。ANRIL的作用机制与HOTAIR相似,可作为一个分子支架招募多个染色质修饰复合体PRC1和PRC2,ANRIL/PRC1介导INK4b-ARF-INK4α位点沉默,动态调控基因转录活性[37]。在T-ALL发病机制中还与突变激活NOTCH信号有关[38]。lncRNA LUNAR1是受到NOTCH信号调控的原癌lncRNA,可以通过增强并维持IGF1R mRNA表达从而促进T-ALL细胞增殖[39]。在B-ALL中,lncRNA BARL-2(B-ALL-associated long RNAs-2)被认为是与糖皮质激素治疗反应有关的lncRNA[40]。
lncRNA-p21(TP53COR1,tumor protein p53 pathway corepressor 1)是细胞周期依赖性蛋白激酶抑制物家族中的重要成员——p21蛋白基因间转录产物,定位于在CDKN1A基因上游,长约3.1 kb,其表达直接受p53基因调控,以p53依赖方式诱导细胞凋亡。在慢性粒细胞白血病(CLL)中,lncRNA-p21表达异常[41,42]。另外,大约有50%的B-CLL和套细胞淋巴瘤患者存在13q14.3的缺失,表明在这个区域定位着肿瘤抑制基因,此基因参与了这些疾病的发生。在这个400 kb缺失的区域里,有两个位置相邻的亚区,分别为DLEU1和DLEU2,其具体致病机制尚未明确[43]。Wolf等[44]认为DLEU1是B细胞特定肿瘤相关基因(BCMS)的一部分,其至少有50个外显子。通过RNA序列分析发现BCMS转录物属于lncRNA类,所以BCMS是13q14中涉及B-CLL生成最有可能的肿瘤抑制基因。
有关上述lncRNA的总结见表1。总之,lncRNA可在多个层面调控血细胞的成熟、分化。目前,与血细胞分化、成熟及恶变有关的lncRNA逐步被发现,而且与疾病的诊断及预后判断相关,但只有少部分lncRNA被标记和注释,所以还需要开展大量的鉴定和功能分析工作。利用高通量基因测序、基因芯片及生物信息学技术,结合分子生物学、细胞生物学、蛋白质组学及动物实验,从lncRNA碱基序列及二维、三维立体空间结构出发,进一步深入研究lncRNA在血液系统疾病发生、发展过程中的调控作用及其机制,以期为寻找血液病治疗靶点提供新思路。
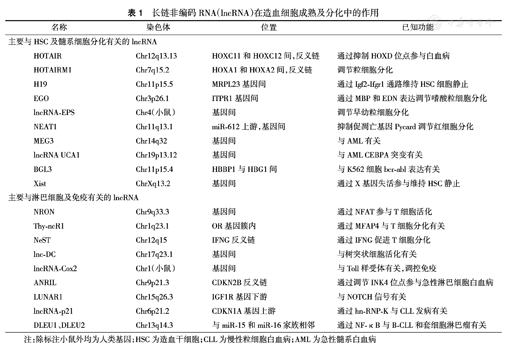
长链非编码RNA(lncRNA)在造血细胞成熟及分化中的作用
长链非编码RNA(lncRNA)在造血细胞成熟及分化中的作用
| 名称 | 染色体 | 位置 | 已知功能 | |
|---|---|---|---|---|
| 主要与HSC及髓系细胞分化有关的lncRNA | ||||
| HOTAIR | Chr12q13.13 | HOXC11和HOXC12间,反义链 | 通过抑制HOXD位点参与白血病 | |
| HOTAIRM1 | Chr7q15.2 | HOXA1和HOXA2间,反义链 | 调节粒细胞分化 | |
| H19 | Chr11p15.5 | MRPL23基因间 | 通过Igf2-Ifgr1通路维持HSC细胞静止 | |
| EGO | Chr3p26.1 | ITPR1基因间 | 通过MBP和EDN表达调节嗜酸粒细胞分化 | |
| lncRNA-EPS | Chr4(小鼠) | 基因间 | 调节早幼粒细胞分化 | |
| NEAT1 | Chr11q13.1 | miR-612上游,基因间 | 抑制促凋亡基因Pycard调节红细胞分化 | |
| MEG3 | Chr14q32 | 基因间 | 与AML有关 | |
| lncRNA UCA1 | Chr19p13.12 | 基因间 | 与AML CEBPA突变有关 | |
| BGL3 | Chr11p15.4 | HBBP1与HBG1间 | 与K562细胞bcr-abl表达有关 | |
| Xist | ChrXq13.2 | 基因间 | 通过X基因失活参与维持HSC静止 | |
| 主要与淋巴细胞及免疫有关的lncRNA | ||||
| NRON | Chr9q33.3 | 基因间 | 通过NFAT参与T细胞活化 | |
| Thy-ncR1 | Chr1q23.1 | OR基因簇内 | 通过MFAP4与T细胞分化有关 | |
| NeST | Chr12q15 | IFNG反义链 | 通过IFNG促进T细胞分化 | |
| lnc-DC | Chr17q23.1 | 基因间 | 与树突状细胞活化有关 | |
| lncRNA-Cox2 | Chr1(小鼠) | 基因间 | 与Toll样受体有关,调控免疫 | |
| ANRIL | Chr9p21.3 | CDKN2B反义链 | 通过调节INK4位点参与急性淋巴细胞白血病 | |
| LUNAR1 | Chr15q26.3 | IGF1R基因下游 | 与NOTCH信号有关 | |
| lncRNA-p21 | Chr6p21.2 | CDKN1A基因上游 | 通过hn-RNP-K与CLL发病有关 | |
| DLEU1、DLEU2 | Chr13q14.3 | 与miR-15和miR-16家族相邻 | 通过NF-κB与B-CLL和套细胞淋巴瘤有关 | |
注:除标注小鼠外均为人类基因;HSC为造血干细胞;CLL为慢性粒细胞白血病;AML为急性髓系白血病
无



























