
探讨计算生物组学模型(CBM)在难治性急性髓系白血病(AML)患者治疗中的价值。
回顾性分析2018年11月收治的1例采用由Cellworks肿瘤治疗应答指数(TRI)检测预测的个性化治疗方案治疗的难治性AML患者临床资料,总结诊疗过程及治疗效果,并复习AML治疗中CBM应用的相关文献。
患者为43岁女性,伴t(6;11)(q27;q23),确诊后经2次诱导治疗均失败且化疗耐受性差。2019年3月行Cellworks TRI检测,预测最佳方案为克拉屈滨+曲美替尼+阿糖胞苷三药联合方案。患者采用该方案治疗1个疗程后复查达到完全缓解且微小残留病阴性,后成功实施亲源半相合异基因造血干细胞移植术。2020年11月疾病复发,无病生存19个月;2021年4月死亡,总生存时间为28.5个月。
基于CBM的TRI检测为难治性AML患者提供了新的治疗思路,依据基因组学预测出的个性化治疗建议可能改善患者生存。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
急性髓系白血病(AML)是一种起源于髓系干祖细胞的血液系统恶性肿瘤,难治性AML患者预后较差,不适合进行强化挽救性化疗的患者中位总生存(OS)时间仅为4~9个月[1]。伴t(6;11)AML有独特的临床特征,易复发,预后差,2年OS率为13%,中位OS时间为6~12个月[2,3]。高危AML患者的生存不能从传统方案中获益,随着靶向药物和蛋白基因组学新技术的问世,如何更好地进行精准化、个体化治疗显得尤为重要[4]。肿瘤治疗应答指数(TRI)检测依托于计算机生物模拟和人工智能技术,运用患者全外显子信息建立个性化疾病模型,生成一个特异性的蛋白网络图,基于表型作用对药物组合的疗效进行排序,进而确定理想疗法。现报道1例采用由Cellworks TRI检测预测的个性化建议治疗的难治性AML患者临床资料,并复习相关文献,旨在为难治性AML治疗提供参考。
患者,女性,43岁,2018年11月因月经量增多伴全血细胞减少2个月就诊。血常规:白细胞计数2.8×109/L,血红蛋白58 g/L,血小板计数15×109/L,中性粒细胞数0.8×109/L,淋巴细胞数1.4×109/L,单核细胞数0.6×109/L,幼稚细胞占0.08。骨髓涂片:原始粒细胞及早幼粒细胞占0.12,原始单核细胞及幼稚单核细胞占0.21。骨髓活组织检查:骨髓造血组织增生明显活跃,幼稚细胞比例增高(占0.05~0.10),巨核细胞增多,可见微小、单圆、多圆巨核细胞。骨髓染色体:46,XX,t(6;11)(q27;q23)[20]。骨髓流式细胞术检测:CD117阳性细胞占有核细胞的9%;CD34部分阳性,CD117、HLA-DR、CD13、CD33均阳性,CD19、CD56、CD3、CD5、CD7均阴性;粒细胞相对比例降低,可见其免疫表型CD13、CD16、CD15、CD11b表达紊乱。诊断为AML-M4,伴t(6;11),AML可能由骨髓增生异常综合征(MDS)转变而来。2018年12月11日行首次诱导治疗,具体方案:地西他滨30 mg/d,第1天至第3天;阿糖胞苷每12 h 25 mg,第4天至第14天;伊达比星10 mg/d,第4天至第5天;粒细胞集落刺激因子(G-CSF)300 μg/d,第3天至第10天(白细胞计数>20×109/L时停用)。化疗后出现重度骨髓抑制,粒细胞缺乏期6 d,血象恢复过程中白细胞数极速增加,2018年12月30日复查血常规:白细胞计数135×109/L,血红蛋白57 g/L,血小板计数41×109/L,幼稚细胞占0.86。评估疗效为未缓解。2019年1月6日行第2次诱导治疗,具体方案:阿扎胞苷100 mg/d,第1天至第5天;阿柔比星40 mg/d第1天,20 mg/d第2天至第5天;高三尖杉酯碱2 mg/d,第6、8、10天;依托泊苷50 mg/d第7、9、11天;阿糖胞苷50 mg/d第6天至第11天;G-CSF 300 μg/d第6天至第11天(白细胞计数>20×109/L时停用)。化疗后再次出现重度骨髓抑制,并合并肺部感染,粒细胞缺乏期12 d;骨髓涂片显示有核细胞增生显著活跃,单核细胞占0.43,白血病细胞占0.39;外周血幼稚细胞占0.15。评估疗效仍为未缓解。因此,患者为难治性AML,且一般状况差,难以耐受强化疗。
为选用更合适的治疗方案,经患者及家属同意,于2019年3月6日抽取患者骨髓和外周血标本进行Cellworks TRI检测,预测最佳方案为克拉屈滨+曲美替尼+阿糖胞苷三药联合方案。2019年4月1日开始采用该三药联合方案:克拉屈滨7.8 mg/d,第1天至第5天,静脉滴注;阿糖胞苷每12 h 20 mg,第1天至第10天,静脉滴注;曲美替尼2 mg/d,第1天至第14天,口服。治疗第5天患者发生重度骨髓抑制,进入粒细胞缺乏期,随后出现高热、黑便、全身水肿,体质量增加8 kg。粒细胞缺乏期19 d,在积极行升白细胞、抗感染、输血、利尿等对症支持治疗下血象恢复,病情好转。2019年5月8日复查骨髓提示完全缓解(CR),骨髓微小残留病阴性。2019年6月2日接受亲源半相合异基因造血干细胞移植术。2019年7月26日复查骨髓示CR,嵌合度99.9%;血常规:白细胞计数3.4× 109/L,血红蛋白68 g/L,血小板计数66×109/L,中性粒细胞数1.7×109/L,单核细胞数0.8×109/L。2020年11月疾病复发,无病生存19个月。复发后分别给予长效干扰素、阿扎胞苷、维奈克拉联合阿糖胞苷治疗均无效。2021年4月因病情进展死亡,OS时间为28.5个月。
首先将本例患者的二代测序(NGS)报告和存在的KMT2A-AFDN(即MLL-AF6)融合基因输入TRI生物模拟平台。患者NGS报告中显示观察到52个突变,关键突变及其影响如下:BRAF V600E、KMT2C、ERCC6和TP53的致病性错义突变,ERCC2和KMT2D的移码突变;KMT2C和KMT2D的缺失通过H3K4甲基化导致DNA错配修复(MMR)信号通路的下调[5,6,7,8];由于BRAF有害的错义突变,细胞外信号相关激酶(ERK)信号通路上调[9]。预测到克拉屈滨和曲美替尼联合的推荐方案应答指数最佳,而经模拟预测显示,加入低剂量阿糖胞苷的三药联合推荐方案具有协同作用且应答良好。推荐方案所依据的分子基因层级的发病机制(图1):(1)曲美替尼是一种可逆的MEK1和MEK2活化的变构抑制剂,MEK1和MEK2是ERK信号通路的上游调控因子,促进细胞增殖。BRAF突变导致的ERK信号通路上调可使患者对曲美替尼敏感[10]。(2)阿糖胞苷是嘧啶类似物,在复制过程中掺入DNA中,干扰DNA合成,导致单链断裂,最终使细胞凋亡[11]。MMR信号通路下调可使患者对阿糖胞苷敏感[12]。(3)克拉屈滨是一种嘌呤类似物,它被脱氧腺苷激酶(dCK)磷酸化为活性形式2CdATP,然后掺入DNA中,干扰DNA合成,导致单链断裂,最终导致细胞凋亡[13]。克拉屈滨还能抑制核糖核酸还原酶,导致白血病细胞中三磷酸阿糖胞苷积聚,增加了阿糖胞苷联合克拉屈滨的疗效[14,15]。
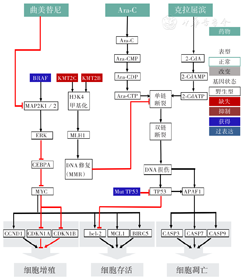

注:TRI为肿瘤治疗应答指数;MMR为错配修复;Ara-C为阿糖胞苷;Ara-CMP为单磷酸阿糖胞苷;Ara-CDP为二磷酸阿糖胞苷;Ara-CTP为三磷酸阿糖胞苷;2-CdA为二氯脱氧腺苷;2-CdAMP为二氯脱氧腺苷二磷酸;2-CdATP为二氯脱氧腺苷三磷酸;在TRI模拟中,克拉屈滨、曲美替尼和阿糖胞苷联合用药使肿瘤细胞增殖和存活显著降低
21世纪以来,随着表观遗传学及生物技术的发展,AML逐步走向精准治疗时代。当前通过NGS、蛋白质组学等组学技术进行的精准诊疗也逐渐被重视。本例患者在TRI检测中筛选出关键性的BRAF V600E突变和KMT2C/KMT2D缺失。BRAF突变导致RAS-RAF-MEK-ERK信号通路激活,促进了细胞增殖、分裂。KMT2C和KMT2D缺失导致DNA MMR信号通路下调,促进了肿瘤的发生。TRI预测有效的三药方案中,曲美替尼抑制MEK1和MEK2活化,可抑制细胞增殖分裂;克拉屈滨和阿糖胞苷可干扰DNA合成,两者具有协同促细胞凋亡作用;三药最大限度地达到了精准治疗作用,最终改变了患者的临床结局。
计算生物组学模型(CBM)技术是基于目前已发表的数万篇文献,利用人工智能在分子水平上模拟了人类肿瘤细胞的分子生理学过程,可通过输入患者个体的变异基因信息,绘制特异性的蛋白网络图,预测药物的敏感性[16]。既往研究表明,伴有MYD88突变的RPCI-WM1细胞中突变导致ERK信号激活,CBM成功模拟ERK信号激活的过程,并预测ERK上游信号MEK分子抑制剂能有效抑制RPCI-WM1细胞的增殖和活力,该细胞株的体外药物实验验证了预测的准确性[17]。本例患者检出了在AML中发生率约为1%的BRAF突变,该突变导致ERK信号激活,基于建立的数据模型预测MEK分子抑制剂有效,采用含曲美替尼方案治疗后患者首次达到CR。
作为新兴的人工智能技术,CBM相关的临床研究正在开展(表1)[18,19,20,21,22,23,24,25,26,27,28,29,30,31,32,33,34],对其准确性的验证尤为重要,众多研究也紧紧围绕这个主题而展开。回顾性总结Cellworks数据库1 517例AML患者的数据,统计分析显示CBM预测临床疗效的阳性预测值为92%,阴性预测值为79%,灵敏度为84%,特异度为89%,准确率为86%,CBM的准确率明显高于对照组(临床医师)的准确率(61%),提示CBM可为临床决策提供帮助。值得关注的一项较为重要的iCare1前瞻性研究报道了50例诊断为AML和MDS患者采用CBM评估61种治疗方案的反应,结果显示真阳性33例,真阴性22例,假阳性3例,假阴性3例,阳性预测值92%,阴性预测值88%,灵敏度92%,特异度88%,准确率90%[27]。提示CBM在AML和MDS中有较高的临床预测价值。预测方案最终的目标是用于临床,本例患者直接采用CBM预测推荐的方案治疗,取得缓解并生存获益。
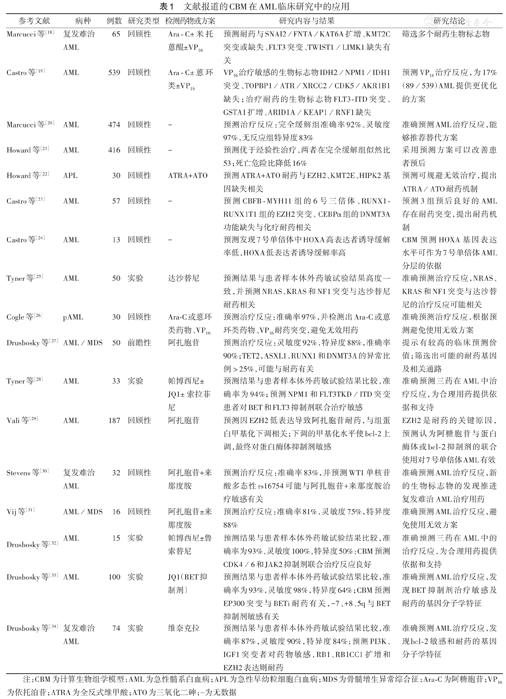
文献报道的CBM在AML临床研究中的应用
文献报道的CBM在AML临床研究中的应用
| 参考文献 | 病种 | 例数 | 研究类型 | 检测药物或方案 | 研究内容与结果 | 研究结论 |
|---|---|---|---|---|---|---|
| Marcucci等[18] | 复发难治AML | 65 | 回顾性 | Ara-C±米托蒽醌±VP16 | 预测耐药与SNAI2/FNTA/KAT6A扩增、KMT2C突变或缺失、FLT3突变、TWIST1/LIMK1缺失有关 | 筛选多个耐药生物标志物 |
| Castro等[19] | AML | 539 | 回顾性 | Ara-C±蒽环类±VP16 | VP16治疗敏感的生物标志物IDH2/NPM1/IDH1突变、TOPBP1/ATR/XRCC2/CDK5/AKR1B1缺失;治疗耐药的生物标志物FLT3-ITD突变、GSTA1扩增、ARID1A/KEAP1/RNF1缺失 | 预测VP16治疗反应,为17%(89/539)AML提供更优化的方案 |
| Marcucci等[20] | AML | 474 | 回顾性 | - | 预测治疗反应:完全缓解组准确率92%、灵敏度97%,无反应组特异度83% | 准确预测AML治疗反应,能够推荐替代方案 |
| Howard等[21] | AML | 416 | 回顾性 | - | 预测优于经验性治疗,两者在完全缓解组似然比53;死亡危险比降低16% | 采用预测方案可以改善患者预后 |
| Howard等[22] | APL | 30 | 回顾性 | ATRA+ATO | 预测ATRA+ATO耐药与EZH2、KMT2E、HIPK2基因缺失相关 | 预测可规避无效治疗,提出ATRA/ATO耐药机制 |
| Castro等[23] | AML | 57 | 回顾性 | - | 预测CBFB-MYH11组的6号三倍体、RUNX1-RUNX1T1组的EZH2突变、CEBPα组的DNMT3A功能缺失与化疗耐药相关 | 预测3组预后良好的AML存在耐药突变,提出耐药机制 |
| Castro等[24] | AML | 13 | 回顾性 | - | 预测发现7号单倍体中HOXA高表达者诱导缓解率低,HOXA低表达者诱导缓解率高 | CBM预测HOXA基因表达水平可作为7号单倍体AML分层的依据 |
| Tyner等[25] | AML | 50 | 实验 | 达沙替尼 | 预测结果与患者样本体外药敏试验结果高度一致,并预测NRAS、KRAS和NF1突变与达沙替尼耐药相关 | 准确预测治疗反应,NRAS、KRAS和NF1突变与达沙替尼的治疗反应可能相关 |
| Cogle等[26] | pAML | 30 | 回顾性 | Ara-C或蒽环类药物、VP16 | 预测治疗反应:准确率97%,并检测出Ara-C或蒽环类药物、VP16耐药突变,避免无效用药 | 准确预测治疗反应,根据预测避免使用无效方案 |
| Drusbosky等[27] | AML/MDS | 50 | 前瞻性 | 阿扎胞苷 | 预测治疗反应:灵敏度92%,特异度88%,准确率90%;TET2,ASXL1,RUNX1和DNMT3A的异常比例>25%,可能与耐药有关 | 提示有较高的临床预测价值;筛选出可能的耐药基因及相关通路 |
| Tyner等[28] | AML | 33 | 实验 | 帕博西尼±JQ1±索拉菲尼 | 预测结果与患者样本体外药敏试验结果比较,准确率为94%;预测NPM1和FLT3TKD/ITD突变患者对BET和FLT3抑制剂联合治疗敏感 | 准确预测三药在AML中治疗反应,为合理用药提供依据和支持 |
| Vali等[29] | AML | 187 | 回顾性 | 阿扎胞苷 | 预测因EZH2低表达导致阿扎胞苷耐药,与组蛋白甲基化下调相关;下调的甲基化水平使bcl-2上调,最终对蛋白酶体抑制剂敏感 | EZH2是耐药的关键原因,预测认为阿糖胞苷与蛋白酶体或bcl-2抑制剂的联合使用对7号单倍体AML有效 |
| Stevens等[30] | 复发难治AML | 32 | 回顾性 | 阿扎胞苷+来那度胺 | 预测治疗反应:准确率83%,并预测WT1单核苷酸多态性rs16754可能与阿扎胞苷+来那度胺治疗敏感有关 | 准确预测AML治疗反应,新的生物标志物的发现推进复发难治AML治疗用药 |
| Vij等[31] | AML/MDS | 16 | 回顾性 | 阿扎胞苷±来那度胺 | 预测治疗反应:准确率81%,灵敏度75%,特异度88% | 准确预测AML治疗反应,避免使用无效方案 |
| Drusbosky等[32] | AML | 15 | 实验 | 帕博西尼±鲁索替尼 | 预测结果与患者样本体外药敏试验结果比较,准确率为93%,灵敏度100%,特异度50%;CBM预测CDK4/6和JAK2抑制剂联合治疗反应良好 | 准确预测三药在AML中的治疗反应,为合理用药提供依据和支持 |
| Drusbosky等[33] | AML | 100 | 实验 | JQ1(BET抑制剂) | 预测结果与患者样本体外药敏试验结果比较,准确率为93%,灵敏度98%,特异度64%;CBM预测EP300突变与BETi耐药有关,-7、+8、5q与BET抑制剂敏感有关 | 准确预测AML治疗反应,发现BET抑制剂治疗敏感及耐药的基因分子学特征 |
| Drusbosky等[34] | 复发难治AML | 74 | 实验 | 维奈克拉 | 预测结果与患者样本体外药敏试验结果比较,准确率87%,灵敏度90%,特异度84%;预测PI3K、IGF1突变者对药物敏感,RB1、RB1CC1扩增和EZH2表达则耐药 | 准确预测AML治疗反应,发现bcl-2敏感和耐药的基因分子学特征 |
注:CBM为计算生物组学模型;AML为急性髓系白血病;APL为急性早幼粒细胞白血病;MDS为骨髓增生异常综合征;Ara-C为阿糖胞苷;VP16为依托泊苷;ATRA为全反式维甲酸;ATO为三氧化二砷;-为无数据
CBM在耐药机制探讨和药物作用靶点的筛选方面也有独特优势。有研究者采用CBM回顾性分析了187例伴7号单倍体AML基因组数据,发现EZH2是耐药的关键原因,并预测阿糖胞苷与蛋白酶体或bcl-2抑制剂的联合使用对7号单倍体AML有效[29]。另一项研究中采用CBM分析了13例7号单倍体AML基因组数据,发现HOXA活性低是7号单倍体AML治疗缓解率高的基因特征[24]。因此对于7号单倍体AML患者,可采用HOXA进行分类,为制订治疗计划提供了依据,可规避决策风险及不必要的治疗相关不良反应。有研究者采用CBM分析了65例采用MEC(米托蒽醌+依托泊苷+阿糖胞苷)方案治疗的复发难治AML患者基因组数据,发现了与治疗反应相关的众多生物标志物[18],为筛选关键作用靶标提供了依据。CBM令人瞩目,但仍面临疾病模型病种不全面、药品获取困难、超适应证用药、费用昂贵等问题,影响了其在我国的进一步推广和应用。
难治性AML有着明显的异质性,2年OS率为29%,暂无统一的标准治疗方案[35]。CBM基于个体的基因信息,可准确预测出最佳个体治疗方案,为难治性AML治疗提供了新的思路,可最大限度地实现个体化精准治疗。随着更多耐药机制的探明以及药物作用靶点的发现,难治性AML患者的临床结局有望得到进一步改善。
所有作者均声明不存在利益冲突



























