
在新冠病毒当前疫情应对临床场景中,快速准确核酸检测对疫情防控至关重要。随着对新型冠状病毒的认识深入,对临床感染病例经验积累,新型冠状病毒核酸检测方法已有快速进展。本文对新型冠状病毒核酸检测方法临床应用情况做一概述。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
新型冠状病毒(SARS-CoV-2)为单股正链RNA病毒,经呼吸道传播可以引起新型冠状病毒肺炎(COVID-19),目前已经在世界范围内发生大流行[1,2,3]。基因组测序显示,新冠病毒与蝙蝠体内检测到的冠状病毒高度同源,病毒全基因组序列一致性为88%[4]。新冠病毒传染力极强,主要通过飞沫传播与密切接触传播。截至2020年4月10日,全球已有211个国家或地区出现新型冠状病毒确诊病例。我国制定抗击疫情策略"应收尽收,应检尽检,应治尽治",其目的在于尽量发现传染源,减少漏诊病例,保护人群安全。在历次国家颁布的诊疗方案中,新冠病毒核酸检测均作为诊断的"金标准";在此背景下,各类新冠病毒检测试剂迅速研发、经过审批后应用于疫情防控。体外定性检测新冠病毒感染可检测新冠病毒核酸、新冠病毒抗体。截至目前,国家药品监督管理局(NMPA)批准新冠病毒核酸检测试剂共计18种,新冠病毒核酸检测为确诊首选检测方法,而抗体检测一般用于补充检测(与核酸检测协同使用)或回顾性血清学调查。核酸检测使用的标本类型包括鼻咽拭子等上呼吸道标本,肺泡灌洗液等下呼吸道标本,以及其他血液,体液,粪便标本。核酸检测提供了精准、有效的诊断。下文现就新冠病毒核酸检测的各类技术原理及质量控制作综合阐述。
通过对基因序列的分析,SARS-CoV-2基因的保守序列包括开放阅读框架1ab(open reading frame 1ab,ORF1ab)基因,核衣壳蛋白(nucleocapsid protein,N)基因,包膜蛋白(envelope,E)基因,棘突蛋白(spike,S)基因和膜蛋白(membrane,M)基因等[5,6](图1)。ORF1ab编码16种非结构蛋白,下游包括至少12个ORF[5]。目前临床上使用的荧光PCR法新冠病毒核酸检测试剂主要包括单靶标(ORF1ab)、双靶标(ORF1ab和N,ORF1ab和E)和三靶标(ORF1ab、N和E)三种类型。
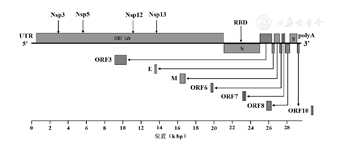

注:保守序列包括ORF1ab(开放阅读框架1ab),S(棘突蛋白),E(包膜蛋白),M(膜蛋白)和N(核衣壳蛋白)基因等。ORF1ab基因区域所编码的非结构蛋白(Nsp)包括Nsp3(木瓜蛋白酶),Nsp5(胰凝乳蛋白酶),Nsp12(RNA依赖的RNA聚合酶,RdRp)和Nsp13(解旋酶)等共16种。
新冠病毒的ORF1ab基因区域长度为21 291 nT,可以编码16种非结构蛋白(non-structural proteins,Nsps)[5],包括两种病毒半胱氨酸蛋白酶,Nsp3(木瓜蛋白酶)和Nsp5(胰凝乳蛋白酶),Nsp12(RNA依赖的RNA聚合酶,RdRp)和Nsp13(解旋酶)等[7]。其中RdRp基因所编码的RNA聚合酶参与病毒核酸的复制[4]。
通过对COVID-19患者不同部位标本进行病毒核酸检测,发现对于ORF1ab基因,ddPCR的拷贝数和RT-PCR的Ct值高度相关(R2 = 0.83)[8]。Yan等[9]的研究发现,采用环介导的等温扩增法对新冠病毒的ORF1ab和S基因进行扩增,最适温度为63 ℃,灵敏度分别为20 copies/reaction(ORF1ab)和200 copies/reaction(S)。经130例疑似COVID-19患者进行验证,58例患者被证明阳性,72例被证明阴性,灵敏度和特异度均为100%。
N基因表达的核衣壳蛋白,可以与病毒的RNA结合形成病毒的核心,同时也是病毒复制与转录的重要蛋白元件[10,11]。Yu等[8]对比分析ddPCR和RT-PCR法定量检测新冠病毒核酸,对于N基因,ddPCR的拷贝数和RT-PCR的Ct值显示出高度相关(R2 = 0.87)。感染早期由于病毒基因的复制和转录,N蛋白表达增加,N基因的核酸载量也会增加,故以N基因为靶标检测新冠病毒有助于COVID-19的早期诊断。同时通过在血清水平对抗体的检测,验证N蛋白抗体在轻度感染患者中具有更高的灵敏度[12]。
包膜蛋白由病毒的E基因表达。E蛋白是冠状病毒结构中完整的膜蛋白,也是病毒中最小的结构蛋白[13]。当病毒处于复制周期时,E蛋白在宿主细胞内大量表达,然而只有小部分参与构成了病毒体的膜蛋白,大部分E蛋白保留在细胞内的内质网、高尔基体和内质网-高尔基体中间体(ERGIC),参与病毒的组装和出芽[14,15]。
Nalla等[16]通过对7种不同的新冠病毒核酸探针/引物和1种核酸检测试剂盒分析,发现所使用的E基因和N2基因探针/引物最敏感,且所有测试方法均对新冠病毒具有高度特异性,无论使用何种引物/探针或试剂盒,均未发现与其他呼吸道病毒有交叉反应。
除了以上主要检测三种靶基因(荧光PCR法),也可以通过恒温扩增芯片法对S基因进行检测。S基因编码棘突(S)蛋白,SARS-CoV可以借助于S蛋白的细胞受体血管紧张素转换酶2(ACE2),从而进入宿主细胞[17]。经序列对比分析,新冠病毒的S基因序列与蝙蝠来源的冠状病毒(Bat-SL-CoVZXC21和Bat-SL-CoVZC45)同源性较高,分别为75.2%和74.7%,而与SARS-CoV GZ02的S基因序列同源性相对较低(72.7%)[4]。另一项研究通过SWISS-MODEL对新冠病毒、蝙蝠SARS样-CoV(Rs4874和Rp3)和SARS-CoV的S蛋白的受体结合结构域(RBD)进行了蛋白同源性模拟分析,预测发现SARS-CoV-2和Rs4874的RBD结构与SARS-CoV相似,而与Rp3存在差异[5]。另外,相对于其他人类冠状病毒(OC43和HKU1),新冠病毒的S蛋白N端与SARS-CoV的S蛋白N端更相似[5]。最新的研究显示,SARS-CoV-2与SARS-CoV可以使用相同的细胞受体ACE2进入宿主细胞[18,19],且SARS-CoV-2与ACE2结合的亲和力是SARS-CoV与ACE2结合亲和力的10-20倍[20]。综上所述,SARS-CoV-2通过与ACE2结合而进入宿主细胞,从而增加人与人之间的传播。值得注意的是多重检测芯片不仅可以检测SARS-CoV-2的S和N基因,同时也可以对甲型流感病毒、新型甲型H1N1流感病毒、甲型H3N2流感病毒、乙型流感病毒和呼吸道合胞病毒的核酸进行检测,对COVID-19轻型和其他呼吸道病毒引起的肺炎进行一次检测,多种病原鉴别诊断。
荧光PCR技术是检测新冠病毒核酸的推荐技术,灵敏度较高,重复性较好,但用时较长。除荧光PCR技术外,新冠病毒检测方法还包括数字PCR技术、测序技术,等温扩增技术等。各项检测方法技术原理及特点详见表1。

新冠病毒核酸检测方法
新冠病毒核酸检测方法
| 方法 | 技术原理 | 特点 |
|---|---|---|
| 荧光PCR法 | 实时荧光PCR(real-time polymerase chain reaction, RT-PCR)设计特异性引物和TaqMan探针,对整个PCR过程实时监测,对新冠病毒靶基因进行定性检测。 | 灵敏度高,重复性好,但用时较长。 |
| 等温扩增法(实时荧光核酸等温扩增) | 实时荧光核酸等温扩增检测技术(Simultaneous Amplification and Testing,SAT)使用M-MLV反转录酶、T7 RNA聚合酶和优化探针来检测RNA。 | 高特异性,高灵敏度,反应稳定。 |
| 等温扩增法(环介导的等温扩增) | 环介导的等温扩增技术(loop-mediated isothermal amplification, LAMP)利用特异位点设计的4条引物[21]以及链置换DNA聚合酶进行扩增[22],2002年该技术被优化,加入两条环引物[23]使目的基因扩增速度加快,扩增效率得到提高。 | 灵敏度高,特异性强,成本低,操作简单。 |
| 等温扩增法(切口延伸等温扩增) | 美国的快速核酸检测ID NOW平台以切口延伸等温扩增技术(Niking Enzyme Amplification Reaction, NEAR)为基础来检测病毒核酸,检测仪中包括有扩增管、样品管、转移管等,等温扩增反应及结果读取由仪器完成。 | 体积较小,检测快,但重复性不好且通量低。 |
| 双扩增法 | RNA恒温扩增后经过多生物素信号放大,最后通过化学发光检测。 | 高通量,高效率。 |
| RNA恒温扩增-金探针层析法 | RNA恒温扩增后与特异探针杂交最后通过层析显色判定结果。 | 直接裂解样本,效率高,操作简单,设备要求低。 |
| RNA捕获探针法 | 特异靶标捕获法磁珠提取技术和实时荧光等温扩增检测技术相结合。 | 在同一反应管中完成核酸提取及扩增,可连续并行检测。 |
| 杂交捕获免疫荧光法 | 由杂交捕获和免疫荧光相结合的技术,即HC-IFA技术,通过探针捕获靶基因,应用免疫荧光技术来判定结果。 | 设备体积小,对单个样本检测用时更短。 |
| 联合探针锚定聚合测序法 | 配合测序系统使用的全基因组测序技术。 | 准确率高,成本高,时间长,不适用于临床检测。 |
| 数字PCR法 | 基于数字PCR系统设计优化相应的引物、探针、对照、内参基因等来检测新冠病毒核酸。 | 较荧光PCR技术灵敏度更高,具有高可靠性和高重复性,但用时更长,成本更高。 |
检验流程中有众多因素可以影响核酸检测的结果,因此要求检测人员遵照操作流程,在核酸检测前、中、后阶段做好核酸检测的质量控制。
检验前的质量控制,包括样本的采集、样本的转运和病毒灭活。研究发现患者下呼吸道样本的核酸定量检测和病毒载量分析有助于评估疾病进展,尤其是在病毒载量低的情况下[8,24]。但是需要注意采集下呼吸道标本易产生飞沫和气溶胶,增加医务人员的感染风险。
检验中的质量控制,包括核酸的提取、核酸的扩增和质控品的设置。为保证检测的可信度,每批检测样本都要设置严格的阳性质控和阴性质控,并根据所采用的检测方法,严格按照使用说明和实验室要求进行检测。
检测后质量控制,主要包括检测结果的正确判读和准确报告。对于结果的正确判读,要求检验人员对Ct值进行正确判定。
目前,临床上使用的SARS-CoV-2核酸检测试剂多使用多通道校正的全自动荧光PCR检测仪共同检测。采用荧光PCR法的核酸检测试剂,循环数设置一般为40~45个循环,Ct一般为37,若Ct < 37结果为阳性,Ct ≥ 40结果为阴性,若Ct为37~ 40,需进行复测确定。
对于目前临床上使用较多的双靶标SARS-CoV-2核酸检测试剂来说,根据WHO发布的COVID-19实验室检测技术指南[25],和中国疾病预防控制中心发布的《新型冠状病毒肺炎实验室检测技术指南(第六版)》[26]指出,在空白对照、阴性对照和阳性对照均满足实验要求时,双靶标(ORF1ab、N)均为阳性时,该样本报告阳性;双靶标均为阴性时,该样本报告为阴性。当单一靶基因为阳性时需要重新采样进行重新检测。两种标本核酸检测结果同时出现单靶标阳性或同种类型标本两次采样检测为单靶标阳性均可判定为阳性。
目前,临床中使用的SARS-CoV-2核酸检测方法以RT-PCR试剂为主,单靶标试剂因费用低主要用于不确定样本复测或已确诊病例随访检测;双靶标检测试剂应用较多,在筛查样本中需要注意两个基因靶标检测结果不一致的情况:如果同一份样本只有一个基因靶标出现阳性扩增曲线时需要参考其他品牌试剂检测结果或通知临床第二天继续采样再次送检复测。
数字PCR法检测核酸灵敏度高可以进行定量检测但是操作过程复杂结果回报时间长;等温扩增法检测核酸对扩增设备和检测设备要求低,但是其灵敏度尚缺乏多中心、大样本临床数据验证,所以在选择检测方法时需要结合自身使用目的、使用场所和条件。
核酸检测作为当前COVID-19诊断的"金标准",对临床病例识别和判断具有重要意义,然而检测流程中任何一个环节出现问题都可能导致假阴性结果出现而发生漏检;同时应该保证实验室环境和质控对照质量避免假阳性误报。因此按照标准检测流程操作、统一实验室结果判定标准对核酸检测结果的稳定性具有重要意义。
所有作者均声明不存在利益冲突



























