
新型冠状病毒(severe acute respiratory syndrome coronavirus 2,SARS-CoV-2)易发生变异。刺突(spike,S)蛋白是SARS-CoV-2与宿主细胞结合的重要结构,S蛋白的基因突变可能会改变病毒传染性、致病性、抗体应答等临床特征。世界卫生组织根据病毒的传染性、致病性和免疫逃逸能力,提出存疑变异株(variant of interest,VOI)和关切变异株(variant of concern,VOC)的分类。本文将对D614G变异株及流行的Alpha(B.1.1.7)、Beta(B.1.351)、Gamma(P.1)、Delta(B.1.617.2)和Epsilon(B.1.427/ B.1.429)变异株的最新研究进展进行概述。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
新型冠状病毒(severe acute respiratory syndrome coronavirus 2,SARS-CoV-2)是单股正链RNA病毒,直径60~140 nm,该病毒基因组约由29 903个核苷酸构成,编码4种结构蛋白:核蛋白(nucleocapsid,N)、包膜(envelop,E)、基质蛋白(membrane,M)、刺突蛋白(spike,S),以及RNA依赖性RNA聚合酶(RNA-dependent DNA polymerase,RdRp )[1,2]。S蛋白是重要的结构蛋白,其主要功能是通过受体结合结构域(receptor-binding domain,RBD)、N端结构域(N-Terminal RNA-binding domain,NTD)促进病毒包膜与宿主细胞的血管紧张素转换酶2(angiotensin converting enzyme 2,ACE2)结合,使宿主细胞和病毒融合。由于ACE2在广泛部位和器官(如呼吸道、心血管、胃肠道、肾脏等)高度表达[3],新型冠状病毒肺炎(coronavirus disease 2019,COVID-19)不仅能引起患者的呼吸系统症状外,还可引发肌痛、腹泻、嗅觉及味觉减退等症状,重症患者还可能发生血管血栓形成、凝血功能障碍等肺外并发症[1]。
SARS-CoV-2变异,尤其是S蛋白上的特定位点的突变,不仅能影响病毒与人体细胞的亲和力,改变病毒的传染性和致病性,S蛋白作为诱发机体产生细胞和体液免疫的重要成分还可影响机体免疫应答、疫苗的抗体转化和单克隆抗体的中和作用。目前SARS-CoV-2变异株不断出现,COVID-19流行仍在全球持续。只有了解流行变异株的临床特征,才能全面快速地控制疫情。总结SARS-CoV-2变异株常见的命名和分型方法及目前流行变异株的基本临床特征和研究进展,有助于更好地理解病毒的变异规律,启发控制流行和预防再流行的新思路。
冠状病毒相比于其他RNA病毒具有较高的复制保真性,预测SARS-CoV-2的突变速率约为10-6每位点每循环,比流感病毒慢3~4倍[4]。但自2019年12月至今(截至2021年7月15日),有超过2 367 530个SARS-CoV-2基因组序列在网络基因组共享平台公开,并且数量还在继续增长[5]。SARS-CoV-2新基因组生成速度超出其他冠状病毒的变异速度[6]。
在全球大流行背景下,研究者们分析病毒的突变位点、构建系统发育树,为分析变异规律、传播模式以及病毒溯源、确定病毒传播链等提供理论依据。这些工作不能离开网络基因组信息共享分析平台,如:全球共享流感数据库(global initiative on sharing all influenza data,GISAID)和基因组信息分析平台:Nextstrain网站、cov-lineages网站等。除了基因信息的及时共享,连续、被广泛接受的基因分型方法也十分重要,将几种主流分型方式列举如下。
当前我国学者根据单核苷酸多态性(single nucleotide polymorphism,SNP)对SARS-CoV-2进行分型。在中国分型法中,S型:基因组第28 144位核苷酸为C,第8 782位核苷酸为T;L型:第28 144位为T,第8 782位为C。S型与蝙蝠、穿山甲冠状病毒相似度更高,推测S型在进化上更加原始。L型首先分为欧洲家系和全球家系;L型欧洲家系再细分为欧洲家系I(第28 881位G→A,第28 882位G→A,第28 883位G→C)和欧洲家系II(第25 563位G→T),欧洲家系II进一步又分为欧洲家系II.1(第1 059位C→T),欧洲家系II.2(第2 416位C→T)和欧洲家系II.3(第18 877位C→T,第25 563位G→T)[7],由于欧洲家系II.1在美洲流行,又称作美洲家系[8]。我国武汉早期流行株以S型为主,目前国际上以L型为主[9]。
GISAID是全球流感共享数据库,目的是助力全球呼吸道传染病相关病毒研究。研究人员收集SARS-CoV-2的全基因组序列,统计系统发育簇的基因组距离,进行基因分型。依据共享突变,病毒变异株的分型根据实际突变字母标记,并归类于几种主要的进化支,如:S蛋白上D614G是一个新进化支的主要突变位点之一,据此命名为G簇变异株。与中国分型法一致,总体上将SARS-CoV-2分类为S和L簇[9],每一簇变异株产生新的突变,再细分为不同子簇。L簇进一步分为V和G簇。G簇进一步分类:GH簇、GR簇、GV簇和GRY簇[10]。
以系统发育归类命名全球暴发谱系(phylogenetic assignment of named global outbreak lineages,Pango lineages)分型法[11]中的两大主要分支A和B分别对应中国分类法中的S型(A)和L型(B);在A、B两主分支下细分出众多子分支。为了便于交流及实际应用,规定迭代过程中同一首字母开头的分支最多存在三个子级别(如B.1.1.1),之后会为下一子分支指定另一字母[6],例如B.1.1.28.1即P.1株。
Nextstrain分型法是Nextstrain.org网站的研究人员,按照推算的病毒出现年份和时间顺序来命名簇,根据SNP命名他们的亚簇和子簇。最先出现的19A和19B主簇,即2019年发现的A和B簇的SARS-CoV-2,分别对应于中国分型法中L型(19A)、S型(19B)[12]。与GISAID分型法和Pango谱系分型法不同,Nextstrain分型法显示持续时间及地理分布特征。
美国卫生与公共服务部下属的新冠病毒多机构小组将变异株分为存疑变异株(variant of interest,VOI),关切变异株(variant of concern,VOC),严重后果变异株(variant of high consequence,VOHC)[13]。世界卫生组织(World Health Organization,WHO)也做出VOC和VOI的分类[14],并于2021年5月31日发表倡议,建议使用希腊字母标注VOC和VOI,简化公共交流,抵制命名造成的污名化和歧视,同时也支持GISAID、Nextstrain和Pango分型继续用于科学交流与研究[15]。
WHO提出的VOI是指出现可能或已知影响传染性、疾病严重程度、免疫逃逸、诊断和治疗的特异位点突变的病毒株,在多个国家的相对流行率不断增加,全球公共卫生面临新的风险。VOC是指符合VOI定义,经评估证实存在以下至少一种特点的变异株:①病毒传染性增加;②致病性增强或临床症状改变;③公共卫生及社会措施或诊断、疫苗、治疗方法的有效性降低。目前指定的VOCs有Alpha、Beta、Gamma、Delta变异株(表1)[14]。下面我们将逐一介绍目前发现的最主要的几种流行变异株。
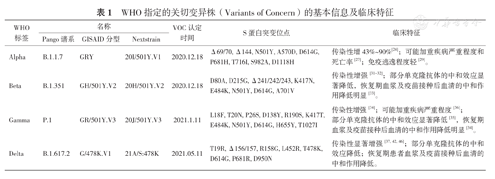
WHO指定的关切变异株(Variants of Concern)的基本信息及临床特征
WHO指定的关切变异株(Variants of Concern)的基本信息及临床特征
| WHO标签 | 名称 | VOC认定时间 | S蛋白突变位点 | 临床特征 | ||
|---|---|---|---|---|---|---|
| Pango谱系 | GISAID分型 | Nextstrain | ||||
| Alpha | B.1.1.7 | GRY | 20I/501Y.V1 | 2020.12.18 | Δ69/70, Δ144, N501Y, A570D, D614G, P681H, T716I, S982A, D1118H | 传染性增43%~90%[24];可能加重疾病严重程度和死亡率[27];免疫逃逸程度轻[29]。 |
| Beta | B.1.351 | GH/501Y.V2 | 20H/501Y.V2 | 2020.12.18 | D80A, D215G, Δ241/242/243, K417N, E484K, N501Y, D614G, A701V | 传染性增强[31,32];部分单克隆抗体的中和效应显著降低,恢复期血浆及疫苗接种后血清的中和作用降低明显[33]。 |
| Gamma | P.1 | GR/501Y.V3 | 20J/501Y.V3 | 2021.1.11 | L18F, T20N, P26S, D138Y, R190S, K417T, E484K, N501Y, D614G, H655Y, T1027I | 传染性增强[34];可能加重疾病严重程度[36];部分单克隆抗体的中和效应显著降低[35],恢复期血浆及疫苗接种后血清的中和作用降低明显[34]。 |
| Delta | B.1.617.2 | G/478K.V1 | 21A/S:478K | 2021.05.11 | T19R, Δ156/157, R158G, L452R, T478K, D614G, P681R, D950N | 传染性显著增强[37,42,46];部分单克隆抗体的中和效应降低;恢复期患者血浆及疫苗接种后血清的中和作用降低。 |
SARS-CoV-2的D614G变异是病毒S蛋白上第614位天冬氨酸(D)突变为甘氨酸(G)。这种突变几乎总伴随着另外三个核苷酸突变:C241T、C3037T和C14408T,可引起5'UTR区域的同义突变和RdRp发生P323L氨基酸突变[16]。
自从2020年3月上旬首次被识别后,D614G变异株迅速成为全球优势病毒株。在2020年3月之前,在抽样的997个全球序列中仅占10%,而在2020年4月至2020年5月18日间,D614G病毒株占了12 194个抽样序列的78%。根据GISAID网站追踪,D614G的转变在全球非同步出现,首先是欧洲,其次是北美和大洋洲,随后是亚洲,逐渐演变为全球最流行的B.1系变异株[16]。
D614G增强病毒的致病性的机制有以下几方面:①使S蛋白中S1和S2之间的结构更加稳定,S1蛋白脱落减少。②使更多的RBD位点处于开放状态,结合位点更多。③改变S蛋白的构象,与ACE2结合更有效[17]。④G614病毒株的培养液比D614病毒株具有更高的病毒滴度[16]。
D614G变异株并没有引起更严重的临床结局。Corman等[18]的研究显示感染G614病毒株的患者的样本进行核酸检测时有更低的循环阈值(Cycle Threshold Value,Ct值),即更高的病毒载量,但重症和危重症患者比例没有增高,提示并未增加疾病的严重程度。G614病毒株和D614对中和抗体的敏感性类似,体液免疫、细胞免疫对二者的抑制效应无明显差别[19]。但有些体外研究显示G614增加了单克隆抗体对病毒的敏感性[20]。推测可能由于D614G变异株的RBD更大,在表达G614的S蛋白中暴露得更多,增加免疫原性[16]。
2020年12月上旬,英国监测报告了英格兰肯特郡(Kent)抽样的255例SARS-CoV-2的测序结果,其中有117株(45.9%)病毒形成一个独特的分支,即Alpha(B.1.1.7)变异株,也称20I/501Y.V1 (Nextstrain命名)。Alpha(B.1.1.7)变异株共有17个突变,其中S蛋白上有9个突变:Δ69/70、Δ144、N501Y、A570D、D614G、P681H、T716I、S982A、D1118H,另外也可能合并E484K、S494P和K1191N。
N501Y的突变是S蛋白RBD第501位氨基酸由天冬氨酸(N)突变为酪氨酸(Y),该突变不仅改变RBD构象使SARS-CoV-2对人体ACE2受体亲和力增加,还可增加病毒粒子与精氨酸-甘氨酸-天冬氨酸基序(RGD motif)结合位点的亲和力[21]。Δ69/70影响RBD构象,叠加增强S蛋白对ACE2受体亲和力,增强病毒传染性并有助于病毒发生免疫逃逸[22,23],这种缺失也是某些检测试剂盒未能检测到病毒S基因的原因[24]。S蛋白只有被切割,暴露出开放结构区域,才能与人体ACE2受体结合。P681H是S蛋白受体S1和S2区域连接处的弗林蛋白酶切割位点的一部分。该连接处的变化增强了S蛋白上切割位点对蛋白酶的敏感性,能明显提高病毒的传播效率[25]。英国学者建立了统计与预测模型,预测比英国先前病毒株的传播力增强了43~90% (95% CIs: 38~130%)[24]。Alpha(B.1.1.7)病毒株在英格兰南部扩展至全国,截至2021年2月,英国95%的新发感染均为B.1.1.7[24],逐渐扩展到全球,截至2021年7月15日,波及至少159个国家[26]。
Alpha(B.1.1.7)变异株可能增加疾病严重程度及死亡风险,收集了2020年11月至2021年2月的英国SARS-CoV-2阳性患者数据,结果显示各年龄阶段感染B.1.1.7株的患者比以往感染SARS-CoV-2患者的死亡风险升高,总体死亡风险增加61%。死亡风险升高的具体机制目前尚不明确,患者病毒载量高可能与病情严重程度相关[27]。
单克隆抗体及疫苗作用后血清、恢复期患者血浆中的中和抗体需要与病毒S蛋白上RBD结合发挥作用,N501Y突变可影响中和抗体作用[28]。Supasa等[29]发现N501Y突变能降低部分抗RBD抗体中和效应,Δ68/70、Δ144使抗NTD抗体的作用减弱,但仍有部分抗体(mAb 40、88、222、316、384、398,AZD1061/8895,REGN10933/10987)所受影响小。该研究还证实B.1.1.7变异株对阿斯利康AZD1222疫苗和辉瑞BNT162b2的中和效价下降2.1~3.3倍。如果变异株同时携带E484K突变,则对mRNA疫苗刺激产生的抗体中和作用进一步减弱[30]。
Beta变异株包括B.1.351及其分支B.1.351.2和B.1.351.3,又称20H/501Y.V2,最早于2020年12月在南非开普敦发现。B.1.351变异病毒的S蛋白上具备8个突变或位点缺失:D80A、D215G、Δ241/242/243、K417N、E484K、N501Y、D614G和A701V。N501Y和D614G突变,及合并RBD上的K417N、E484K突变,都可改变RBD构象,增强与ACE2受体亲和力,增加传染性[31,32],截至2021年7月15日,Beta(B.1.351)变异株已扩散至全球至少106个国家[26]。
Beta(B.1.351)变异株有免疫逃逸的特性。有研究[33]比较了12种针对RBD抗体对B.1.351和B.1.1.7的中和活性,发现E484K突变导致910-30、2-15、LY-CoV555和C121抗体对B.1.351的中和活性完全丧失;K417N导致10-30抗体活性的丧失;二者叠加使REGN10933抗体活性的显著降低。在12中RBD抗体中,910-30和S309抗体对B.1.1.7中和活性降低,其他10种抗体对B.1.1.7的活性无影响或影响轻微。由于Δ241/242/243及其他合并突变,B.1.351对6种NTD的单克隆抗体出现了抵抗性,而B.1.1.7对这6种的抗体抵抗性较B.1.351稍弱。由于E484K突变恢复期血浆对B.1.351病毒株的中和作用减弱约9.4倍,接种Moderna(V1~V12)或辉瑞(V13~V22)疫苗后产生中和效价减弱10.3~12.4倍[33]。B.1.351变异株对单克隆抗体、恢复期血浆的中和作用提出了新的挑战,并威胁到当前疫苗的保护功效,引起人们的广泛关注,研究者们也在致力研制敏感性更高单克隆抗体药物及能产生更强中和抗体的疫苗。
Gamma变异株包括P.1、P.1.1和P.1.2,P.1变异株来自B.1.1.28谱系,又称20J/501Y.V3,最早于2020年12月在巴西玛瑙斯市出现,到2021年1月中旬造成整个城市疫情的大规模暴发,约有85.4%病例为P.1病毒株。P.1病毒株在巴西迅速蔓延,截止2021年7月15日,在超过62个国家流行[26]。该变异株的S蛋白上存在11个突变,除D614G外,还包括RBD中的K417T、E484K和N501Y;NTD中的L18F、T20N、P26S、D138Y和R190S;和靠近Furin裂解位点的H655Y[34]。P.1变异株的RBD突变与B.1.351基本相似,存在可引起免疫逃逸的E484K、N501Y突变。Hoffmann等[35]研究显示相比B.1.1.7变异株,P.1和B.1.351对REGN10989和Bamlanivimab抗体几乎完全耐药,对Casirivimab抗体部分抵抗。与B.1.351类似,恢复期血浆、疫苗接种者血清对P.1病毒株的作用减弱。另一些研究结果显示恢复期血浆、疫苗接种者血清对P.1病毒株中和活性减低程度比B.1.351株更明显[34]。Gamma(P.1)变异株还引起重症和危重症比例升高。研究人员总结了欧洲多个国家COVID-19病例并分析了B.1.1.7、B.1.351和P.1的临床特点,感染这三种变异株病例的住院率显著高于非VOC病例,入住ICU的可能性分别是非VOC病例的2.3、3.3和2.2倍,并显示在各年龄段有病情加重风险[36]。
2021年3月之后出现的印度第二波新冠疫情,2个月内就新增了约0.36%印度人口的感染患者,仅在2021年5月印度新增902余万例COVID-19患者,是自2019年疫情以来各国疫情最严重的一月[37]。上文提到的B.1.1.7、B.1.351和P.1变异株都在印度流行,但最初在印度识别的Delta(B.1.617.2)变异株在印度第二波新冠疫情占主要优势。
B.1.617变异株,包括B.1.617.1、B.1.617.2和B.1.617.3,其S蛋白上特征突变有:L452R、D614G、P681R,可伴随E484Q、Q107H、T19R、T478K、D950N、Δ157/158[38]。而B.1.617.2(关键突变:L452R、T478K和P681R)迅速成为印度甚至全球最主要流行变异株。截至2021年7月15日,在超过99个国家流行[26,39]。与N501Y不同,L452R残基位于S蛋白RBD的疏水结构中,该突变不直接接触ACE2受体,但可导致RBD结构变化,可稳定S蛋白与人体ACE2受体之间的相互作用,增强病毒感染性[40]。T478K位于S蛋白与ACE2受体的相互作用域内,该突变可能会影响病毒与人类细胞的亲和性,可能造成传染性增强[41]。P681R类似Alpha(B.1.1.7)变异株中P681H突变,也能提高病毒的传播效率[25,42]。E484Q具有E484K突变相似的特点,二者都提供了印度变异株免疫逃逸的基础[33]。B.1.617变异株继续进化,出现新的突变:AY.1/ AY.2,被称为"Delta Plus"变异株,在S蛋白上获得了新突变K417N,使病毒更容易粘附在人体细胞上[43],并可能减弱单克隆抗体对病毒的中和作用[44]。2021年7月6日起,AY.1和AY.2变异株与B.1.617.2合并,统称为Delta变异株[14,44]。
Epsilon(B.1.427/ B.1.429)变异株又称20C/L452R,最早于2020年5月在美国加利福尼亚出现,于2021年1月识别并命名,目前被WHO归类为"进一步监测的变异株"[14]。回溯分析发现从2020年9月至2021年1月的测序结果,该变异株相对于野生病毒的传播率增加18.6%~24%[52]。Epsilon(B.1.427/ B.1.429)变异株共有的S蛋白变异有L452R,D614G;而B.1.429的S蛋白上还包含了S13I和W152C。Epsilon(B.1.427/ B.1.429)变异株存在免疫逃逸。McCallum等人[40]评估了35种RBD单克隆抗体的作用效果,L452R突变降低或消除了其中的14种抗体的中和活性;12种NTD单克隆抗体中,S13I突变抑制了5种抗体与病毒结合,并消除了另外5种抗体结合作用,W152C突变降低了对6种NTD抗体的识别,其中2种完全丧失结合能力。该研究还发现Moderna的mRNA1273疫苗接种后人群的血清对B.1.427/B.1.429的中和效力降低2.8倍,辉瑞BNT162b2疫苗下降3倍。
D614G变异是最早被识别并引起重视的变异株,病毒的传播性增强,目前D614G变异为基础的B.1谱系已经成为全球最广泛流行的变异株。随后高传染性、高致病性的Alpha(B.1.1.7)出现,使人们注意到N501Y突变改变S蛋白上RBD等结构从而改变临床特征。同样的变异也发生在Beta(B.1.351)和Gamma(P.1),除D614G和N501Y外,这两种变异株同时还携带E484K和K417T/N突变,增加了病毒RBD与人体ACE2受体的亲和力和病毒的免疫逃逸能力。Delta(B.1.617.2)变异株同时具有L452R、T478K和P681R突变,因传染性增强、致病性强、免疫逃逸,目前在印度和世界广泛传播,是全球最主要流行变异株。Epsilon(B.1.427/ B.1.429)变异株代表突变为L452R,暂被WHO列为"进一步监测的变异株",因其具有明显免疫逃逸表现,对诸多单克隆抗体、恢复期血浆、疫苗产生的抗体的应答欠佳,需要严密监测。
所有作者均声明不存在利益冲突



























