2019新型冠状病毒(2019 novel coronavirus,2019-nCoV)是新近分离鉴定的第7种可以感染人类的冠状病毒,已确认为目前正在暴发流行的COVID-19的病原体。2019-nCoV可在COVID-19患者的支气管肺泡灌洗液、鼻咽拭子、血液等样本中检出,细胞分离培养相对容易。鉴于病毒分离培养和病原学鉴定对病毒复制增殖、致病性、免疫、检测诊断和特异防治等都具有重要意义,此文就2019-nCoV分离、病原学鉴定以及根据宏基因组测序的反向病原学研究现状与进展做一综述。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
2003年SARS和2012年中东呼吸综合征(Middle East respiratory syndrome,MERS)的全球性疫情暴发[1],使冠状病毒(coronavirus,CoV)受到越来越多的关注。2019年12月开始,武汉市暴发2019新型冠状病毒(2019 novel coronavirus,2019-nCoV)感染引起的COVID-19疫情,并迅速波及全国及世界,被WHO列为国际关注的突发公共卫生事件。采用病毒分离培养和宏基因组测序(metagenome next-generation sequencing,mNGS)迅速分离和鉴定引起疫情暴发的病原体并获得全基因组序列,对疫情防控起到了关键作用。海军军医大学全军生物侦检与防护重点实验室承担COVID-19疑似病例核酸检测任务,同时关注着2019-nCoV分离与病原学鉴定进展,现综述如下。
CoV为单股正链RNA病毒,基因组全长约30 kb,由5′端非编码区、非结构蛋白开放阅读框1ab编码区、4种主要结构蛋白编码区以及3′端非编码区组成[2]。结构蛋白编码区主要编码刺突蛋白、包膜蛋白、膜蛋白和核衣壳蛋白(nucleocapsid protein,N)[3]。一般认为野生动物如蝙蝠是CoV的天然宿主,蝙蝠携带的CoV可以通过中间宿主跨物种感染人类[4]。此前已发现6种CoV可以引起人类疾病,其中4种(HCoV-229E、HCoV-NL63、HCoV-OC43和HCoV-HKU1)可引起普通感冒样症状[5],另外2种(SARS-CoV和MERS-CoV)感染后病情较重,可引起重度肺炎、呼吸困难,病死率较高[6]。2003年SARS疫情暴发,造成全球29个国家和地区8 096例感染,其中774例死亡[7]。2012年6月,沙特阿拉伯暴发MERS疫情,随后传及亚洲、非洲、欧洲和美洲的多个国家和地区[8]。据WHO报告,截至2019年11月底,MERS在27个国家和地区共有2 494确诊病例,858例死亡,病死率34.4%[9]。
采集COVID-19患者鼻(咽)拭子、支气管肺泡灌洗液、血液、痰液或粪便等标本[14],低温(2~8 ℃)无菌保存,安全运送,用于核酸、抗原检测或细胞分离培养。其中支气管肺泡灌洗液样本的病毒含量高、活性好,最适合进行病毒分离培养[12,15,16,17]。
以支气管肺泡灌洗液为例,离心去除细胞碎片,取上清液接种于培养状态良好并经PBS洗3遍的人呼吸道上皮细胞,细胞密度25万/孔,37 ℃孵育2 h后弃去培养基,PBS洗去未与细胞结合的病毒。每48小时加150 μl PBS,37 ℃孵育10 min后,收集细胞顶端面样品,1∶3稀释传代到新的细胞中。光学显微镜下每天观察细胞病变,并用实时荧光定量PCR(real-time PCR,RT-PCR)检测上清液中的病毒核酸。经过3次传代后,高速离心收集上清液中病毒用于测序[12,17],并将细胞制成超薄切片用透射电子显微镜(电镜)观察病毒形态。
此外,Zhou等[16]用Vero细胞和Huh7细胞也成功分离了患者肺泡灌洗液中的2019-nCoV,并观察到明显的细胞病变,Lu等[17]从咽拭子样本中也分离出2019-nCoV。
对细胞培养分离的2019-nCoV,用核酸检测、抗原检测、电镜检查以及mNGS等方法进行鉴定。
核酸检测是目前2019-nCoV鉴定和COVID-19患者确诊的重要方法。在获得病毒全基因组序列后,多项研究设计了靶向2019-nCoV的特异引物和对应的荧光探针[12,16,17,18,19](染料法不需要探针,具体序列见表1),通过RT-PCR检测样本中2019-nCoV。
| 基因 | 引物(5′-3′) | 探针 |
|---|---|---|
| orf1ab | F: AGAAGATTGGTTAGATGATGATAGT | FAM-TCCTCACTGCCGTCTTGTTGACCA-BHQ1 |
| R: TTCCATCTCTAATTGAGGTTGAACC | ||
| orf1ab | F: CCCTGTGGGTTTTACACTTAA | FAM-CCGTCTGCGGTATGTGGAAAGGTTATGG-BHQ1 |
| R: ACGATTGTGCATCAGCTGA | ||
| N | F: GGGGAACTTCTCCTGCTAGAAT | FAM-TTGCTGCTGCTTGACAGATT-TAMRA |
| R: CAGACATTTTGCTCTCAAGCTG | ||
| S | F: CCTACTAAATTAAATGATCTCTGCTTTACT | - |
| R: CAAGCTATAACGCAGCCTGTA | - | |
| S | F: CAATGGTTTAACAGGCACAGG | - |
| R: CTCAAGTGTCTGTGGATCACG | - | |
| RdRpa | F: GTGARATGGTCATGTGTGGCGG | FAM-CAGGTGGAACCTCATCAGGAGATGC-BBQ |
| R: CARATGTTAAASACACTATTAGCATA | ||
| Ea | F: ACAGGTACGTTAATAGTTAATAGCGT | FAM-ACACTAGCCATCCTTACTGCGCTTCG-BBQ |
| R: ATATTGCAGCAGTACGCACACA |
注:2019-nCoV:2019新型冠状病毒;orf1ab:开放阅读框1ab; N:核衣壳蛋白;S:刺突蛋白;RdRp:RNA依赖RNA聚合酶;E:包膜蛋白;F:正向;R:反向;FAM:荧光素;-:无;a针对RdRp和E基因的引物和探针由Corman等[19]在2019-nCoV序列确定前设计并测试
截至2020年1月31日,国家药品监督管理局已经应急审批通过7家企业的7个2019-nCoV核酸检测试剂盒(RT-PCR法)产品。
鉴于蝙蝠SARSr-CoV Rp3与2019-nCoV的N具有92%的氨基酸一致性,Zhou等[16]利用抗SARSr-CoV Rp3 N抗体,采用间接免疫荧光法检测到Vero E6细胞培养中的2019-nCoV。作者利用重组蛋白检测COVID-19患者的血清抗2019-nCoV N抗体,7例患者中5例IgG抗体强阳性,3例IgM抗体阳性。
分离培养的病毒感染细胞或上清液可用于透射电镜观察,负染色的2019-nCoV粒子通常为球形并具有一定的多形性,直径60~140 nm,表面呈日冕样并有明显刺突(长9~12 nm),细胞质膜结合囊泡中可观察到充满病毒颗粒的包涵体,见图1[12]。
mNGS在2019-nCoV基因组测序、溯源分析和反向病原学鉴定中发挥了关键作用[12,15,16,17,18,20,21]。
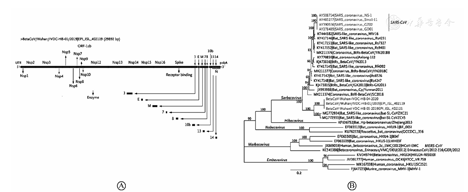
mNGS以支气管肺泡灌洗液或细胞培养物上清液中的病毒RNA为模板,除去宿主核酸,进行cDNA合成用于文库建库,联合Illumina和Nanopore测序平台进行基因组测序,使用CLCBio生物信息学软件进行片段组装,设计特异引物用于PCR并使用5′-和3′-cDNA末端的快速扩增获得全基因组序列[12]。全基因组序列比较和系统树分析有助于明确2019-nCoV与其他病毒的亲缘关系(图2),推测病毒来源、人群传播方式和是否发生重组等[17]。
mNGS在2019-nCoV鉴定中发挥了不可替代的作用,而由mNGS提供的基因组信息在COVID-19病原体确认中又起到了关键作用,这些都得益于反向病原学研究的进展。传统病原学鉴定是先鉴定出病原体,然后确定该病原体与疾病的关系,而近年来提出的反向病原学,则是先进行mNGS找出病原体基因组序列,然后由基因组序列反推出病原体。飞速发展的mNGS技术与生物信息学和病原体基因组学结合,已经成为发现和鉴定病原体的新途径[22],此次mNGS技术在2019-nCoV发现和COVID-19病原体确认中的应用,为未来公共卫生突发事件应对和传染病防控提供了有益借鉴,具有深远意义。
所有作者均声明不存在利益冲突