
随着人工智能(AI)技术的兴起与发展,将AI应用于医疗健康领域正日益受到广泛关注。本文以机器学习、深度学习和迁移学习等方法为代表,概述了在睡眠医学领域的相关研究工作,涵盖了睡眠分期、睡眠纺锤波、睡眠呼吸暂停、快动眼睡眠行为障碍、失眠以及发作性睡病等。同时,分析并探讨了未来AI的发展趋势,充分发挥AI在大数据分析和智能决策上的优势,使其在睡眠医学领域能够得以进一步地推广与应用。

在我国约有超过10%的人存在需要积极干预的睡眠疾病。睡眠疾病不仅会降低生活质量,还会引发一系列疾病,如心脑血管和代谢系统疾病,也与抑郁症等精神心理疾病相关,已逐步发展为危害公共健康的重要问题。多导睡眠监测(polysomnography,PSG)是检测睡眠疾病的“金标准”,然而对于多导睡眠仪监测到的电生理信号的解读,一般需要医技人员通过同步记录患者的电生理信号,并结合睡眠及其相关事件判读手册[如由美国睡眠医学会(American Academy of Sleep Medicine,AASM)发布的手册]以及临床经验来进行分析与判读,这是一个繁琐且耗时的过程。临床睡眠数据分析,不仅专业性要求高、解读难度大、解读时间冗长,而且易受主观因素的影响。人工智能(artificial intelligence,AI)方法可以提供自动、高效、精准的数据分析,避免主观误差,减轻医生数据分析的工作负荷,因而在睡眠医学领域受到了广泛研究。
AI是指可以模拟、延伸和扩展人类的智能,感知、获取知识并使用知识解决问题的智能系统。随着移动互联网的兴起、信息数据的积累和增长、理论算法的突破、计算能力和硬件平台的提升等,AI技术在计算机视觉、语言识别、图像处理等领域都取得了突破性的进展。近年来,AI在医疗领域,如医学图像处理、睡眠数据分析等方面也有着广泛的应用。国务院《新一代AI发展规划》也指出AI产业进入全球价值链高端的战略目标,明确了新一代AI在智能医疗领域的应用。本文就AI技术在睡眠医学中的应用情况进行综述。
目前,AI技术在睡眠医学领域的主要应用如图1所示,具体包括:通过机器学习、深度学习、迁移学习等实现睡眠分期;脑电图(electroencephalogram,EEG)、眼电图(electrooculogram,EOG)等相关的微小事件,如N2阶段的K复合波、纺锤波、快眼动等的识别;相关睡眠疾病的判别,如睡眠呼吸暂停、发作性睡病等。
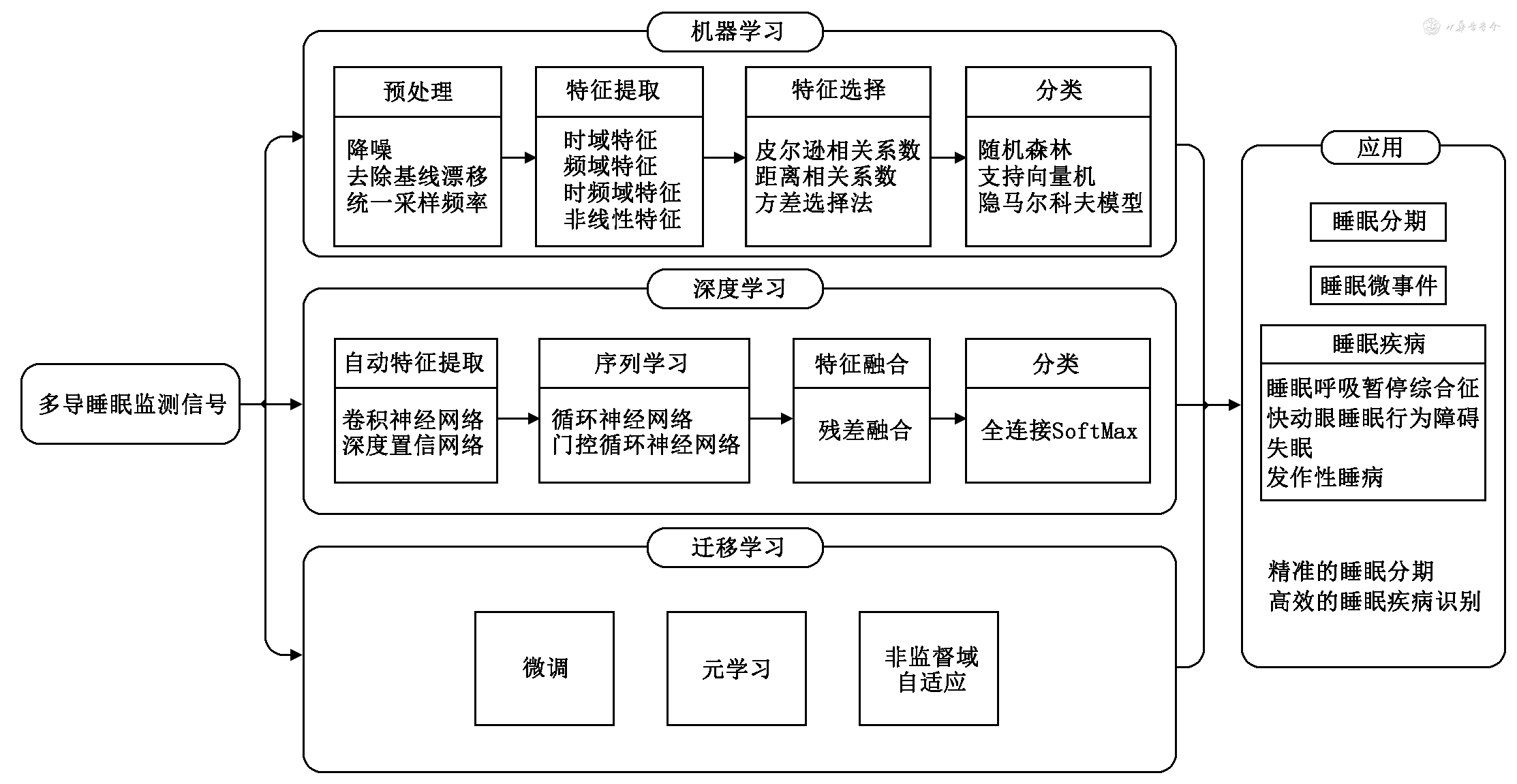

关于自动睡眠分期系统的实现,其本质是寻求一种模式识别方法,核心是从原始的电生理信号中提取与睡眠状态信息关联性高、具有良好类别表达性的特征,再通过分类器在特征空间进行类别划分。由于电生理信号的信息冗余大,数据维度高,特征提取对模型的预测结果影响巨大。目前,多数研究主要利用先验领域知识(传统机器学习)与模型自动学习(深度学习)两种方式来实现特征提取,均达到了较好的准确度,尤其是基于深度学习的睡眠分期技术。此外,由于受试者、实验设备、环境的不同,数据分布会存在一定的偏倚,致使已训练好的分类模型在直接应用于异构数据时,较难达到预期的识别效果。而迁移学习可通过模型迁移、领域自适应等方法有效地缓解此类问题,近年来也得到了广泛的关注。
1. 基于传统机器学习的睡眠分期方法:在过去几十年,不同的机器学习方法先后被运用到睡眠分期中[1, 2, 3, 4, 5, 6, 7]。基于传统机器学习的睡眠分期方法通常分为两步,第一步是信号的预处理、特征的提取与选择,第二步为分类器的选择和应用。首先,通过预处理来减少信号的噪声,然后从电生理信号中提取时域、频域、时频域和非线性等多种特征。之后,为避免特征数目过多而导致分类模型过拟合问题,通常会有一个特征选择的过程。特征选择主要为摒弃无关特征和冗余特征,保留相关特征,达到降维、降低学习难度的目的。最后,便是利用不同的分类器来完成睡眠阶段的多分类任务。在传统的机器学习领域,分类器的种类有很多,目前在睡眠分期领域广泛使用的分类器主要有随机森林(random forest,RF)[1, 2, 3]、支持向量机(support vector machine,SVM)[4, 5, 6]、隐马尔可夫模型(hidden Markov model,HMM)[7]等。这些方法在睡眠分期中都取得了一定的效果。然而,这些分类器的效果很大程度上依赖于前期提取的特征参数,实际上从微弱的EEG、EOG等信号挖掘新的具有高敏感性和特异度的特征参数难度较大,这也成为了制约睡眠分期性能提升的主要瓶颈。
2.基于深度学习的睡眠分期方法:近几年深度学习方法受到了广泛的关注,许多研究人员开始尝试使用深度学习算法,如卷积神经网络(convolutional neural networks,CNN)、深度信念网络(deep belief network,DBN)、循环神经网络(recurrent neural network,RNN)等,实现睡眠阶段的分类。目前,多数基于深度学习的睡眠分期方法可以实现端到端(即原始电生理信号到睡眠阶段)的映射,主要通过CNN、DBN等实现特征的自动提取;通过长短期记忆网络(long short-term memory,LSTM)、双向门限循环神经网络(bidirectional gated recurrent unit,BiGRU)等学习特征中所蕴含的睡眠阶段间的序列关系;然后通过残差连接实现不同类型特征的融合;最后通过全连接的Softmax层对融合后的特征进行分类。
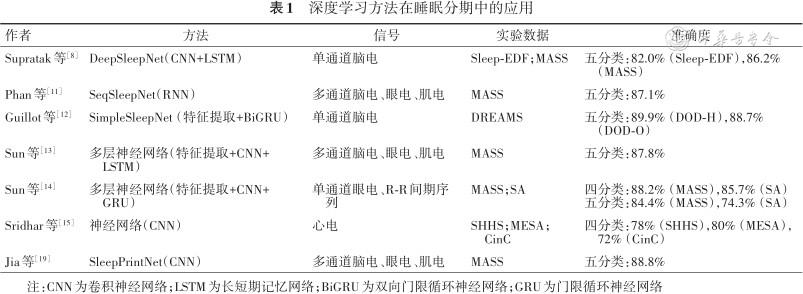
表1列举了部分基于深度学习的睡眠分期研究方法。Supratak等[8]提出了DeepSleepNet的自动睡眠分期网络,DeepSleepNet是一个结合了CNN和LSTM多层次的神经网络,该网络分别在Sleep-EDF(physiobank databases--the sleep-EDF database [expanded])数据集[9]和MASS(Montreal archive of sleep studies,MASS)数据集[10]上进行了测试,睡眠阶段五分类准确度分别为82.0%和86.2%。Phan等[11]提出了SeqSleepNet的自动睡眠分期网络,首次提出了序列对序列的网络结构,即多输入多输出网络,可以一次分析多帧数据。该网络在MASS数据集上进行测试,睡眠阶段五分类准确度为87.1%。Guillot等[12]提出了SimpleSleepNet的自动睡眠分期网络,该网络在Dream Open Datasets数据集上进行了睡眠阶段五分类的测试,在DOD-H数据集上准确度可以达到89.9%,在DOD-O数据集上准确度可以达到88.7%。Sun等[13]出了一个多尺度多层次的自动睡眠分期网络,该网络由多个CNN同时进行特征提取,并结合人工特征提取,对于特征的处理比较全面。该网络在MASS数据集上进行睡眠分期的五分类测试,准确度可以达到87.8%。同时,Sun等[14]提出仅使用EOG和心电图(electrocardiogram,ECG)的R-R间期信号来实现睡眠分期,并在MASS数据集和SA(PhysioBank Databases-St. Vincent's University Hospital/University College Dublin Sleep Apnea Database)数据集[9]上进行了测试,在MASS数据集上睡眠分期五分类准确度可以达到84.4%,在SA数据集上睡眠分期五分类准确度可以达到74.3%。Sridhar等[15]提出只使用从ECG中提取出来的瞬时心率作为输入信号的自动睡眠分期网络来进行睡眠阶段分类。该方法在SHHS[16]、MESA[17]和CinC[18]数据集上进行了四分类的睡眠分期测试,最终的准确度分别是78%、80%、72%。此外,Jia等[19]提出了SleepPrintNet的自动睡眠分期网络,该网络输入包括多通道的EEG、EOG和肌电图(electromyogram,EMG)信号,特征十分丰富,在MASS数据集上进行五分类的睡眠分期测试,准确度可以达88.8%。
深度学习方法在睡眠分期中的应用
深度学习方法在睡眠分期中的应用
| 作者 | 方法 | 信号 | 实验数据 | 准确度 |
|---|---|---|---|---|
| Supratak等[8] | DeepSleepNet(CNN+LSTM) | 单通道脑电 | Sleep-EDF;MASS | 五分类:82.0% (Sleep-EDF),86.2% (MASS) |
| Phan等[11] | SeqSleepNet(RNN) | 多通道脑电、眼电、肌电 | MASS | 五分类:87.1% |
| Guillot等[12] | SimpleSleepNet (特征提取+BiGRU) | 单通道脑电 | DREAMS | 五分类:89.9% (DOD-H),88.7% (DOD-O) |
| Sun等[13] | 多层神经网络(特征提取+CNN+LSTM) | 多通道脑电、眼电、肌电 | MASS | 五分类:87.8% |
| Sun等[14] | 多层神经网络(特征提取+CNN+GRU) | 单通道眼电、R-R间期序列 | MASS;SA | 四分类:88.2% (MASS),85.7% (SA) 五分类:84.4% (MASS),74.3% (SA) |
| Sridhar等[15] | 神经网络(CNN) | 心电 | SHHS;MESA;CinC | 四分类:78% (SHHS),80% (MESA),72% (CinC) |
| Jia等[19] | SleepPrintNet(CNN) | 多通道脑电、眼电、肌电 | MASS | 五分类:88.8% |
注:CNN为卷积神经网络;LSTM为长短期记忆网络;BiGRU为双向门限循环神经网络;GRU为门限循环神经网络
深度学习方法在睡眠分期中具有一定的优势,从特征提取的角度来看,深度学习方法可以通过不同的滤波器组提取不同的形态特征,如细节、结构和形状特征等。此外,深度学习方法可以从连续睡眠时期中提取时间序列信息来帮助分类。相比传统的机器学习方法,深度学习方法在处理时序信号的分类问题上具有一定的优势,在睡眠分期准确性上也有明显地改善。
3.基于迁移学习的睡眠分期方法:近两年,有人尝试着利用迁移学习的方法解决标签数据过少的问题,将某一个数据量比较充足的数据集用于学习,然后将学习到的知识/模型迁移到另一个数据量比较少的数据集上。Phan等[20]以MASS数据集作为源域,以Sleep-EDF和Surry-cEEGrid数据集作为目标域,以SeqSleepNet和DeepSleepNet两个网络模型作为训练网络进行了迁移学习的训练,在两个目标域数据库上都达到了较高的准确度,结果表明了迁移学习方法的可行性。Banluesombatkul等[21]提出了MetaSleepLearner迁移学习网络模型,通过在MASS数据库上学习模型的最佳初始参数,将其应用于不同睡眠中心的睡眠数据上,只需较少的有标注样本对模型进行微调,即可达到较好的分期准确度。
迁移学习可以利用已有的公开数据集对模型进行预训练,微调后再应用于新的睡眠数据分析,有效处理由于多中心、不同采集设备、不同配置等引起的数据一致性差的问题,并提高模型的可用性,而且可以有效节省人工标注样本的时间。
睡眠时,由于人体内环境和大脑皮层神经元兴奋性的变化,EEG、EOG等除了节律特征外还会出现许多特征性波形,如K复合波、纺锤波、快眼动、慢眼动等,这些特征性波形(也称为睡眠微事件)是判别睡眠阶段的主要依据,同时也与睡眠质量、睡眠疾病等息息相关。
近几年随着AI技术的发展,研究人员逐渐开始利用机器学习和深度学习的方法来实现睡眠微事件的识别。表2列举了部分基于机器学习和深度学习的睡眠微事件识别研究方法。

机器学习、深度学习方法在睡眠微事件中的应用
机器学习、深度学习方法在睡眠微事件中的应用
| 作者 | 方法 | 信号 | 实验数据 | 结果 |
|---|---|---|---|---|
| Lachner-Piza等[22] | 机器学习:SVM-纺锤波检测 | 脑电 | DREAMS;MASS | DREAMS:灵敏度为53%,精确度为37%,特异度为96%;MASS:灵敏度为77%,精确度为46%,特异度为96% |
| Hernández-Pereira等[24] | 机器学习:多种分类器-K复合波检测 | 脑电 | 自采集数据 | 准确度为91.4%,灵敏度为89.19% |
| Babadi等[25] | 机器学习:贝叶斯-纺锤波检测 | 脑电 | 自采集数据 | 灵敏度为83.4%,特异度达到97.2% |
| Kulkarni等[26] | 深度学习:CNN+RNN-纺锤波检测 | 脑电 | MASS;DREAMS | MASS:灵敏度为(90.07±2.16)%,特异度为(96.19±0.71)%;DREAMS:灵敏度为(77.85±4.28)%,特异度为(94.2±1.26)% |
注:SVM为支持向量机;CNN为卷积神经网络;RNN为循环神经网络
1.基于传统机器学习的睡眠微事件识别方法:通过提取电生理信号的时频域、非线性等特征,再结合传统的机器学习方法,可实现单一睡眠微事件的分类。Lachner-Piza等[22]使用SVM,实现了利用单通道EEG来检测睡眠纺锤波,无需进一步的参数微调就能在异构数据上获得稳定的性能。该算法在DREAMS数据集[23]上测试结果的灵敏度为53%,精确度为37%,特异度为96%,在MASS数据集上测试结果的灵敏度为77%,精确度为46%,特异度为96%。Hernández-Pereira等[24]提取并筛选Bankman特征来改善非线性分类器的泛化能力来实现了K复合波的识别。文中分析比较了特征选择方法和多种分类器(前馈神经网络、逻辑回归、SVM、近似SVM),最终仅使用36%的特征实现了高达91.4%的准确度,以及89.19%的灵敏度。Babadi等[25]提出了一种数据驱动的贝叶斯算法,将睡眠纺锤波视为一种随机过程,利用Karhunen-Loève变换和贝叶斯假设检验来计算纺锤波出现的瞬时概率,从而实现睡眠纺锤波的检测,灵敏度为83.4%,特异度达到97.2%。
2.基于深度学习的睡眠微事件识别方法:相比传统机器学习,深度学习方法需要更多的数据进行训练,但在性能上也有所提升。Kulkarni等[26]提出一种新型的深度学习算法(SpindleNet),将CNN和RNN相结合,在低采样率、低信噪比的单通道EEG信号上,实现了高准确度、高速率的睡眠纺锤波检测。同时模型也具有良好的泛化能力,在MASS数据集上灵敏度为(90.07±2.16)%,特异度为(96.19±0.71)%;在DREAMS数据集上的灵敏度为(77.85±4.28)%,特异度为(94.2±1.26)%。
目前,多数睡眠微事件识别算法仅能实现单一类别的微事件识别,对不同的睡眠事件需采用不同识别的方法,同时多数算法只实现了睡眠微事件的分类并没有实现微事件的精准定位。然而,睡眠微事件的出现时刻、出现频率、持续长短与睡眠阶段、质量、病理、疾病等息息相关,可为临床睡眠疾病诊断、睡眠机制研究提供丰富的信息。后期AI算法可以在多类别微事件识别以及微事件定位方法深入研究探讨。
目前关于自动识别睡眠呼吸暂停的研究中,大部分是基于SVM、RF等机器学习的方法,近期少量研究开始使用深度学习方法比如LSTM来处理这一问题(表3)。

机器学习、深度学习方法在睡眠呼吸暂停中的应用
机器学习、深度学习方法在睡眠呼吸暂停中的应用
| 作者 | 方法 | 信号 | 实验数据 | 结果 |
|---|---|---|---|---|
| Hassan和Haque[27] | 机器学习:RUSBoost | 心电 | Apnea-ECG;SA | 二分类(片段),准确度为88.88%,灵敏度为87.58%,特异度为91.49% |
| Zarei和Asl[28] | 机器学习:径向基函数核的SVM | 心电 | Apnea-ECG;SA | 二分类(片段),准确度为94.63%,灵敏度为94.43%,特异度为94.77%;二分类(患者):准确度为95.71%,灵敏度为95.83%,特异度为95.66% |
| Dong等[29] | 机器学习:局部聚类系数 | 心电(R-R间期序列) | Apnea-ECG;Sleep Heart Rate and Stroke Volume Data Bank | 二分类(患者),准确度为90.1%,灵敏度为88.29%,特异度为90.5% |
| Bhattacharjee等[30] | 机器学习:KNN | 脑电 | SA;MIT-BIH | SA数据库:二分类(片段),准确度为89.22%,灵敏度为95.21%,特异度为83.23%;MIT-BIH数据库:二分类(片段),准确度为86.14%,灵敏度为91.60%,特异度为80.82% |
| Lee等[31] | 机器学习:决策树 | 脑电、心电、血氧饱和度 | 自采集数据 | 二分类(患者),准确度为87.95%,灵敏度为88.1%;三分类(患者),准确度为75.44% |
| Jung等[32] | 机器学习:回归 | 心电(呼吸) | 自采集数据;Apnea-ECG | 预测AHI:绝对误差为(3.65±2.98)事件/h,皮尔逊相关系数为0.97(P<0.01) |
| Van Steenkiste等[34] | 深度学习:LSTM | 心电(呼吸) | SHHS | 二分类(片段),准确度为60.1%,灵敏度为52.1%,特异度为61.8% |
| Faust等[35] | 深度学习:LSTM | 心电(R-R间期序列) | Apnea-ECG | 二分类(片段),准确度为81.30%,灵敏度为59.90%,特异度为91.75% |
| Li等[36] | 机器学习+深度学习:特征提取:SAE;分类:SVM、ANN和HMM | 心电 | Apnea-ECG | 二分类(片段),准确度为84.7%,灵敏度为88.9% 二分类(患者),准确度为100% |
注:SVM为支持向量机;KNN为K邻近;LSTM为长短期记忆网络;SAE为稀疏自编码器;ANN为人工神经网络;HMM为隐马尔可夫模型
1.基于传统机器学习的睡眠呼吸暂停识别方法:利用ECG信号结合机器学习方法实现睡眠暂停的识别是比较常见的,有部分研究直接使用ECG,有部分研究是通过从ECG中提取R-R间期序列或心率变异性(heart rate variability,HRV),也有研究是从ECG中提取出呼吸信号(ECG-derived respiration,EDR)。Hassan和Haque[27]结合具有可调Q因子的小波变换(tunable-Q factor wavelet transform,TQWT)和RUSBoost(random under sampling boosting),用来自动识别阻塞型睡眠呼吸暂停(obstructive sleep apnea,OSA),OSA片段分类准确度为88.88%,灵敏度为87.58%,特异度为91.49%。Zarei和Asl[28]先对单导联ECG进行小波变换,然后从小波变换系数中提取非线性特征,接着利用序列前向选择进行特征选择,最后利用SVM进行分类。该方法识别OSA片段的准确度为94.63%,灵敏度为94.43%,特异度为94.77%;诊断OSA患者的准确度为95.71%,灵敏度为95.83%,特异度为95.66%。Dong等[29]基于R-R间期序列来识别OSA患者,识别准确度达90.10%,灵敏度为88.29%,特异度为90.50%。除了使用ECG信号,也有研究人员利用EEG信号来识别呼吸暂停。Bhattacharjee等[30]提出了一种基于多频子带帧的特征提取方法,捕获EEG数据帧中的特征,根据这些特征利用K近邻(K nearest neighbor,KNN)算法进行二分类,只划分SA和非SA事件。在多个的公开数据集上进行了算法测试,该算法与现有的基于相同数据集的算法相比,精确率相似,但是敏感性和准确度有很大的提升。Lee等[31]结合EEG、ECG和血氧饱和度以获取丰富的生理信息,运用机器学习的方法来检测睡眠呼吸暂停事件。文章将患病严重程度划分为二组三类:正常、异常(轻微、严重),提取统计特征后用方差分析进行特征选择,然后用决策树对OSA分组分类。
在睡眠医学中,除了识别患者是否患有睡眠呼吸暂停,还会利用呼吸暂停低通气指数(apnea-hypopnea index,AHI)来评判病症的严重程度。机器学习可以实现分类或回归,所以Jung等[32]从自采集数据集的单导联ECG信号中识别出呼吸信号,接着将呼吸周期变化系数作为预测因子,通过回归分析建立AHI预测模型。在Apnea-ECG数据库[33]上使用该回归模型预测AHI的绝对误差为(3.65±2.98)事件/h,AHI预测值与真实值之间的皮尔逊相关系数为0.97(P<0.01)。这是首个不需要整夜数据来预测AHI的研究,仅使用入睡阶段150 s的ECG信号来建立模型。
2.基于深度学习的睡眠呼吸暂停识别方法:Van Steenkiste等[34]用呼吸信号识别呼吸暂停事件,考虑了从胸腹部呼吸运动或ECG中获得的呼吸信号。对呼吸信号进行预处理并划分成片段后,研究者使用平衡自助法(balanced Booststrap)来解决数据不平衡问题,接着将平衡后的片段送入LSTM中进行分类。利用腹部呼吸绑带得到的呼吸信号,模型可实现77.2%的准确度、62.3%的灵敏度和80.3%的特异度;利用EDR,模型仅可实现60.1%的准确度、52.1%的灵敏度和61.8%的特异度。Faust等[35]先利用Ornstein-Uhlenbeck三阶高斯过程进行带通滤波,然后送入LSTM中分类。在Apnea-ECG数据库上,模型的准确度为99.80%,灵敏度为99.85%,特异度为99.73%。
3.融合深度学习和机器学习的睡眠呼吸暂停识别方法:考虑到深度学习适合用来自动提取特征,Li等[36]利用稀疏自编码器(sparse auto encoder,SAE)这种深度学习方法先从单导联ECG中提取特征,然后分别利用SVM和人工神经网络(artificial neural network,ANN)这两种机器学习的分类器对特征进行分类。考虑到时间相关性,接着将分类的结果送入HMM中,最终进行决策融合。模型识别含OSA的ECG片段时准确度为85%,灵敏度为88.9%;基于对OSA片段的识别,模型识别含OSA的记录时准确度可达100%。
快动眼睡眠行为障碍(REM sleep behavior disorder,RBD)是一种异态睡眠,特征是快速眼动睡眠期(rapid eye movement,REM)肌肉张力活动增多甚至伴发暴力活动及梦境回放。Milerska等[37]利用半自动的方式检测7例成人RBD患者肌张力减退不明显的REM睡眠(REM sleep without atonia,RSWA)。对肌电和ECG进行预处理和滤波后,使用滑动平均滤波器提取曲线的包络,然后识别ECG中的QRS复合波,同时识别EMG中的肌肉活动来检测RSWA,可得平均精度为(64.24±12.34)%,灵敏度为(91.63±10.07)%。
目前关于利用AI方法识别RBD方面研究较少,但是RBD中的伤害性梦境可能导致患者在睡眠中做出攻击性行为,如何利用AI方法快速精准地实现RBD患者睡眠动作影像的分析,为医生临床诊断提供支持仍有待进一步深入探讨。
尽管AI方法在自动睡眠分期、睡眠呼吸暂停识别等方面已取得了一定进展,但关于AI技术在自动监测失眠的相关工作却不多。Shahin等[38]将两通道EEG提取的57个特征送入深度神经网络(deep neural network,DNN)进行二分类识别,以准确地区分失眠患者和健康人。作者研究了两种不同的方法,第一种方法将整晚EEG数据作为输入送入网络,而第二种方法则先对睡眠阶段进行分类,然后再将分类的结果输入网络来区别失眠患者和健康人。第一种方法的最高准确度可达90%,第二种方法的最高准确度可达92%,也反映了利用深度学习实现失眠障碍辅助诊断的可行性。Yang和Liu[39]提出了一种基于单通道EEG的一维CNN(1D-CNN)模型来识别失眠障碍。该研究使用了9例失眠患者和9名健康受试者的单通道EEG。根据AASM标准,构造了4个子数据集:所有睡眠阶段数据集,REM睡眠阶段数据集,轻度睡眠阶段数据集(LSS数据集,包括S1和S2期)和深度睡眠阶段数据集(SWS数据集,包括S3和S4期)。随后,将4个子数据集输入到1D-CNN中,探讨和比较了不同睡眠阶段信号是识别失眠障碍的准确度。实验表明,利用REM,LSS和SWS时期组成的3个子数据集的1D-CNN获得了更高的平均准确度。其中,利用REM和SWS结合1D-CNN识别失眠障碍的平均准确度可分别达98.98%和99.16%。
由于失眠的异质性强,PSG对失眠障碍患者的病因监测及排除诊断非常重要,研究表明利用深度学习方法实现失眠障碍人群的自动识别的可行性,后期可深入研究失眠患者的电生理信号特征,利用深度学习算法辅助挖掘失眠的EEG生物标志物。
发作性睡病是一种罕见的慢性睡眠障碍,以REM睡眠异常为特征,表现为睡眠-清醒失调(白天嗜睡、猝倒、入睡幻觉、睡眠瘫痪和睡眠不安稳)以及运动、认知、精神、代谢和自主神经功能紊乱。发作性睡病可以分为发作性睡病1型和发作性睡病2型:发作性睡病1型会发生猝倒且脑脊液中下丘脑泌素-1(hypocretin-1,Hcrt-1)显著下降;而发作性睡病2型的多数患者脑脊液中Hcrt-1浓度无明显下降且其病理原因还需要探究。目前利用AI技术自动识别发作性睡病患者的研究较少,而且是针对病理研究较为成熟的发作性睡病1型。Zhang等[40]应用马尔可夫模型发现发作性睡病夜间睡眠结构紊乱,睡眠稳定性差,可能与下丘脑分泌素缺乏有关。为实现发作性睡病1型患者的识别,Stephansen等[41]先利用神经网络和集成学习对夜间PSG(EEG、EMG和EOG)实现自动睡眠分期,并提出hypnodensity graph从概率上表示睡眠分期结果,接着基于hypnodensity graph提取特征,建立识别发作性睡病1型的方法。睡眠分期的准确度可达87%。识别1型发作性睡病的特异度达96%,灵敏度达91%。
目前AI技术在睡眠医学领域的研究主要实现了睡眠阶段的分类、睡眠微事件、睡眠呼吸暂停、发作性睡病等识别。从数据层面上来看,移动互联网技术促进了医疗服务信息化的快速发展,积累了种类繁多且具有研究价值的临床数据。然而,已有的研究工作较局限于公开数据库,尽管准确度较高,但缺少在大量的临床样本上进行验证,使得所建立的模型/系统泛化能力受到限制,尚未能在实际应用中取得预期效果。从方法层面上来看,机器学习、深度学习等技术使得模型/系统能够处理和应对更趋复杂的问题,但往往会忽视临床的可解释性和可使用性。诸如,深度学习模型,能够实现端到端的映射,但所提取的网络特征较为抽象,缺乏临床依据,不易被专业医生接受;同时,现有的多数睡眠事件识别算法仅能实现单一类别的事件识别,对不同睡眠事件需采用不同的识别方法;而且,多数睡眠事件识别算法仅能实现睡眠事件有无的判别,对于睡眠事件的其他信息如睡眠事件的发生时刻、持续时间等并没有涉及。从应用层面上来看,睡眠健康的需求正逐步趋于多样化和多层次化,不仅体现在睡眠相关疾病的研究、诊断与治疗上,同时也与不同年龄层、不同行业的普通人群息息相关。如何借助大数据平台、互联网技术,将AI技术从临床应用进一步拓展到个人的睡眠健康管理等领域,将具有重要的社会意义和应用价值。
综上所述,未来的AI技术在睡眠医学领域的发展需构建睡眠大数据平台,从临床的实际需求出发,对大量的异构数据进行分析并提取具有临床可解释性的特征,同时融合医生的专业技能与经验,提升模型/系统的泛化能力,以充分发挥AI在大数据分析和智能决策上的优势,且能够得以逐步推广与应用,进而促进全民睡眠健康事业的发展。
所有作者均声明不存在利益冲突



























