
筛选急性髓系白血病(AML)潜在的生物标志物和药物,以期提高白血病的治愈率。
从基因表达综合(GEO)数据库下载包括AML患者和健康供者的基因芯片GSE90062数据集(包括3个白血病干细胞样本和3个正常骨髓造血干细胞样本)和GSE17054数据集(包括9个白血病干细胞样本和4个正常骨髓造血干细胞样本)。通过GEO2R软件分析GEO数据库两数据集AML患者和健康供者骨髓之间的共同差异表达基因(DEG)。利用DAVID在线软件对共同DEG进行基因本体(GO)生物功能富集分析和京都基因与基因组百科全书(KEGG)通路富集分析,利用STRING数据库构建蛋白相互作用(PPI)网络,借助CytoHubba插件筛选相关强度前10的关键基因。应用基因表达谱交互分析(GEPIA)数据库从10个关键基因中再次确认关键基因;采用Kaplan-Meier生存曲线评估10个关键基因不同水平AML患者的预后,比较采用log-rank检验;利用药物-基因相互作用数据库(DGIdb)筛选关键基因相关药物。
从GEO数据库筛选出AML患者骨髓中较健康供者骨髓上调的共同DEG 75个和下调的共同DEG 61个。在GO富集分析中,共同DEG主要与细胞凋亡、细胞迁移相关;在KEGG通路分析中,这些基因主要与细胞凋亡、造血通路相关。经PPI网络筛选出的强相关性前10个关键基因是ITGA4、ITGAL、HNRNPA3、CDC42、PRF1、SRSF3、HNRNPD、GTPBP4、CXCR4、RPL35A,其中6个基因(ITGA4、ITGAL、HNRNPA3、GTPBP4、CXCR4、RPL35A)通过GEPIA2数据库再次确认。Kaplan-Meier生存曲线分析显示,与ITGAL低表达患者相比,ITGAL高表达AML患者总生存不佳,差异有统计学意义(P=0.010);其余9个关键基因高表达与低表达患者间总生存差异均无统计学意义(均P>0.05)。从DGIdb筛选出4种可能与ITGAL相关的药物,分别为efalizumab、odulimomab、lifitegrast、rovelizumab。
ITGAL在AML患者骨髓中过表达,是AML患者的不利预后因素。筛选出的靶向ITGAL的潜在药物efalizumab、odulimomab、lifitegrast、rovelizumab为AML的治疗提供了新思路。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
急性髓系白血病(AML)是起源于造血干细胞的恶性克隆性疾病,异质性强,预后差[1,2]。因此,寻找新的分子生物标志物对研究AML的发病机制和寻找新的治疗方式具有重要意义。当前"旧药新用"受到极大关注。"旧药"为美国食品药品管理局(FDA)批准的药物或临床已安全应用的药物,如阿司匹林是一种具有抗炎和镇痛作用的常用药物,广泛应用于降低心脑血管疾病的风险,然而,有证据表明,定期服用阿司匹林可以降低前列腺癌发生的风险[3]。本研究采用生物信息学方法分析公共数据库数据,探寻AML的潜在生物标志物和可用药物,为AML的诊断和治疗提供新思路。
从基因表达综合(GEO)数据库(https://www.ncbi.nlm.nih.gov/geo/)的基因表达数据(包括高通量数据、杂交阵列和微阵列数据)检索含有AML患者和健康供者的基因芯片,包括GSE90062(2017年发布)和GSE17054(2009年发布)两个数据集。其中,GSE90062包括3个白血病干细胞样本和3个正常骨髓造血干细胞样本,GSE17054包括9个白血病干细胞样本和4个正常骨髓造血干细胞样本;样本未进行法美英(FAB)分型。
通过GEO2R在线工具(https://www.ncbi.nlm.nih.gov/geo/geo2r/)筛选GEO数据库AML和健康供者样本之间的DEG,并计算log2FC(FC为变化倍数)和P值,采用t检验验证DEG,设定筛选条件为|log2FC|>1,P<0.05为差异有统计学意义。再通过Venn制图工具(http://bioinformatics.psb.ugent.be/webtools/Venn/)筛选出GSE90062和GSE17054两个数据集的共同DEG。logFC<-1代表下调基因,logFC>1代表上调基因。
基因本体(GO)富集用于分析DEG的生物学特性[4],京都基因与基因组百科全书(KEGG)通路数据库存储了大量关于基因组、生物途径、疾病、化学物质和药物的数据[5]。通过DAVID在线软件(https://david.ncifcrf.gov)对筛选出的共同DEG进行GO生物功能富集分析和KEGG信号通路富集分析(以P<0.05为差异有统计学意义)。
将共同DEG谱上传至STRING数据库(https://cn.string-db.org/),构建共同DEG的PPI网络。通过Cytoscape软件(http://www.cytoscape.org/)进行可视化分析。因具有较高连通度的节点在维持整个网络的稳定性方面更为重要,使用Cytoscape软件中的CytoHubba插件,计算每个蛋白质节点的分值,筛选出关联强度排前10的关键基因。
GEPIA具有差异表达分析、生存分析和类似基因鉴定等功能,通过GEPIA2网站可对癌症基因组图谱(TCGA)数据库和基因型组织表达(GTEx)数据库共9 736个肿瘤样本、8 587个正常样本的RNA-seq表达数据进行分析[6]。在GEPIA2网站(http://gepia2.cancer-pku.cn/)中,将筛选出的10个关键基因上传,从中再次筛选AML关键基因,绘制再筛选的关键基因表达的箱线图;采用Kaplan-Meier生存曲线评估10个关键基因不同水平AML患者的总生存,组间比较采用log-rank检验。
药物-基因相互作用数据库(DGIdb)(https://dgidb.genome.wustl.edu/)整合了来自多种渠道的药物与基因间的相互作用等信息[7]。将1.4中筛选出的10个关键基因上传到DGIdb中,从现有药物中筛选AML潜在药物。
通过GEO2R工具,从GEO数据库中的GSE90062基因芯片数据集中提取了1 857个上调基因和1 103个下调基因,从GSE17054基因芯片数据集中提取了829个上调基因和2 591个下调基因。经Venn分析,获得两数据集的DEG交集,包括136个共同DEG,其中75个上调基因(MYO1B、GPR107、TIA1、CLHC1、NETO2、CLN8、TIGAR、DOCK8、PAG1、MEF2A、WT1AS、TSGA10、ZNF641、CEL、CARS、ADRB1、TANK、DNAJB4、MAP1LC3B、IL13RA1、ATXN3、SETD5、CYP2E1、PRF1、CD9、ADGRE2、ITPR2、SEMA4B、FANCF、MR1、DUSP10、SEL1L、RNF213、INTS7、RABIF、ERCC4、KIAA0513、CPEB2、SEPSECS、IL3RA、ZNF322、ST20、LAT、LPAR5、NTN1、MICAL2、RNASEL、HMGCL、PMAIP1、PCMTD1、GIN1、GSDMB、MRPS16、CEP19、RIT1、NABP1、PANK2、GAS7、PLPP1、FOXP1、RPAP2、HIST1H2BD、CACNA2D4、KLF13、GPT2、CFP、KCTD21、WNK1、THAP6、ZBTB46、ITGAL、CNST、DCLRE1C、KLHL24、CTSC),61个下调基因(CXCR4、BIRC5、TMEM200A、PHACTR1、DHCR7、PROS1、COCH、SRSF3、LRRFIP1、ZBTB24、FAM13A、ZNF165、ABCD3、WDR61、HNRNPD、CNTLN、FMR1、GFOD1、MPHOSPH6、MPP6、TMEM97、METAP2、L3HYPDH、RPL35A、ASXL1、CCP110、RRP15、C8orf33、SFXN2、DACH1、SRI、TRAF3、HSPA4L、C12orf29、TM2D3、CHCHD7、ZC3HAV1、TIMM10B、COX20、LIN7A、PRPS1、CDC42、SLC35G1、ARRB1、RRP1B、ITGA4、HMGN5、SESN3、CD59、INPP4B、IFI6、SNRPA1、PCGF5、GTPBP4、ANKRD13C、BAG2、GCFC2、TMEM201、FERMT1、HNRNPA3、TOE1)。
对136个共同DEG进行GO富集分析显示,对于生物学过程(BP),这些共同DEG主要集中在细胞凋亡过程、GTP酶活性负调控、整合素介导的信号通路、DNA损伤的细胞反应、选择性谷氨酸受体活性的调控、细胞-基质黏附、磷酸酶活性负调控、Cdc42蛋白信号转导、树突细胞迁移、链间交联修复、细胞对病毒的反应、蛋白质泛素化的负调控、细胞质转化的负调控、T细胞介导的细胞毒性;对于细胞成分(CC),共同DEG主要富集于细胞质、前缘膜、胞液、信使核糖核蛋白复合体、细胞膜、核质、中心粒、内质网膜、突触、树突棘、Cdc73/Paf1复合体;对于分子功能(MF),共同DEG主要集中于离子通道连接、泛素蛋白连接酶结合、细胞因子结合、RNA结合、分子伴侣结合、磷酸酶结合、大孔道通路活性。对136个共同DEG进行KEGG信号途径分析显示,这些DEG主要富集于细胞凋亡、造血通路中。其中,GO富集分析显示,ITGAL基因参与BP中的整合素介导的信号通路、细胞-基质黏附,CC中的细胞膜,MF中的蛋白结合信号通路(表1)。
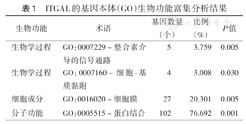
ITGAL的基因本体(GO)生物功能富集分析结果
ITGAL的基因本体(GO)生物功能富集分析结果
| 生物功能 | 术语 | 基因数量(个) | 比例(%) | P值 |
|---|---|---|---|---|
| 生物学过程 | GO:0007229~整合素介导的信号通路 | 5 | 3.759 | 0.005 |
| 生物学过程 | GO:0007160~细胞-基质黏附 | 4 | 3.008 | 0.030 |
| 细胞成分 | GO:0016020~细胞膜 | 27 | 20.301 | 0.005 |
| 分子功能 | GO:0005515~蛋白结合 | 102 | 76.692 | 0.001 |
应用STRING数据库预测共同DEG之间的PPI网络见图1A,关联强度前10的关键基因是ITGA4、ITGAL、HNRNPA3、CDC42、PRF1、SRSF3、HNRNPD、GTPBP4、CXCR4、RPL35A(图1B)。


注:GEO为基因表达综合;AML为急性髓系白血病;DEG为差异表达基因;PPI为蛋白相互作用;图1A中为具有潜在交互作用的共同DEG,剔除了边缘DEG;粉色节点表示上调基因,蓝色节点表示下调基因
将10个关键基因上传到GEPIA2网站,其中6个基因(ITGA4、ITGAL、HNRNPA3、GTPBP4、CXCR4、RPL35A)通过GEPIA分析得到再次确认,ITGA4、ITGAL、CXCR4在AML样本中表达明显高于正常样本,HNRNPA3、GTPBP4、RPL35A表达明显低于正常样本,差异均有统计学意义(均P<0.05)(图2)。通过Kaplan-Meier生存曲线分析显示,与ITGAL低表达的AML患者相比,ITGAL高表达的患者总生存不佳,差异有统计学意义(P=0.010)(图3);其余9个关键基因高表达与低表达患者间总生存差异均无统计学意义(均P>0.05)。
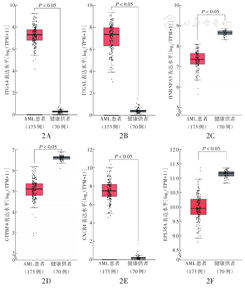

注:GEPIA为基因表达谱交互分析;AML为急性髓系白血病;TPM为每千个碱基的转录每百万映射读取的转录本
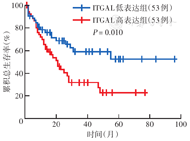

注:GEPIA为基因表达谱交互分析;TCGA为癌症基因组图谱;GTEx为基因型组织表达;AML为急性髓系白血病
将筛选的10个关键基因上传到DGIdb进行分析,验证出ITGA4、ITGAL、CXCR4基因与15种药物相匹配(表2)。其中,ITGAL与4种药物匹配,分别为efalizumab、odulimomab、lifitegrast、rovelizumab。
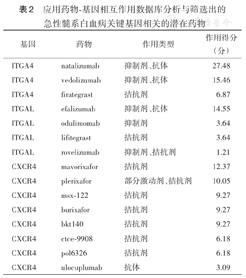
应用药物-基因相互作用数据库分析与筛选出的急性髓系白血病关键基因相关的潜在药物
应用药物-基因相互作用数据库分析与筛选出的急性髓系白血病关键基因相关的潜在药物
| 基因 | 药物 | 作用类型 | 作用得分(分) |
|---|---|---|---|
| ITGA4 | natalizumab | 抑制剂、抗体 | 27.48 |
| ITGA4 | vedolizumab | 抑制剂、抗体 | 15.46 |
| ITGA4 | firategrast | 拮抗剂 | 6.87 |
| ITGAL | efalizumab | 抑制剂、抗体 | 14.55 |
| ITGAL | odulimomab | 抑制剂 | 3.64 |
| ITGAL | lifitegrast | 拮抗剂 | 3.64 |
| ITGAL | rovelizumab | 抑制剂、拮抗剂 | 1.21 |
| CXCR4 | mavorixafor | 拮抗剂 | 12.37 |
| CXCR4 | plerixafor | 部分激动剂、拮抗剂 | 10.05 |
| CXCR4 | msx-122 | 拮抗剂 | 9.27 |
| CXCR4 | burixafor | 拮抗剂 | 9.27 |
| CXCR4 | bkt140 | 拮抗剂 | 9.27 |
| CXCR4 | ctce-9908 | 拮抗剂 | 6.18 |
| CXCR4 | pol6326 | 拮抗剂 | 6.18 |
| CXCR4 | ulocuplumab | 抗体 | 3.09 |
目前AML的治疗方法主要有化疗、靶向治疗、造血干细胞移植和免疫治疗等,尽管近年来治疗方式不断发展,但AML患者的5年生存率仍在30%左右,主要原因是复发率高、难治率高[8],且存在不良反应大、耐药等问题[9,10]。因此,迫切需要寻找新的分子生物标志物,探索AML的分子机制,以指导AML的早期诊断和治疗。随着生物信息学方法的快速发展,有越来越多的数据库和分析数据的手段可供选择,为筛选基因改变和寻找新的分子生物标志物提供了便捷的方式。本研究从GEO数据库中选取AML患者的基因表达芯片,通过GEO2R工具分析GSE90062和GSE17054数据集,以识别AML样本和健康供者样本之间的DEG,最终共筛选出两个数据集136个共同DEG。通过对共同DEG构建PPI网络,筛选出关联强度前10位的关键基因,进一步分析显示,与ITGAL低表达的AML患者相比,ITGAL高表达的患者总生存差,提示ITGAL与AML的进展具有相关性。
ITGAL是整合素家族的成员,其异常表达与免疫调节和致癌有关。遗传关联研究表明,ITGAL表达的增高会增加炎症性肠病发生的风险[11]。我国学者通过实验证明胃癌样本中的ITGAL表达水平显著高于肿瘤周围样本,且ITGAL与包括程序性死亡受体1(PD-1)在内的各种免疫标志物有密切关系[12]。另外,有研究显示ITGAL可能通过调节细胞因子的产生和骨髓源性抑制细胞的数量,在AML中发挥作用[13]。本研究的GO富集分析表明,ITGAL基因参与整合素介导的信号通路、细胞-基质黏附,参与组成细胞膜成分的构成,与蛋白结合信号通路密切相关,可能诱导AML的侵袭和转移。
尽管ITGAL在疾病发生中起着重要作用,但只有少数药物被设计靶向这一基因。本研究采用生物信息学方法从"旧药"中筛选、识别新的抗肿瘤药物,发现ITGAL与4种药物相匹配,即efalizumab、odulimomab、lifitegrast、rovelizumab。
efalizumab是一种针对CD11a的重组人源化单克隆抗体,可抑制淋巴细胞活化,并减少淋巴细胞向炎症部位迁移,具有强大的免疫抑制活性,于2003年获美国FDA批准用于中度至重度慢性斑块状银屑病的治疗。但在后续临床观察中,长期治疗的患者出现了严重不良事件,于2009年被暂停上市许可[14]。odulimomab是一种抗淋巴细胞功能相关抗原1(LFA-1)的单克隆抗体,与LFA-1的a亚单位(CD11a)结合并抑制T细胞的活化,其被批准用于银屑病治疗的Ⅳ期试验,且对缺血和再灌注损伤具有显著的保护作用,可减少移植后肾功能的损害[15]。lifitegrast是一种新型T细胞整合素拮抗剂,通过阻断T细胞上的LFA-1和细胞间黏附分子1(ICAM-1)的相互作用而起效,于2016年被美国FDA批准用于干眼症的治疗[16,17]。rovelizumab是一种针对CD11/CD18细胞黏附蛋白的单克隆抗体,与白细胞整合素受体结合,可阻止中性粒细胞在中枢神经系统的迁移和黏附。这些药物都是针对LFA-1的拮抗剂,LFA-1介导免疫系统中的白细胞和非血细胞之间的相互作用[18],在免疫系统调节中起着重要作用[19],也许可以通过阻断LFA-1与其配体的结合来实现抗白血病作用,这为AML的治疗提供了新思路。
本研究基于从GEO数据库获得的基因表达数据集筛选了136个在AML和健康供者之间共同DEG,并从中分析出10个可能是AML的核心基因。ITGAL可能与AML的发生和预后相关,可能是AML诊断和预后判断的潜在生物标志物。此外,本研究通过筛选的关键基因有针对性地预测了潜在药物,为AML的治疗提供了新思路。但本研究仅涉及生物信息学分析,存在一定的偏差,需要进一步的实验模型来验证研究结果。
林丹霞,胡嘉升.生物信息学方法挖掘急性髓系白血病的生物标志物和相关药物[J].白血病·淋巴瘤,2024,33(5):288-293. DOI:10.3760/cma.j.cn115356-20230414-00089.
所有作者声明无利益冲突



























