
应用新一代长读长测序技术精准确定复制缺陷型痘苗病毒天坛株(non-replicating Tiantan strain of vaccinia virus,NTV)全基因组序列及其开放阅读框(ORFs)组成。
基于本实验室储备的NTV,在鸡胚成纤维细胞上进行扩增并纯化,提取NTV全长基因组核酸。PacBio HiFi测序从头组装获取NTV全长基因组序列。以同源注释的策略确定其ORF组成,并分析其与已知复制缺陷型痘苗病毒毒株ORF异同。
NTV全长共171 729 bp,GC含量为33%,其独特的倒置末端重复(ITR)区域包含发夹结构、两个串联重复区域及3个非重复区域。NTV共有166个ORFs,与第三代天花病毒疫苗改良安卡拉株(MVA-BN)和复制缺陷型哥本哈根株(NYVAC)株相比,主要差异位于ITR及其周围区域,3个毒株共同享有138个ORFs,NTV特有6个编码与病毒逃避宿主抗病毒反应相关的ORFs。
通过三代测序技术精准确定NTV全基因组序列及ORFs组成,并揭示其与其他复制缺陷型痘苗病毒毒株的异同,为新一代猴痘疫苗和痘苗病毒载体研发应用提供重要参考。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
2022年以来,由猴痘病毒感染引起的猴痘(Mpox)暴发流行已成为全球关注的公共卫生事件。2022年9月中国内地(不含港澳台)检测到首例输入性猴痘病例[1],2023年我国几个城市和地区陆续报道了关于猴痘感染的确诊病例[2],截至2023年12月23日,我国累计报道猴痘确诊病例为2 025例。全球117个国家和地区累计报道超过9万例确诊病例及176例死亡病例(https://worldhealthorg.shinyapps.io/mpx_global)。然而,目前针对Mpox仅可通过抗病毒药物进行对症支持治疗[1],因此对于Mpox预防显得格外重要。国际上已批准使用第三代天花病毒疫苗改良安卡拉株(MVA-BN)进行Mpox的预防,其毒力低,安全性更高,且更适用于免疫力低下人群[3]。研究报道称接种针对天花病毒研发的痘苗病毒疫苗可以提供对猴痘病毒感染的交叉免疫保护[4,5]。痘苗病毒天坛株(TTV)作为预防天花病毒的疫苗株已在中国人群中接种了数亿次。因其强免疫原性和低不良反应等优势备受青睐。在天花病毒被宣布消灭后,TTV仍被广泛用作基因表达载体和免疫基因疗法[6,7,8,9]。复制型痘苗病毒疫苗的不良反应包括皮肤损伤及神经系统症状[10],有研究通过剔除某些毒力相关基因和引入带有调节免疫应答的淋巴因子改造痘苗病毒,希望可以在不影响其免疫效果的同时降低它的毒力,然而,按照经典接种方案很难确保在保留其免疫效果的同时减少免疫应答引起的并发症,因此用"非复制型痘苗病毒载体理论"构建了复制缺陷型哥本哈根株(NYVAC)。虽然NYVAC毒副作用大幅度降低但是其在鸡胚细胞中繁殖的滴度也很低。我国研究人员以TTV为基础删除了部分宿主范围及毒力相关的基因,构建了一种复制缺陷型痘苗病毒天坛株(NTV),使其在鸡胚成纤维细胞(CEFs)中具有很强的复制能力和高效传播能力,但无法在人源细胞中包装和产生后代病毒,具有良好的DNA复制、RNA转录与蛋白质翻译功能,可用于外源基因表达载体和新一代疫苗研究[11]。
NTV作为病毒载体已在多领域进行了广泛的应用[12],其与复制型病毒载体表达外源抗原水平相当[13];经腹腔免疫小鼠后诱导的针对目的蛋白的体液免疫水平与重组复制型TTV载体诱导的免疫水平无明显差异[14];重组NTV经皮免疫家兔后可稳定诱发针对痘苗病毒特异性IgG抗体[15]。复制缺陷型疫苗载体具有低毒力、可有效表达外源抗原、不具备传染性等优势,作为新一代正痘病毒疫苗候选株,NTV备受重视[16]。然而NTV全基因组序列尚未进行报道和公布,基因工程删除基因片段所减少编码开放阅读框(ORFs)的功能和影响还未完全验证,需要更深入的探索,故精准测定并注释NTV全基因组是研究的基础。正痘病毒基因组由两端倒置末端重复序列(ITR)区域和中央保守区域组成[17]。使用短读长测序技术无法精确确定ITR区域的串联重复结构和数量。因此,我们希望借助长读长测序技术的优势精准获悉NTV全基因组序列及其ORFs组成,为NTV的基础研究和开发应用提供理论参考。
CEFs细胞采购于北京勃林格殷格翰维通生物技术有限公司,由8日龄SPF级鸡胚制备而成。
NTV由中国疾病预防控制中心病毒病应急技术中心构建。以本实验室保存的预防天花的疫苗毒株TTV(批号7601,从疫苗储备液中挑选出具有代表性的单克隆毒株)为重组用母病毒,pNeoCKLac为重组用质粒,采用蓝白斑双重标记筛选,先挑选出蚀斑为蓝色的NTVLac;再以NTVLac为重组用病毒,挑选出蚀斑为白色的遗传稳定的单克隆病毒,命名为NTV[11]。NTV在TTV基础上缺失了HindⅢ内切酶标记的20个ORFs(共计约21 kb),具体包括C区14个ORFs:C12L、C11R、C10L、WR011、WR013、WR018、C9L、C8L、C7L、C6L、C5L、C3L、C2L、C1L;N区2个ORFs:N1L、N2L;M区2个ORFs:M1L、M2L;K区2个ORFs:K1L、K2L(以哥本哈根株为参考对基因进行命名,部分哥本哈根株未注释的ORFs依据WR株同源ORFs来命名)。
取储备的NTV病毒液以1∶100接种在CEFs上,置于37℃,5%CO2的孵箱中培养,孵育2 h后记为0时,换2%的维持液(MEM+2%FBS)继续培养72 h,将细胞刮下收集至离心管中,离心去上清后加入5 ml PBS重悬,置于-80℃冰箱。
将细胞沉淀取出反复冻融3次,细胞病毒混合液以60瓦超声4 s间隔8 s,共计30个循环;加入1/4无EDTA胰酶37℃下消化30 min,中途轻轻摇晃;4℃下2 500 r/min离心10 min,取上清至新的离心管中;10 mmol/L Tris-HCl(pH9)洗涤沉淀3次,收集所有上清;将病毒上清转移至含有36%蔗糖溶液的超速离心管中,4℃下13 500 r/min超速离心90 min,弃上清,PBS重悬沉淀,置于-80℃备用。
纯化后的病毒与500 μl裂解缓冲液(Buffer AL,德国QIAGEN)、5 μl 2-巯基乙醇(美国GIBCO™)和20 μl的蛋白酶K(德国QIAGEN)混合,置于1.5 ml低DNA吸附管中。上述实验均在BSL-2实验室进行。将混合物在56℃金属浴中孵育10 min;加入400 μl DNA提取液(苯酚、氯仿和异戊醇,比例为25∶24∶1);轻轻混合,4℃下13 000 r/min离心10 min;将含有DNA的分层转移到低DNA吸附管中;重复上述步骤两次;向管中加入等体积的氯仿-异戊醇混合物(24∶1),充分混合;4℃下13 000 r/min离心10 min。将上清液转移到15 ml离心管中,加入1/10体积的醋酸钠溶液(pH5.2)。轻轻搅拌以促进DNA沉淀。加入2.5倍体积的冰浴无水乙醇,-20℃冰箱过夜;预冷离心机至4℃,12 000 r/min离心10 min,弃上清;加入1 ml冰浴的70%乙醇重悬沉淀。再次在4℃下12 000 r/min离心3 min;重复冲洗2次,弃上清;随后用20 μl无核酸酶的水溶解沉淀,得到DNA产物,-20℃冰箱保存。使用NanoDrop(美国Thermo)和Qubit (美国Thermo)测量DNA产物浓度。
将全长基因组DNA运输至武汉未来组生物科技有限公司进行PacBio HiFi测序,返回原始测序数据。
下机数据使用CCS[18]转换为FastQ格式;使用FastQC[19]进行质量控制评价;FastP[20]过滤掉过短片段、低质量片段以及测序接头;随后使用Canu[21]对测序数据进行从头组装;以生成的一致性序列为参考基因使用Minimap2[22]将测序原始数据进行回帖比对;通过SAMtools[23]将比对后的文件进行转换和排序;利用IGV[24]可视化直观评价组装效果,使用Freebayes[25]寻找存在争议的位点,进行序列抛光,最终获得完整的全基因组序列(图1)。序列已提交至国家基因组科学数据中心(NGDC),序列编号为C_AA058750.1。
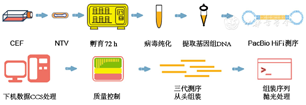

注:CEF:鸡胚成纤维细胞;NTV:复制缺陷型痘苗病毒天坛株
利用GATU[26]基因组同源注释软件,以痘苗病毒哥本哈根株(M35027)、WR株(NC_006998)、李斯特株(AY678276)、安卡拉株(AM501482)为参考序列,ORFs相似度达98.5%进行保留。
NTV全基因组测序与分析参见图1,所获得的NTV基因组序列全长171 729 bp,GC含量33%。NTV独特的ITR结构为9.2 kb,由发夹结构、2组串联重复区域(TR)和3组非重复区域(NR)组成。以第一个错配残基"T"为序列起始位点[25],发夹结构为101 bp(1~101 bp、171 629~171 729 bp);NRⅠ为86 bp(102~187 bp、171 543~171 628 bp);TRⅠ是由70 bp为单位的片段递归排列组成,共计重复23次;NRⅡ由720 bp组成(1 796~2 515 bp、169 215~169 934 bp);TRⅡ包含54 bp为单位片段的54次重复;NRⅢ(5 736~7 107 bp、162 478~165 994 bp)在两侧是不同的,左侧NRⅢ在基因工程改造时被人为删除了部分片段。
应用广泛的3种复制缺陷型痘苗病毒(NTV、NYVAC和MVA-BN)ITR结构存在明显差异。NTV发夹结构类似于"S"型发夹[26](图2A),而NYVAC发夹结构类似于"F"型发夹[27,28],MVA-BN的发夹结构为165 bp;NRⅠ区域包含"AT-rich sequence"被认为对病毒复制至关重要[29],3个毒株在此区域完全保守;在TRⅠ,NYVAC包含70 bp为单位的片段累计37次重复,MVA-BN包含70 bp为单位的片段累计13次重复;以"DdeⅠ"和"AluⅠ"出现的位置定位NRⅡ所在,NTV与NYVAC在NRⅡ保持完全一致,值得注意的是,MVA-BN中NRⅡ的划分并不是很清晰,根据文献报道,此NR区域被定位在1 191~1 395 bp[30],仅包含"DdeⅠ"酶切位点;NYVAC在TRⅡ表现为54 bp单元的片段重复排列9次,而MVA-BN在此处表现出高度复杂性,由多组54 bp重复单元和112 bp重复单元交叉组成;NRⅢ是ITR组份中差异最大的部分,编码一定数量的ORFs,是展现不同痘苗病毒各自特性重要的编码区域,NTV的NRⅢ为3.5 kb,MVA-BN的NRⅢ为3.8 kb,而NYVAC却长达8 kb(图2B)。
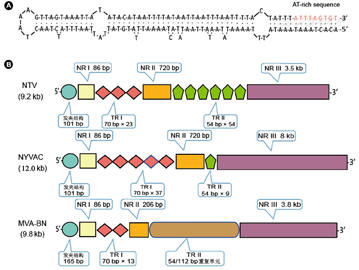

注:A:复制缺陷型痘苗病毒天坛株(NTV)的发夹结构始于序列中第一个错配残基"T"的位置;B:3株复制缺陷型痘苗病毒倒置末端重复序列(ITR)精细结构。NYVAC:复制缺陷型哥本哈根株;MVA-BN:第三代天花病毒疫苗改良安卡拉株
3株复制缺陷型痘苗病毒基因组间差异具体表现为ORFs组成的不同,这些差异ORFs编码的蛋白质涉及病毒宿主范围、复制转录翻译能力及毒力大小等方面。采用同源注释的方式为NTV确定了166个ORFs,NYVAC在哥本哈根株基础上删除了部分基因,最终编码241个ORFs,而MVA-BN在安卡拉株的基础上天然缺失多个片段,最终编码158个ORFs(表1)。痘苗病毒各代表株间主要差异位于ITR及其周围区域(图3A),解析存在差异的ORFs是研究毒株间生物学差异的重要途径。
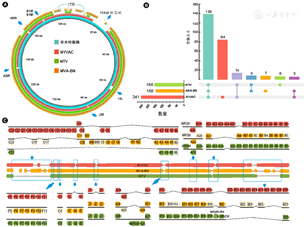

注:A:以哥本哈根株作为参考序列将复制缺陷型痘苗病毒天坛株(NTV)、第三代天花病毒疫苗改良安卡拉株(MVA-BN)和复制缺陷型哥本哈根株(NYVAC)进行多序列比对;B:3株复制缺陷型痘苗病毒ORFs组成差异汇总;C:3株复制缺陷型痘苗病毒ORFs组成差异在基因组分布示意图

NTV、MVA-BN和NYVAC相比于野生株比较ORFs缺失/失活汇总
Comparison of ORF deletions/inactivations in NTV, MVA-BN, and NYVAC compared to the wild type strain
NTV、MVA-BN和NYVAC相比于野生株比较ORFs缺失/失活汇总
Comparison of ORF deletions/inactivations in NTV, MVA-BN, and NYVAC compared to the wild type strain
| ORFs | NTV | MVA-BN | NYVAC |
|---|---|---|---|
| C23L | + | 伪基因 | + |
| CVA002 | / | 伪基因 | / |
| CVA003 | / | 伪基因 | / |
| C19L | / | 伪基因 | + |
| C18L | / | - | + |
| CVA005 | / | 伪基因 | / |
| C17L | / | 伪基因 | + |
| CVA006 | / | 伪基因 | / |
| CVA007 | / | 伪基因 | / |
| CVA008 | / | 伪基因 | / |
| CVA009 | / | - | / |
| C12L | - | - | + |
| C11R | - | + | + |
| C10L | - | + | + |
| WR011 | - | 伪基因 | / |
| CVA014 | / | 伪基因 | / |
| CVA015 | / | 伪基因 | / |
| WR013 | - | + | / |
| CVA017 | / | 伪基因 | / |
| CVA018 | / | 伪基因 | / |
| CVA019 | / | 伪基因 | / |
| CVA020 | / | 伪基因 | / |
| CVA021 | / | 伪基因 | / |
| WR018 | - | + | / |
| C9L | - | 伪基因 | + |
| CVA023 | / | 伪基因 | / |
| CVA024 | / | 伪基因 | / |
| C8L | - | + | + |
| C7L | - | + | - |
| C6L | - | + | - |
| C5L | - | - | - |
| C4L | / | - | - |
| C3L | - | - | - |
| C2L | - | - | - |
| C1L | - | - | - |
| N1L | - | + | - |
| N2L | - | + | - |
| M1L | - | - | - |
| M2L | - | - | - |
| K1L | - | - | - |
| K2L | - | + | + |
| F5L | + | 伪基因 | + |
| F11L | + | 伪基因 | + |
| O1L | + | 伪基因 | + |
| I4L | + | + | - |
| J2R | + | + | - |
| A25L | / | 伪基因 | + |
| A26L | + | + | - |
| A39R | + | 伪基因 | + |
| A56R | + | + | - |
| B2R | + | 伪基因 | + |
| B4R | + | 伪基因 | + |
| B8R | + | 伪基因 | + |
| B13R | + | 伪基因 | - |
| B14R | + | 伪基因 | - |
| B19R | + | 伪基因 | + |
| CVA212 | / | - | / |
| CVA213 | / | - | / |
| CVA214 | / | - | / |
| CVA215 | / | - | / |
| CVA216 | / | - | / |
| CVA217 | / | 伪基因 | / |
| CVA218 | / | - | / |
| CVA219 | / | - | / |
| CVA220 | / | - | / |
| B21R | / | - | + |
| CVA223 | / | 伪基因 | / |
| CVA224 | / | 伪基因 | / |
| B24R | / | - | + |
| B25R | / | 伪基因 | + |
| CVA227 | / | - | / |
| CVA228 | / | - | / |
| B29R | + | 伪基因 | + |
注:ORFs:开放阅读框;NTV:复制缺陷型痘苗病毒天坛株;MVA-BN:第三代天花病毒疫苗改良安卡拉株;NYVAC:复制缺陷型哥本哈根株;"-"代表此ORF在野生株基础上缺失;"+"代表此ORF在对应的复制缺陷型毒株中可编码蛋白;"/"代表此ORF在野生株中不存在;"伪基因"代表此ORF被认定为失活
3株复制缺陷型痘苗病毒一致编码138个ORFs(图3B),NTV特有编码类A型包涵体蛋白、2型丝氨酸蛋白酶抑制剂样蛋白及锚蛋白样蛋白等与病毒逃避宿主抗病毒反应相关的6个ORFs。MVA-BN编码特有的7个ORFs,其中有2个ORFs与天花病毒孟加拉国变异株所编码的ORFs同源,其余ORFs与宿主范围、毒力等功能相关。NYVAC特有编码84个ORFs,其中占比最多为预测的ORFs、其次为两侧ITR,编码与宿主范围和毒力相关的ORFs(表2)。图3C详细直观地展示了3个毒株间全基因组ORFs组成差异,存在编码序列(coding sequence, CDS)异常的ORFs被认为是假性基因用虚线构画,核苷酸片段缺失处用虚线跨越。
NTV、MVA-BN和NYVAC各株特有的ORFs汇总
Compilation of the unique ORFs in NTV, MVA-BN and NYVAC
NTV、MVA-BN和NYVAC各株特有的ORFs汇总
Compilation of the unique ORFs in NTV, MVA-BN and NYVAC
| 痘苗病毒 | 开放阅读框 |
|---|---|
| NTV | VACWR148、VACWR161、B13R、B14R、ankyrin-like、B ORF C10L |
| MVA-BN | MVA007、MVA008、C7L、C6L、N1L、N2L、MVA188 |
| NYVAC(除预测ORFs外) | C22L、C21L、C20L、C19L、C18L、C17L、C16L、C15L、C14L、C13L、C12L、C9L、K6L、A25L、A54L、B20R、B21R、B23R、B24R、B25R、B26R、B27R、B28R |
注:NTV:复制缺陷型痘苗病毒天坛株;MVA-BN:第三代天花病毒疫苗改良安卡拉株;NYVAC:复制缺陷型哥本哈根株;ORFs:开放阅读框
随着猴痘病毒感染蔓延全球,针对预防正痘病毒的疫苗研发正在紧张地开展。MVA-BN自鸡胚成纤维细胞连续传代,为天然产生的复制缺陷型毒株,已投入使用。与之不同,NTV经基因工程改造而成。尽管原型株TTV已在中国人群中被广泛接种,其安全性和免疫原性均获肯定,但改造后的NTV仍需进一步评估其安全性和潜在影响。非复制型痘苗病毒以高安全性和低不良反应等优势成为新一代疫苗研发的主要基础,NTV也成为新一代疫苗研发的候选毒株和载体。本研究通过长读长测序技术精准获取NTV全长基因组序列并基于同源注释确定其ORFs组成,明确了3个复制缺陷型毒株的ORFs组成差异,为今后重组疫苗开发和病毒载体构建提供参考。
C11R与K2L在NTV中是缺失的,C11R编码表皮生长因子样蛋白(EGF-like protein),已有研究猜测,生成的多肽可能发挥生长因子的功能,介导病毒与表达EGF受体的细胞结合,有助于病毒刺激表皮细胞[31]。另一项研究提示:使用拮抗剂占据EGF受体,可使痘苗病毒感染被抑制[32],NTV在缺失C11R的情况下可能会减缓感染宿主细胞的速度,表现出较低的不良反应。另外,K2L编码3型丝氨酸蛋白酶抑制剂样蛋白,已有研究证实A56/K2复合物可抑制细胞膜的融合[33],减少感染细胞的病毒量,同时还可防止感染细胞形成合胞体[34]。
VACWR148、B13R和B14R仅在NTV中编码,因此它们并不是痘苗病毒所必需的。其中,VACWR148编码A型包涵体蛋白,其多肽结构被推测可以有效保护病毒颗粒,在感染后期发挥重要作用[35];B13R与B14R编码2型丝氨酸蛋白酶抑制剂样蛋白(SPI-2/CrmA),它能够特异性抑制细胞凋亡,具体来说,SPI-2/CrmA与1型半胱氨酸蛋白酶特异性结合抑制其活性,从而阻断细胞凋亡的发生[36],而启动细胞凋亡在防止病毒传播和扩散中至关重要,病毒通过编码SPI-2/CrmA为病毒复制和扩散提供了有利的环境,同时小鼠免疫试验证明,缺乏B13R基因后病毒诱导的抗体水平升高[37]。因此,在NTV进一步修饰中,可考虑失活上述ORFs来达到预期目的。
MVA-BN与NYVAC复制缺陷的机制已明确,课题组也对NTV复制机制展开了系列的探索。F17、A17和A27蛋白表达受到抑制,提示NTV复制缺陷机制可能与晚期蛋白表达受阻有关,且可能发生在细胞翻译阶段[38,39,40]。本研究精准获取NTV全长基因组序列并基于同源注释确定其ORFs组成,通过进一步明确不同复制缺陷型痘苗病毒宿主范围、毒力的差异及其分子机制,开展NTV的改造与应用评价,可为NTV开发作为新一代正痘病毒疫苗及免疫与基因治疗载体提供科学参考。
张钟贤,吴长城,李涵,等.基于新一代测序技术精准确定复制缺陷型痘苗病毒天坛株全基因组序列及其开放阅读框组成[J].中华微生物学和免疫学杂志,2024,44(6): 502-509. DOI: 10.3760/cma.j.cn112309-20240201-00049.
所有作者声明无利益冲突
感谢阮力研究员在实验上提供技术指导



























