
对从临床伤口标本中分离出的贪铜菌SZY C1进行形态学及分子生物学鉴定和分析,了解其微生物学特性及明确分类学地位。
将菌株SZY C1复苏培养后,依次进行形态学、生理生化鉴定、16S rRNA基因测序、全基因组测序,并应用生物信息学软件对其基因组和毒力基因进行分析。
菌株SZY C1为不发酵糖革兰阴性菌,菌体无鞭毛、不形成芽孢,可在哥伦比亚血琼脂平板上培养24 h呈现灰白色、圆形、突起、不透明、边缘整齐的菌落。基于16S rRNA基因序列分析结果显示,菌株SZY C1与耐金属贪铜菌(Cupriavidus metallidurans)的相似度为98.52%。菌株SZY C1测得基因组大小为5 515 517 bp,G+C含量为67.87%,全基因系统发育分析显示,菌株SZY C1与根际贪铜菌亲缘化关系最近,两者之间平均核苷酸一致性值84.76%,数字DNA-DNA杂交值为29.1%,低于原核生物物种的鉴定阈值。基因预测显示菌株SZY C1携带多种毒力基因、耐药基因和抗重金属基因。
根据表型和基因组分析,菌株SZY C1为贪铜菌属中潜在的新种。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
贪铜菌属(Cupriavidus)是一种条件致病性不发酵糖革兰阴性菌,隶属于变形菌门(Proteobacteria)、β-变形菌纲(Betaproteobacteria)、伯克霍尔菌目(Burkholderiales)、伯克霍尔德菌科(Burkholderiaceae),模式菌株是杀手贪铜菌(Cupriavidus necator)[1]。贪铜菌属具有复杂的分类历史,其最早是由Makkar和Casida于1987年发现并命名[2],且与2001年命名的罗尔斯顿属(Ralstonia)和沃特菌属(Wautersia)具有较近的亲缘关系[3]。在2004年,Vandamme和Coenye[4]通过基因组杂交分析发现,优养沃特菌(Wautersia eutropha)与杀手贪铜菌为同一菌种,且贪铜菌属享有命名优先权,因此,按原核生物命名法,原分类为沃特菌属的全部菌种均被分类到贪铜菌属。目前,该属内已有20个公认物种,其中与人类感染密切相关有央大贪铜菌(C.cauae)[5]、吉拉迪贪铜菌(C.gilardii)[6]、少见贪铜菌(C.pauculus)[7]等,可引起血流感染、肌肉脓肿、导管败血症和直肠周围炎症等[8,9]。此外,巴塞尔贪铜菌(C.basilensis)、耐金属贪铜菌(C.metallidurans)、呼吸道贪铜菌(C.respiraculi)和台湾贪铜菌(C.taiwanensis)也有从囊性纤维化患者的痰液标本中分离,但是否在慢性肺梗阻疾病发挥作用尚不明确[1]。
贪铜菌属微生物的生物学特征主要包括:革兰阴性杆菌、需氧、有周鞭毛;通常形成白色、光滑、边缘整齐的菌落;氧化酶和过氧化氢酶均阳性,不发酵葡萄糖产酸,能够耐受各种金属[10]。本研究从1例右足开放性距骨骨折的患者伤口拭子中分离1株不发酵糖革兰阴性菌,采用传统形态特征、生理生化表型特征和基因组测序等的多相分类方法确认为贪铜菌属潜在的新种,现报道如下。
菌株SZY C1分离自1名骨折患者清创术后伤口标本,采集伤口拭子直接涂片提示革兰阴性球杆菌,常规细菌培养检出1株革兰阴性球杆菌。
将分离纯化的菌株SZY C1分别接种于哥伦比亚血琼脂培养基、普通巧克力培养基、麦康凯培养基、MH培养基、LB培养基和R2A培养基中,放置在含35℃,5%CO2恒温箱培养24~48 h,每天观察平板中菌株的生长情况与菌落形态。参照文献[10]方法,对菌株SZY C1进行生长温度(10~55℃)、耐盐性(0%~7% NaCl)与pH(pH4~10)生长范围测试。SZY C1的初步鉴定还采用革兰染色、过氧化氢酶、氧化酶、吲哚试验等,API ZYM、API 20 NE试剂盒(bioMérieux,法国)手工生化鉴定,VITEK 2系统(bioMérieux,法国)进行全自动细菌生化鉴定,透射电子显微镜观察菌株SZY C1形态。
使用VITEK MS系统(bioMérieux,法国)和EXS3000质谱仪(中元汇吉生物技术股份有限公司,中国)进行质谱鉴定。将分离株SZY C1的菌体均匀涂布在靶板的靶点内,加入1 μl CHCA基质液,待样品干燥结晶后进行质谱鉴定。同时将菌株SZY C1的质谱数据上传至美国CDC microbenet在线数据库(https://microbenet.cdc.gov/)进行比对。
菌株SZY C1 DNA提取、16S rRNA基因的PCR扩增和测序参考文献[11]方案。使用DNAMAN v7.0版本软件拼接序列,同时采用NCBI数据库、EzBioCloud数据库[12]对16S rRNA基因序列进行相似性分析。从NCBI网站中下载与菌株SZY C1高度相似菌种的16S rRNA基因序列,MEGA 11.0软件中的Neighbor-Joining法和Maximum-likelihood法构建基于16S rRNA基因的系统发育树。
菌株SZY C1基因组测序利用北京百迈客生物科技有限公司的Illumina novaseq 6000平台上进行,SPAdes软件[13]组装,将组装好的基因组框架图序列上传到NCBI数据库。分别使用JSpeciesWS(http://jspecies.ribohost.com/jspeciesws/)、GGDC(https://ggdc.dsmz.de/distcalc2.php)和ANI Calculator(http://enve-omics.ce.gatech.edu/aai/)计算分离株SZY C1与相关模式菌株的平均核苷酸一致性(average nucleotide identity,ANI)、数字DNA-DNA杂交值(digital DNA-DNA hybridization,dDDH)和平均氨基酸一致性(average amino acid identity,AAI)。使用GTDB-Tk(https://github.com/Ecogenomics/GtdbTk)构建全基因组系统发育树。
采用法国梅里埃公司VITEK 2 Compact全自动微生物鉴定与药敏分析仪及配套GN335卡对菌株SZY C1进行药敏试验,具体操作参见说明书。参考CLSI M100-S33中非肠杆菌科细菌的药敏标准进行药敏结果判定。
菌株SZY C1在哥伦比亚血琼脂平板、巧克力平板、麦康凯平板、LB琼脂平板、MH琼脂平板和R2A平板上均能生长。其中,在哥伦比亚血琼脂平板上培养24 h呈现灰白色、圆形、突起、不透明、边缘整齐的菌落(图1)。革兰染色阴性,菌体单个或成对排列,无芽孢,电镜显示菌株SZY C1在磷酸盐缓冲液中形态为球杆状(图2)。菌株SZY C1在14~40℃均可生长,最适合生长温度为35℃,最适pH为7.0,最适生长盐浓度为1%,可在厌氧环境下生长。使用VITEK 2 Compac系统GN卡进行鉴定,结果显示菌株SZY C1为少见贪铜菌,其鉴定概率为95%,生物编码为4042001103500243。菌株SZY C1与近缘模式菌株生理生化特性差异详细见表1。
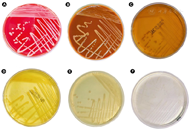

注:A:哥伦比亚血琼脂培养基;B:普通巧克力培养基;C:麦康凯培养基;D:MH培养基;E:LB琼脂培养基;F:R2A琼脂培养基
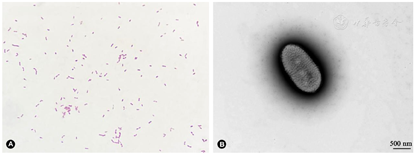

注:A:菌株SZY C1革兰染色镜下形态(×1 000);B:菌株SZY C1的透射电镜图
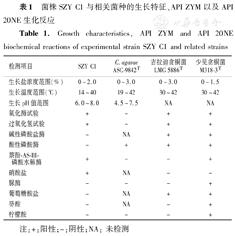
菌株SZY C1与相关菌种的生长特征、API ZYM以及API 20NE生化反应
Growth characteristics, API ZYM and API 20NE biochemical reactions of experimental strain SZY C1 and related strains
菌株SZY C1与相关菌种的生长特征、API ZYM以及API 20NE生化反应
Growth characteristics, API ZYM and API 20NE biochemical reactions of experimental strain SZY C1 and related strains
| 检测项目 | SZY C1 | C.agavae ASC-9842T | 吉拉迪贪铜菌LMG 5886T | 少见贪铜菌M318-3T |
|---|---|---|---|---|
| 生长盐浓度范围(%) | 0~2.0 | 0~3.0 | 0~3.0 | 0~1.5 |
| 生长温度范围(℃) | 14~40 | 19~42 | 30~42 | 30~42 |
| 生长pH值范围 | 6.0~8.0 | 4.5~7.5 | NA | NA |
| 氧化酶试验 | + | - | + | + |
| 过氧化氢试验 | + | - | + | + |
| 碱性磷酸盐酶 | - | NA | + | + |
| 酸性磷酸酶 | - | + | + | + |
| 萘酚-AS-BI-磷酸水解酶 | + | - | - | + |
| 硝酸盐 | + | NA | - | - |
| 脲酶 | - | - | - | + |
| 葡萄糖酸盐 | - | NA | + | + |
| 癸酸 | - | NA | - | + |
| 柠檬酸 | - | - | - | + |
注:+:阳性;-:阴性;NA:未检测
菌株SZY C1使用VITEK MS质谱检测系统鉴定为少见贪铜菌,置信度为99.9%。EXS3000质谱仪鉴定为少见贪铜菌,鉴定分值为2.09,为种水平鉴定。与CDC microbenet在线数据库进行比对鉴定为耐金属贪铜菌,鉴定分值为1.852,为属水平鉴定。
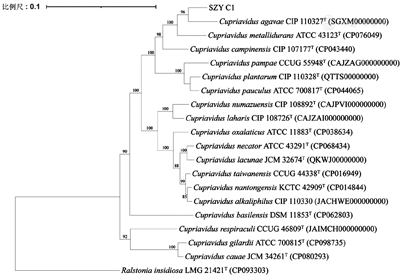
将菌株SZY C1的16S rRNA基因序列提交到GenBank/ EMBL/DDBJ,获得序列号为OR145515。与EzBioCloud数据库中序列相似度比对结果显示,菌株SZY C1与耐金属贪铜菌(Cupriavidus metallidurans LMG 1195T)、少见贪铜菌(Cupriavidus pauculus LMG 5886T)的16S rRNA基因序列相似性最高,分别为98.52%和98.38%,低于16S rRNA基因物种鉴定阈值98.65%[17,18]。系统进化树显示,菌株SZY C1与这2株菌的进化距离最近,形成一个独立的进化分支(图3),提示从16S rRNA基因分类水平上支持菌株SZY C1属于贪铜菌属中的一个新种。
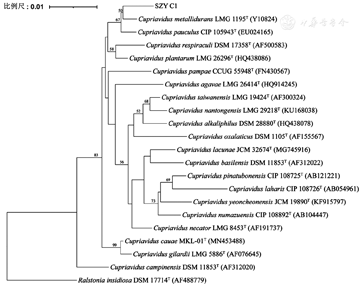

菌株SZY C1测得基因组大小为5 515 517 bp,G+C含量为67.87%,编码基因4 869个,携带2个rRNAs、58个tRNAs、1个基因组岛,序列登录号为JAUANU000000000。从表2可见,菌株SZY C1与根际贪铜菌(Cupriavidus agavae CIP 110327T)的ANI值、AAI值和数字DNA-DNA杂交值为最高,分别为84.76%、86.07%和29.10%,低于目前被广泛接受的用于界定原核物种的阈值(95%ANI,95%AAI和70%数字DNA-DNA杂交值)[17,18]。基于120个串联单拷贝蛋白质构建系统发育树显示分离株SZY C1与根际贪铜菌(Cupriavidus agavae CIP 110327T)亲缘关系最近,所在分支上的置信度值均较高,表明进化关系是可靠的(图4)。综合系统发育分析和基因组亲缘关系指数表明,分离株SZY C1属于贪铜菌属中潜在的一个新种。

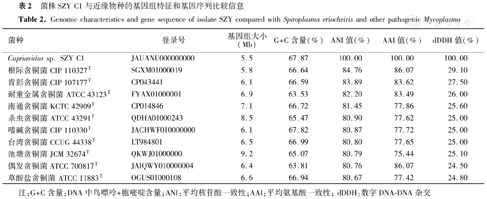
菌株SZY C1与近缘物种的基因组特征和基因序列比较信息
Genomic characteristics and gene sequence of isolate SZY compared with Spiroplasma eriocheiris and other pathogenic Mycoplasma
菌株SZY C1与近缘物种的基因组特征和基因序列比较信息
Genomic characteristics and gene sequence of isolate SZY compared with Spiroplasma eriocheiris and other pathogenic Mycoplasma
| 菌种 | 登录号 | 基因组大小(Mb) | G+C含量(%) | ANI值(%) | AAI值(%) | dDDH值(%) |
|---|---|---|---|---|---|---|
| Cupriavidus sp. SZY C1 | JAUANU000000000 | 5.5 | 67.87 | 100.00 | 100.00 | 100.00 |
| 根际贪铜菌CIP 110327T | SGXM01000019 | 5.8 | 66.64 | 84.76 | 86.07 | 29.10 |
| 肯彭贪铜菌CIP 107177T | CP043441 | 6.1 | 66.59 | 83.89 | 83.62 | 27.50 |
| 耐重金属贪铜菌ATCC 43123T | FYAX01000001 | 6.9 | 63.53 | 82.20 | 83.49 | 26.00 |
| 南通贪铜菌KCTC 42909T | CP014846 | 7.1 | 66.72 | 81.45 | 77.86 | 25.60 |
| 杀虫贪铜菌ATCC 43291T | QDHA01000243 | 8.5 | 65.47 | 80.90 | 77.62 | 25.00 |
| 嗜碱贪铜菌CIP 110330T | JACHWF010000000 | 6.1 | 67.82 | 80.87 | 77.72 | 25.00 |
| 台湾贪铜菌CCUG 44338T | LT984801 | 6.5 | 66.99 | 80.80 | 77.65 | 25.00 |
| 池塘贪铜菌JCM 32674T | QKWJ01000000 | 9.2 | 65.07 | 80.79 | 75.44 | 25.10 |
| 偶发贪铜菌ATCC 700817T | JAIQWY010000004 | 6.4 | 63.81 | 80.76 | 86.07 | 24.50 |
| 草酸盐贪铜菌ATCC 11883T | OGUS01000108 | 6.6 | 66.94 | 80.67 | 77.42 | 24.80 |
注:G+C含量:DNA中鸟嘌呤+胞嘧啶含量;ANI:平均核苷酸一致性;AAI:平均氨基酸一致性;dDDH:数字DNA-DNA杂交
如表3所示,分离株SZY C1对替卡西林/克拉维酸、哌拉西林/他唑巴坦、头孢他啶、头孢吡肟、多西环素、左氧氟沙星、妥布霉素、亚胺培南、美罗培南、阿米卡星、环丙沙星、米诺环素和头孢曲松敏感,但对氨曲南耐药。
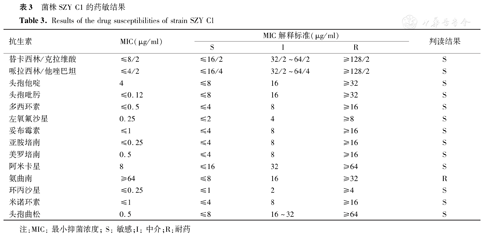
菌株SZY C1的药敏结果
Results of the drug susceptibilities of strain SZY C1
菌株SZY C1的药敏结果
Results of the drug susceptibilities of strain SZY C1
| 抗生素 | MIC(μg/ml) | MIC解释标准(μg/ml) | 判读结果 | ||
|---|---|---|---|---|---|
| S | I | R | |||
| 替卡西林/克拉维酸 | ≤8/2 | ≤16/2 | 32/2~64/2 | ≥128/2 | S |
| 哌拉西林/他唑巴坦 | ≤4/2 | ≤16/4 | 32/2~64/4 | ≥128/2 | S |
| 头孢他啶 | 4 | ≤8 | 16 | ≥32 | S |
| 头孢吡肟 | ≤0.12 | ≤8 | 16 | ≥32 | S |
| 多西环素 | ≤0.5 | ≤4 | 8 | ≥16 | S |
| 左氧氟沙星 | 0.25 | ≤2 | 4 | ≥8 | S |
| 妥布霉素 | ≤1 | ≤4 | 8 | ≥16 | S |
| 亚胺培南 | ≤0.25 | ≤4 | 8 | ≥16 | S |
| 美罗培南 | 0.5 | ≤4 | 8 | ≥16 | S |
| 阿米卡星 | 8 | ≤16 | 32 | ≥64 | S |
| 氨曲南 | ≥64 | ≤8 | 16 | ≥32 | R |
| 环丙沙星 | ≤0.25 | ≤1 | 2 | ≥4 | S |
| 米诺环素 | ≤1 | ≤4 | 8 | ≥16 | S |
| 头孢曲松 | 0.5 | ≤8 | 16~32 | ≥64 | S |
注:MIC:最小抑菌浓度;S:敏感;I:中介;R:耐药
与VFDB数据库、CARD数据库和重金属抗性基因数据库比对分析,对菌株SZY C1的基因组毒力基因、耐药基因、重金属抗性基因进行注释。结果显示,SZY C1菌株共注释得到25种毒力基因,主要与运动、黏附、效应器输送,营养代谢和免疫适应等相关,其中与运动相关的编码鞭毛蛋白基因占比最多。菌株SZY C1共检出10个耐药基因,主要与耐多药外排泵功能相关。此外,菌株SZY C1基因组还检出多个重金属抗性基因,结果详见表4与表5。

菌株SZY C1毒力基因比对结果
Comparison results of virulence gene of strain SZY C1
菌株SZY C1毒力基因比对结果
Comparison results of virulence gene of strain SZY C1
| 毒力功能 | 毒力因子 | 毒力基因 |
|---|---|---|
| 运动 | 鞭毛 | cheB、cheR、cheW、cheY、cheZ、flgC、flgG、flgH、flgI、flhA、fliA、fliG、fliI、fliM、fliN、fliP、fliQ、motA |
| 黏附 | Ⅳ型菌毛 | pilG、pilT、pilU |
| 效应器输送系统 | xcp分泌系统 | xcpT |
| 营养代谢 | 铜绿假单胞菌铁载体 | pvdA |
| 免疫适应 | Brk | brkB |
| 其他 | 异柠檬酸裂合酶 | icl |
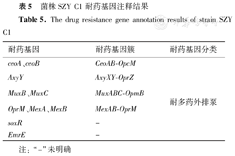
菌株SZY C1耐药基因注释结果
The drug resistance gene annotation results of strain SZY C1
菌株SZY C1耐药基因注释结果
The drug resistance gene annotation results of strain SZY C1
| 耐药基因 | 耐药基因簇 | 耐药基因分类 |
|---|---|---|
| ceoA、ceoB | CeoAB-OpcM | 耐多药外排泵 |
| AxyY | AxyXY-OprZ | |
| MuxB、MuxC | MuxABC-OpmB | |
| OprM、MexA、MexB | MexAB-OprM | |
| soxR | - | |
| EmrE | - |
注:"-"未明确
贪铜菌属中的菌种主要从世界各地许多不同类型的环境中分离出来,如土壤、根瘤、水、废水、污染沉积物、火山泥流、金属工厂等,这些物种普遍携带大量编码抗重金属基因[19,20]。尽管贪铜菌属在人类感染中的致病性尚不清楚,但其致病性的证据正在增加。吉拉迪贪铜菌从免疫力低下患者的血液、咽喉、脓肿或粪便标本中分离出,并引起菌血症和败血症的全身性感染,也可导致再生障碍性贫血儿童感染性死亡[7,8,21]。而与吉拉迪贪铜菌亲缘关系密切的央大贪铜菌也可引起败血症[6]。少见贪铜菌是一种机会性致病菌,尤其是感染人类免疫缺陷病毒、血液系统恶性肿瘤以及进行器官移植的患者,在感染少见贪铜菌后引起人菌血症、腹膜炎、腱鞘炎等疾病,且有院内传播的证据[22,23,24,25]。本研究从1例右足开放性距骨骨折的患者伤口拭子中分离1株ZSY C1,基于表型实验和全基因组测序分析,确定为贪铜菌属一个未分类的新菌种。
菌株SZY C1与耐金属贪铜菌(Cupriavidus metallidurans LMG 1195T)间的16S rRNA基因序列相似度为98.52%,低于16S rRNA基因序列物种鉴定阈值98.65%[17,18],提示菌株SZY C1很可能是贪铜菌属内的一个细菌新种。值得注意的是,与16S rRNA基因相比,基于全基因组的系统发育学分类能够提供更高的分辨率,尤其适用于亲缘关系相近的物种[26]。全基因分析显示菌株SZY C1与根际贪铜菌(Cupriavidus agavae CIP 110327T)亲缘关系更近,两者间的ANI值为84.76%,数字DNA-DNA杂交值为29.10%,同时低于ANI细菌物种边界阈值95%~96%和数字DNA-DNA杂交物种边界阈值70%[27],综合基因组亲缘关系指数和系统发育分析表明,菌株SZY C1属于贪铜菌属中未分类的一个潜在新种。
全自动表型鉴定系统和MALDI-TOF MS主要是利用微生物的生化反应和微生物肽质量指纹特征与事先建立的数据库比对实现微生物菌落的快速鉴定。然而,即使鉴定得分足以确定微生物的物种水平,仍常存在物种错误鉴定的情况。在本研究中,菌株SZY C1使用VITEK 2和VITEK MS鉴定为少见贪铜菌,置信度值为99.9%。同属的央大贪铜菌同样存在这样问题,使用VITEK MS鉴定为吉拉迪贪铜菌,置信度值为99.9%[28]。这可能由于不同菌株之间的生化反应和核糖体蛋白质特征峰的重叠,或者微生物数据库的数量有限所引起。例如在VITEK 2数据库只含有少见贪铜菌,而贪铜菌属在VITEK MS和EXS3000质谱数据库中都只含吉拉尔迪贪铜菌、杀虫贪铜菌、少见贪铜菌和呼吸道贪铜菌等4个物种。因此,应尽快完善临床贪铜菌属数据库建设及更新鉴定的技术,以便更准确地鉴定贪铜菌及其相关物种。
为了鉴定菌株SZY C1的毒力基因和抗生素耐药性基因,本研究选择了VFDB数据库和CARD数据库对基因组注释。菌株SZY C1注释到的毒力基因主要与运动、黏附、效应器输送和营养代谢免疫适应毒力因子相关。其中与黏附相关的Ⅳ型菌毛基因在菌株SZY C1都有检出,Ⅳ型菌毛可以促进细菌和外部环境之间的电子转移,已被证明在细菌黏附与摆动发挥重要作用[29]。菌株SZY C1基因组注释结果显示存在多种抗生素耐药基因,这些基因包括多种耐多药外排泵的基因,如CeoAB-OpcM、AxyXY-OprZ、MuxABC-OpmB、MexAB-OprM等。这些外排泵基因与铜绿假单胞菌的外排泵基因高度相似,外排泵基因簇MexAB-OprM可以降低铜绿假单胞菌对美罗培南、其他β-内酰胺类和氨基糖苷类药物敏感性[30]。而外排泵基因MuxABC-OpmB也可引起氨苄西林和羧苄西林的耐药性增加[31]。研究表明耐药基因出现点突变后可能产生耐药性[19],由于贪铜菌携带多种耐药基因,因此需要密切关注贪铜菌出现耐药基因突变的可能性。有研究评估贪铜菌属的临床分离株的耐药性,结果显示米诺环素和头孢吡肟对贪铜菌表现良好的抗菌活性,但对阿莫西林、阿莫西林/克拉维酸、替莫西林和氨曲南普遍耐药[32]。本次菌株SZY C1的药敏试验显示除了对氨曲南耐药外,对大部分的抗生素均为敏感。表明了贪铜菌属对抗生素的敏感性存在一定的种间差异。美罗培南、氨基糖苷类和多黏菌素作为革兰阴性杆菌感染治疗最主要的抗菌药物之一,对贪铜菌属部分菌株缺乏可靠的敏感性[32]。因此,贪铜菌感染的治疗应当结合抗菌药敏试验,避免经验用药。
菌株SZY C1基因组还检出多个抗重金属基因。这些抗重金属基因有助于抵抗铜、银、镁、钴、汞、钼、砷、锌、镉、铁、锑和镍等多种重金属。贪铜菌主要通过在细胞内螯合金属来增强对金属抗性,例如CopZ基因与铜、银和镉离子的排出有关[33,34]。贪铜菌属中的多个菌种具备抗重金属的能力,包括少见贪铜菌、耐重金属贪铜菌、吉拉迪贪铜菌、肯彭贪铜菌等[35,36,37,38]。值得注意的是,金属抗性和抗生素耐药性之间还存在关联,抗生素耐药基因与抗重金属基因可能共享可移动遗传元件,如整合性接合元件ICEHs1[39]。
综上所述,本研究通过对菌株SZY C1进行表型特征试验、16S rRNA基因、全基因组测序和系统发育分析,证实了SZY C1是一种贪铜菌属中的潜在新种。由于贪铜菌普遍携带大量耐药基因和抗重金属基因,因此我们需要密切关注与人类疾病感染有关的贪铜菌,以充分评估其对公众健康可能构成的潜在风险。
康世越,邓晨晖,陈晓慰,等. 1株伤口分离贪铜菌的分类鉴定及生物学特征分析[J].中华微生物学和免疫学杂志,2024,44(6): 510-518. DOI: 10.3760/cma.j.cn112309-20231031-00115.
所有作者声明无利益冲突



























