
建立一种基于临床标本核酸的肺炎支原体(Mycoplasma pneumoniae, Mp)核酸检测及基因分型方法。
通过基因组学比对,选取Mp Genotype 1型和Genotype 2型菌株特异性靶序列设计合成引物和探针,建立Mp双荧光探针PCR检测分型方法,同时评价该方法的特异度、准确度、检测限和重复性。采用建立的荧光PCR方法对临床标本核酸进行检测,并与已报道的荧光PCR方法进行比对。
对与Mp种属关系相近的其他种支原体以及常见呼吸道细菌、病毒等18种病原体的核酸进行检测,结果显示均无交叉反应;对90份Mp核酸的检测及分型的准确度为100%。该方法对于Genotype 1型和Genotype 2型Mp检测限均为1.0拷贝/μl的核酸样本,组内、组间重复性实验变异系数均小于2.5%。对88份临床标本核酸的检测中,与已报道的荧光PCR方法RepMp1体系的检测结果相比,Kappa值为0.675,P值为0.267,显示出高度的一致性。
本研究所建立的Mp核酸检测及基因分型方法敏感度和特异度高,分型准确,可应用于各级省市疾控系统对Mp的监测与防控,促进Mp防控体系的完善,增强监测能力,对Mp的预警预测具有重要意义。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
肺炎支原体(Mycoplasma pneumoniae, Mp)是引起人类呼吸道感染的常见病原菌之一,尤其是儿童[1,2]。国外研究表明,每年10%~30%的社区获得性肺炎(community-acquired pneumonia, CAP)由Mp感染引发[3,4,5]。我国一项历时11年,覆盖23万人群的多中心研究表明,Mp引起的呼吸道感染在我国学龄儿童中占比接近60%,高居第一位[6]。Mp的人群携带率也是临床非常关注的数据,北京某区2023年1 303例健康体检儿童的结果显示,在健康儿童人群中,Mp检出率约为4.1%[7]。Mp基因组小且保守,既往的组学研究表明,Mp在基因组水平主要可分为Genotype 1型菌株和Genotype 2型菌株两种基因型别,其差异主要位于由多种RepMp重复序列构成的重要黏附蛋白基因序列,例如p1、p40、p90等[8,9,10]。Kenri等[11]通过对日本1976—2005年临床Mp菌株分型研究,发现每10年左右Mp发生一次基因型转化,提出基因型的变化引发的抗原变迁是Mp流行暴发的原因之一[12,13]。此外,有研究表明,Genotype 2型Mp菌株在组学水平上毒力因子表达更多,其感染后临床症状较感染Genotype 1型Mp菌株更加严重[14,15]。因此,对Mp基因型检测与监测,不仅有助于个体化临床治疗,根据监测结果调整治疗方案,使得患儿的治疗更具针对性[16],而且有助于了解区域Mp抗原变迁情况,对Mp暴发预警有协助作用[17]。
目前,常用的Mp基因分型技术主要有p1基因限制性片段长度多态性分析(restriction fragment length polymorphism, RFLP)[13,18,19,20,21]、多位点可变数目串联重复序列分型(multiple-locus variable-number tandem repeat analysis, MLVA)[22]、质谱[23]和多位点序列分析(multilocus sequence typing, MLST)[24]等。这些方法主要依赖于普通PCR扩增,敏感度相对较低,一般需要对Mp进行培养后,提取高浓度的核酸才能得出有效的结果,因此检测周期长、操作复杂,不适用于临床快速检测以及疾控监测的需求。鉴于上述Mp检测及分型弊端,本研究建立一种不依赖分离培养,直接从临床标本中对Mp检测及分型的荧光PCR技术,实现标本中Mp检测与分型同步。
以下菌株核酸均来自中国疾病预防控制中心传染病预防控制所:Mp Genotype 1型菌株(ATCC29342)、Mp Genotype 2型菌株(ATCC15531)、解脲脲原体(ATCC27618)、生殖支原体(ATCC33530)、猪鼻支原体(ATCC17981)、梨支原体(ATCC25960)、人型支原体(ATCC23114)、穿透支原体(ATCC55252)、口腔支原体(ATCC23714)、发酵支原体(ATCC19989)、沙眼衣原体(临床分离株)、脑膜炎奈瑟菌B群(临床分离株)、脑膜炎奈瑟菌C群(临床分离株)、肺炎克雷伯菌(临床分离株)、金黄葡萄球菌(临床分离株)、结核分枝杆菌(临床分离株)、流感杆菌(临床分离株)、空肠弯曲菌(临床分离株)、鲍曼不动杆菌(临床分离株)、脓肿分枝杆菌(临床分离株)。
2×Taqman PCR MasterMix购自北京索莱宝公司;QuantStudio™ 6 Flex实时荧光定量PCR系统购自美国Thermo Fisher公司;QIAamp核酸小量提取试剂盒购自德国QIAGEN Gmbh公司;Qubit仪、高速离心机购自美国Thermo Fisher公司;微量移液器购自德国Eppendorf公司;超低温冰箱购自中国美菱低温科技股份有限公司;生物安全柜购自青岛海尔深谷医疗股份有限公司。
参照GenBank上登录的Mp基因组序列:使用Mavue(The Darling lab at the University of Technology Sydney)软件全基因组序列进行比对,筛选出Genotype 1型菌株ATCC29342 (NCBI编号:NC_000912.1)mpn459编码基因区域和Genotype 2型菌株309(NCBI编号:AP012303.1)的MPNA5864~MPNA5865区域,使用Primer Express 3.0.1软件(Thermo Fisher,美国)设计Genotype 1型菌株和Genotype 2型菌株的扩增引物和MGB(minor groove binder)探针。Type 1F: 5′-TGTCGCCTTTTCCAGATTCAC-3′,Type 1R: 5′-AACTGGAAGTACTGAGGATGAATGA-3′,Type 1P: 5′-FAM-AATTTCTGAACCAACAACTT-MGB-3′;Type 2F: 5′-CCCAAACGAAAGGAAATTGTTC-3′,Type 2R: 5′-GAAAGACTATTCGCCTTACAACCAA-3′,Type 2P: 5′-VIC-ACCTAAAGCAGGATCGG-MGB-3′。引物和探针购自生工生物工程(上海)股份有限公司。
使用购买的2×Taqman PCR MasterMix试剂,将实时荧光PCR的引物和探针的浓度在5.0~25.0 μmol/L之间进行不同浓度的配比,筛选出最佳的引物和探针浓度。在优化好引物和探针的条件下,将实时荧光PCR的退火温度设为54~61℃,退火时间设为10~20 s,Mg2+浓度设为4.0~6.0 mmol/L,根据扩增搜集荧光量的多少来判断最佳退火温度、时间和体系的镁离子浓度。
选择与Mp种属关系较近的8种其他支原体以及10种其他常见呼吸道病原体核酸作为特异度试验模板,以Genotype 1型菌株ATCC29342和Genotype 2型菌株ATCC15531核酸作为阳性对照,无核酸水作为无模板阴性对照,验证方法的特异度。
选取实验室保存的90株Mp菌株核酸进行鉴定和基因分型试验,其中Genotype 1型Mp菌株80株,Genotype 2型Mp菌株10株,其依据RepMp1[25]体系鉴定为Mp,依据Mptype体系[26]鉴定为Genotype 1型和Genotype 2型菌株。
将ATCC29342和ATCC15531纯菌提取核酸,使用Qubit仪器测定核酸浓度,用无核酸水将两种基因型Mp核酸分别定量为106拷贝/μl。然后进行10倍梯度稀释,累计稀释7个梯度。分别以106~1.0拷贝/μl为模板,建立分型检测方法标准曲线,确定最小检出量的拷贝数。
选取菌株浓度为102拷贝/μl的Mp核酸作为模板进行3次平行试验,每次上样5.0 μl,计算Ct值变异系数。在第1、3、5天时上述模板以相同反应条件进行组间重复试验。
随机选取实验室保存的88份临床咽拭子标本核酸,分别使用靶基因为RepMp1重复序列的real-time PCR检测方法[25]和本检测与分型方法进行Mp核酸检测,并比对结果。
优化后的检测体系为:Solarbio 2×mix 12.5 μl (北京索莱宝),Type 1F 0.5 μl(25.0 μmol/L),Type 1R 0.5 μl(25.0 μmol/L),Type 1P 0.5 μl(25.0 μmol/L),Type 2F 0.5 μl(25.0 μmol/L),Type 2R 0.5 μl(25.0 μmol/L),Type 2P 0.5 μl(25.0 μmol/L),模板5.0 μl,添加Mg2+浓度为4.0 mmol/L,无核酸水补充至总体积25.0 μl。反应程序为:90℃ 10 min,然后95℃ 15 s,59℃ 15 s,45个循环,可获取最低的扩增Ct值以及较高的荧光强度(图1)。
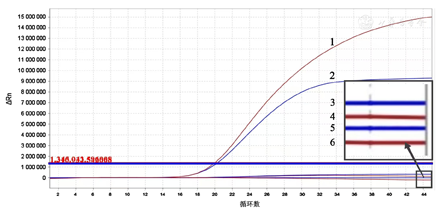

注:1:ATCC29342(FAM);2:ATCC15531(VIC);3:ATCC29342(VIC);4:ATCC15531(FAM);5:无模板阴性对照(VIC);6:无模板阴性对照(FAM)
特异度结果显示(图2),本研究建立的方法能特异性地扩增出Mp,解脲支原体、生殖支原体、猪鼻支原体、梨支原体、人型支原体、穿透支原体、口腔支原体、发酵支原体核酸这些与Mp种属关系较近的支原体均未有特异性扩增;沙眼衣原体、脑膜炎奈瑟菌B群、脑膜炎奈瑟菌C群、肺炎克雷伯菌、金黄葡萄球菌、结核分枝杆菌、流感杆菌、空肠弯曲菌、鲍曼不动杆菌、脓肿分枝杆菌和空白对照组也均未有特异性扩增。
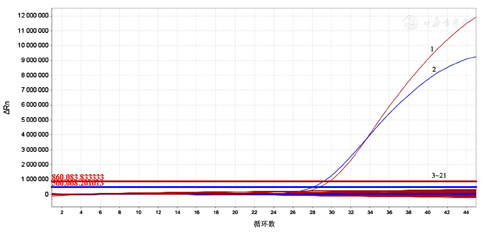

注:1:ATCC29342;2:ATCC15531;3~21:分别为解脲支原体、生殖支原体、猪鼻支原体、梨支原体、人型支原体、穿透支原体、口腔支原体、发酵支原体、沙眼衣原体、脑膜炎奈瑟菌B群、脑膜炎奈瑟菌C群、肺炎克雷伯菌、金黄葡萄球菌、结核分枝杆菌、流感杆菌、空肠弯曲菌、鲍曼不动杆菌、脓肿分枝杆菌和空白对照
对90株实验室保存的Mp菌株核酸进行鉴定,结果均为阳性,其中Genotype 1型Mp菌株80株,分型准确率为100%,Genotype 2型Mp菌株10株,分型准确率为100%。
以106~1.0拷贝/μl浓度的核酸作为模板,从高到低共7个滴度,外加无模板阴性对照,每个滴度做复孔,按照上述体系进行扩增。结果显示(图3),本研究方法的检测敏感度为1.0拷贝/μl。该方法出现的荧光信号强度与核酸浓度呈线性关系(图4),对Genotype 1型菌株的标准曲线回归方程为:y=-3.283x+38.775, R2=0.997;对Genotype 2型菌株的标准曲线回归方程为:y=-3.333x+35.545,R2=0.997。提示本方法能够用于检测Mp,且Ct值与标准品的浓度具有良好的相关性。
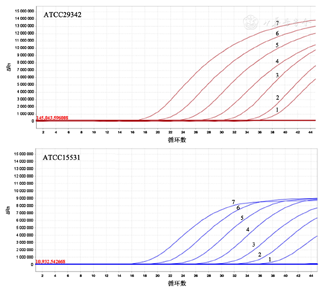

注:1~7:分别为1.0、101、102、103、104、105、106拷贝/μl
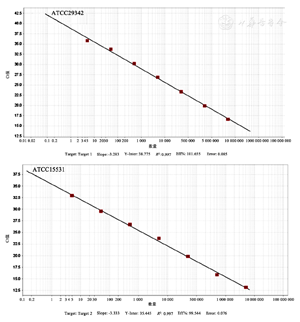

选取浓度为102拷贝/μl的标本核酸作为模板进行3次平行试验,3个平行反应均获得扩增曲线。通过计算得出(表1),Ct值变异系数分别为0.31%和1.29%。在第1、3、5天对上述模板进行组间重复试验(表2),Ct值变异系数分别为2.31%和0.57%。组内、组间重复性试验变异系数均小于2.5%,表明本方法的重复性良好。
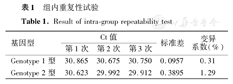
组内重复性试验
Result of intra-group repeatability test
组内重复性试验
Result of intra-group repeatability test
| 基因型 | Ct值 | 标准差 | 变异系数(%) | ||
|---|---|---|---|---|---|
| 第1次 | 第2次 | 第3次 | |||
| Genotype 1型 | 30.865 | 30.675 | 30.750 | 0.0957 | 0.31 |
| Genotype 2型 | 30.623 | 29.992 | 29.912 | 0.3895 | 1.29 |
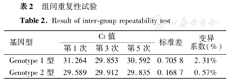
组间重复性试验
Result of inter-group repeatability test
组间重复性试验
Result of inter-group repeatability test
| 基因型 | Ct值 | 标准差 | 变异系数(%) | ||
|---|---|---|---|---|---|
| 第1次 | 第3次 | 第5次 | |||
| Genotype 1型 | 31.264 | 29.853 | 30.592 | 0.705 8 | 2.31% |
| Genotype 2型 | 29.589 | 29.912 | 29.835 | 0.168 7 | 0.57% |
采用文献报道的RepMp1体系对88份临床标本核酸进行检测,Mp阳性率为37.5%(33/88),同时采用本研究方法检出Mp Genotype 1型菌株24株,Mp Genotype 2型菌株4株,阳性率为31.8%(28/88),经配对卡方检验,Kappa值为0.675,P值为0.267,两种检测方法的差异无统计学意义。
2023年我国多省份相继暴发Mp疫情,使得其无论在个体诊治还是群体防控中均受到前所未有的重视,国家加强了对Mp的检测与监测。中国疾病预防控制中心发布开展急性呼吸道传染病多病原监测试点工作的通知,将Mp列为呼吸道症候群必检病原。对于疾控监测来说,单一的核酸检测并不能满足监测需求,需要进一步强化Mp基因型监测,了解Mp人群的抗原变迁情况。
目前,Mp分型技术主要用于科研,通常依赖于获取菌株后培养所得的纯菌核酸进行分型研究[20,22,23],检测敏感度不足,限制了其在疾病监测中的应用。Sun等[27]建立了临床标本的p1基因与MLVA联合扩增的分型技术,可用于临床标本核酸的直接检测与分型,提升了标本的分型敏感度,但其扩增靶点过多,实际应用过程过于复杂。课题组针对临床标本的Mp分型难点,建立了基于TaqMan探针的高敏感度和特异度双荧光PCR检测分型体系[26],并广泛应用[28,29,30,31]。该方法使用的TaqMan探针信噪比较差,在检测低浓度Genotype 2型菌株时,其荧光信号受信噪比影响,导致其强度较低,给结果判定会带来一定影响。因此本研究在该方法基础上,重新设计引物探针,力求将序列间干扰降低,同时使用信噪比更优秀的MGB技术来修饰探针,有效实现了低浓度Genotype 2型菌株扩增荧光信号强度问题。
本方法不但可用于临床标本的直接检测,而且能做到Mp检测和分型同步进行。在分型方面,对90株Mp分型的准确率为100%,具有准确的分型能力。标准曲线扩增结果表明,Genotype 1和2型核酸的最低检出限均为1.0拷贝/μl,因此该方法的Mp检测与分型的最低检测限为1.0拷贝/μl。课题组使用既往的靶基因为RepMp1重复序列的Mp real-time PCR检测方法与本研究方法进行临床标本Mp核酸检测,两者的Mp检测率分别为37.5%和31.8%,经配对卡方检验,两种检测方法的差异无统计学意义。表明本方法在具备Mp基因分型的同时,还兼具高敏感度的Mp核酸检测能力,可满足临床标本检测以及病原的防控监测需求。
综上所述,本研究建立了一种快捷的可用于标本中Mp检测以及分型的荧光PCR方法,该方法可广泛应用于各级省市疾控系统对Mp的监测与防控,可促进Mp防控体系的完善,增强监测能力,对支原体肺炎的预警预测具有重要意义。
张倚玮,刘立雍,何利华,等.基于标本核酸的肺炎支原体基因分型荧光PCR方法的建立和应用[J].中华微生物学和免疫学杂志,2024,44(8):653-659. DOI: 10.3760/cma.j.cn112309-20240627-00229.
所有作者声明无利益冲突



























