
重组酶聚合酶扩增技术作为一种新型的核酸恒温扩增检测技术,近年来已经展现出其巨大的应用潜力和发展前景。高敏感度、高特异度、对仪器依赖程度低以及可整合多种检测模式等独特的优势,使其在多个领域,特别是基层和现场即时检测中具有广泛的应用价值。本文对重组酶聚合酶扩增技术的发展历程、检测原理、研究动态及应用进展进行综述,并对其未来发展进行展望,以期为基于重组酶聚合酶扩增技术的病原体快速检测方法的研发和临床应用提供有益参考。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
聚合酶链反应(polymerase chain reaction,PCR)自1985年诞生以来,犹如为分子生物学领域的发展注入了一针强心剂,促进了分子生物学领域的飞速发展[1]。然而,随着研究的深入和应用的普及,PCR技术的局限性开始显现,如其高度依赖检测设备、耗时、操作复杂、人员要求高等,限制了PCR技术在特定环境或场景中的应用,促使研究人员探索和发展新技术新方法来弥补现有缺陷[2],Piepenburg等[3]在2006年发明的重组酶聚合酶扩增技术(recombinase polymerase amplification,RPA)就是其中之一,突破了传统PCR技术的限制,极大拓宽了核酸检测的应用范围。
RPA技术作为一项新型等温扩增技术,能够在37~42℃的温度下即可实现对待测样本的快速扩增[3],与传统PCR技术相比,极大地简化了实验流程,降低了对精密仪器的依赖。可实现现场实时检测,可与多种临床先进技术结合是RPA技术独特的优势[4],因此,该技术受到研究人员和临床的广泛关注。本文将从RPA技术的发展历程、检测原理、研究动态及应用最新进展进行阐述,并对RPA未来发展趋势进行展望(图1)。

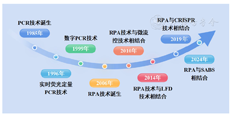
1985年Mullis等[1]发明的PCR技术是生命科学领域里程碑式的突破,经过30多年的发展,其已广泛应用于各个领域。基于科学研究和临床诊断领域对核酸检测方法定量检测的需求,实时荧光定量PCR(real-time fluorescence quantitative PCR,qPCR)技术由Heid等[5]在1996年提出,自此实现了对核酸的定量检测分析,极大推动了PCR技术的研究和广泛应用。20世纪末期,研究人员对检测方法敏感度等性能指标提出了更高的需求,作为第3代PCR技术的数字PCR(digital PCR,dPCR)在1999年被Vogelstein和Kinzler[6]报道,该技术可实现检测靶标的绝对定量,无需标准曲线,为遗传分析和分子诊断领域带来了巨大进步。
步入21世纪,即时检测(point-of-care testing,POCT)的需求与日俱增,PCR及其衍生技术的局限性促使了等温扩增技术的诞生[7]。RPA技术随之横空出世,该技术可实现非实验室条件下核酸的快速恒温扩增检测,故其迅速引起各领域的广泛关注。随后,RPA技术的研究不断取得创新性突破,Lutz等[8]于2010年提出RPA与微流控技术相结合对金黄葡萄球菌抗生素耐药基因mecA进行检测,其简单的处理步骤、高敏感度及高通量的特点展示了RPA技术在床旁检测应用中的独特潜力。2014年,Jaroenram和Owens[9]提出RPA与侧流层析试纸技术(lateral flow dipstick technology,LFD)结合,形成了RPA-LFD检测新思路,解决了因核酸染料对扩增过程抑制作用而降低检测系统敏感度的问题。该新方法能直接实现肉眼判定检测结果,为即采即检的检测需求提供了一种更便捷、更快速、更灵活的解决方案。Khan等[10]于2019年针对当时RPA的非特异性扩增导致假阳性的问题,提出了将RPA技术与短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)相结合,可对目标基因进行双重特异性识别,提高了检测特异度和敏感度,是RPA技术发展史上的又一大突破。2024年,Feng等[11]提出了一种基于RPA的新型检测策略,该方法利用链霉素亲和素-生物素系统(streptomycin avidin-biotin system,SABS)结合RPA-LFD,可提高检测敏感度,为靶标的定量检测提供了一种新的手段[12]。随后,研究人员持续探索了将RPA技术与多种前沿技术相结合的可能性,成功克服了传统检测方法所面临的众多挑战和限制,进而扩展了适用于检测的样本类型,并且增强了检测方法开发过程中的适应性和创新性(图2)。

RPA技术通过模拟DNA体内扩增的过程,在等温条件下扩增目的片段,其反应原理是在多种酶和蛋白质(重组酶、单链结合蛋白及聚合酶)的协同作用下,借助寡核苷酸和其他组分,实现体外靶核酸的快速扩增[2]。
重组酶与引物结合形成重组酶-引物复合物,该复合物在双链DNA模板中找到引物的同源序列,通过重组酶的链置换活性将引物插入到同源位点,单链结合蛋白结合替换下来的模板链,形成稳定的D环结构以保持单链的稳定性。接下来,重组酶从D环解体,同时聚合酶与引物的3′-OH末端结合,用于链延伸,形成新的互补链,重复上述步骤以实现模板上目标区域的指数扩增。整个反应过程非常快,一般情况下,可以在15 min左右完成扩增过程[13](图3)。
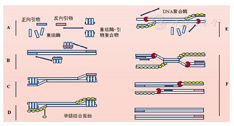

注:A:重组酶-引物复合体;B:复合物特异识别DNA双链;C:重组酶的链置换活性将引物插入到同源位点;D:单链结合蛋白与解离的单链形成稳定的D环结构;E:重组酶从D环解离,聚合酶绑定;F:聚合酶延伸引物,启动新DNA链的合成;新合成的DNA双链重复进行A~F的过程,实现目的DNA的序列扩增
RPA技术目前常见的检测方式主要有3种,分别是凝胶电泳、荧光探针以及结合侧流层析试纸条[4]。第一种方式是基础型RPA检测方案仅需在反应体系中加入一对特异性引物,先对产物纯化,再进行琼脂糖凝胶电泳即可实现对检测结果的判读[13];第二种方式是向反应体系中加入核酸外切酶Ⅲ(exonucleaseⅢ,exo)和exo荧光探针,或加入甲酰胺嘧啶-DNA糖基化酶(formamide pyrimidine DNA glycosylase,fpg)和fpg荧光探针,利用便携式等温扩增核酸检测仪即可实现荧光实时检测[14];第三种方式是侧流层析结合重组酶聚合酶扩增技术的检测方法,向反应体系中加入核酸外切酶Ⅳ(endonuclease Ⅳ,nfo)和nfo荧光探针,仅需使用一台水浴锅或恒温金属浴便可实现扩增,将扩增产物稀释后滴加在侧流层析试纸条上即可实现检测结果的肉眼可视化判读[15]。
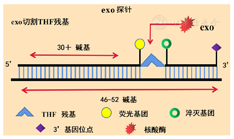
RPA技术涉及到的几种主流探针包括:exo荧光探针、fpg荧光探针和nfo荧光探针。其中,exo探针中一般含有1个四氢呋喃(tetrahydrofuran residue,THF)残基,其两侧分别为荧光基团和淬灭基团。一般情况下,exo探针长度为46~52碱基,THF残基的位置距离探针的5′端至少30碱基,距离探针的3′端至少15碱基。探针3′末端通过适当的修饰被封闭,以阻止聚合酶的进一步延伸。在合成双链DNA时,THF残基作为ExonucleaseⅢ的底物,被探针在THF残基的位置切割,使得荧光基团和淬灭基团分开,从而产生荧光信号[16,17](图4)。

nfo探针长度为46~52碱基,其内部核苷酸被THF残基取代。THF残基位于距离探针5′端至少30个碱基,距离探针3′端至少15个碱基。5′端用荧光基团标记,3′端标记封闭基团。在扩增过程中,当nfo探针与模板链互补时,核酸外切酶Ⅳ识别并切割THF残基位点,使探针3′端的封闭基团失效,从而聚合酶可以继续延伸,扩增获得既有探针标记物又有引物标记物的扩增产物。然后加入对应的标记抗体,就能看到试纸条检测线上的结果条带[18](图5)。
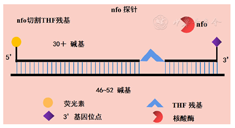

fpg探针法需要在RPA技术的基础上,加入甲酰胺嘧啶-DNA糖基化酶及fpg荧光探针,fpg荧光探针添加了4个修饰,分别为荧光基团、dR连接臂修饰、淬灭基团和3′端封闭基团,fpg酶会对dR连接臂(dR group,脱氧核糖核苷酸中的碱基被荧光基团替换,荧光基团与五碳糖通过C-O-C的结构相连,合成探针时可加入此修饰)位点进行特异性识别并切割,荧光基团从探针上分离,从而远离淬灭基团,即可发出荧光信号,然后通过检测仪器来实时监控荧光值的变化[18](图6)。

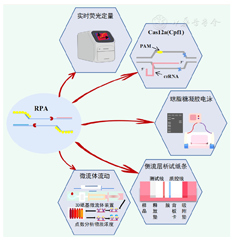
传统的基于PCR的分子检测方式耗时且需要精密昂贵的基因扩增仪,难以满足临床即采即检即报的需求。RPA技术因具有快速、操作简便、敏感度高和无需特殊设备等优点,近几年,在临床检测方面出现了较为新颖的技术模式和解决方案,充分展示了该技术的优势和临床应用潜力[19]。Ying等[20]提出了一种新型的实时重组酶聚合酶扩增检测方法(real-time recombinase polymerase amplification,RT-RPA),用于人乳头瘤病毒(human papilloma virus,HPV)16、18两种亚型的分型检测。此方法能够在较低病毒拷贝数的临床样本中检测HPV,并在25 min内报告结果。这种基于实时重组酶聚合酶扩增原理开发的RT-RPA检测方法解决了临床上对宫颈癌快速筛查的需求,体现了RPA技术在床旁检测环境中的独特优势。Liu等[21]在数字微流控(digital microfluidics,DMF)平台上将RPA与CRISPR进行整合优化,建立一种新方法用于幽门螺杆菌的基因分型检测,该系统可在30 min内实现不同样本中幽门螺杆菌多靶点检测,方法的敏感度和特异度均为100%,为临床检测幽门螺杆菌提供了一种检测性能更优、操作更便捷的检测方案。Hu等[22]开发了一种基于RPA结合CRISPR/Cas12a的分子检测方法,靶向检测临床样本中的诺如病毒,通过测流层析试纸条显示结果,检测性能良好,可用于腹泻疫情的快速鉴别检测(图7)。通过将RPA技术与其他前沿技术相结合,解决了临床对传统检测方法存在的耗时长、操作复杂及设备要求高等问题,极大提高了临床实验室的工作效率。

食品安全领域检测需求复杂繁多,涵盖如微生物检测、农药残留检测、兽药残留检测、非法添加剂检测、过敏原检测、重金属检测、食品掺假检测、转基因成分检测、营养成分检测等方面。其复杂的样本类型和特殊的场景需求对检测方法提出了挑战[23]。RPA技术作为一种新型的分子检测技术,对样本的高度适应性和在POCT方向广阔的应用潜力,使其在食品安全领域有着广泛应用。其中,Li等[24]通过RPA技术对沙门菌的invA基因进行等温扩增,通过光敏比色法实现直接定量,敏感度和特异度分别达到90%和95%,并可在1 h内完成检测,满足了食品安全的即时检测需求。Wang等[25]建立了RPA技术结合3条侧流层析条带的生物传感器,用于检测霍乱弧菌和创伤弧菌。仅需在37℃条件下反应30 min,即可在试纸上肉眼观察结果,且其检测性能与基于PCR的方法一致。展示了RPA技术的可塑性与便捷性,在应对资源匮乏场景下食源性病原体的快速检测有明显的优势。Huang等[26]采用RPA-CRISPR/Cas12a检测系统对羊奶粉中牛源性成分的掺假情况进行定性检测,此方法45 min内即可完成鉴定,且性能良好,为打击食品掺假提供了一种敏感度高且特异度强的技术参考[27]。RPA技术为食品安全管理提供了一种便携、特异度强及可视化的现场检测方式,特别对于食源性病原微生物检测方面有较为广阔的应用前景[7]。
环境监测涵盖了广泛的领域,包括空气、水体、土壤、噪声、辐射等多个方面。在这些领域里,常见的检测需求包括但不限于环境中重金属检测、有机污染物检测、微生物污染检测、土壤质量检测、生物多样性等方面。值得我们关注的是,RPA技术在环境微生物检测领域具备独特优势。环境中的病原体因其丰度低、种类多和高风险异质性对基于PCR的传统检测方式构成了挑战,常规检测方法存在成本高、通量有限、操作复杂和精确度低等局限性。RPA技术不受植物源扩增抑制剂的影响、特异度高且灵活便携,在现场植物诊断和环境安全监测方面具有较大的应用[28]。例如,Arif等[29]将RPA-LFD两者联合,在37℃下即可从受感染植物环境中检测出美国产毒细菌的毒素,比常规PCR方法敏感度更高,且不受植物抑制剂影响。Kim等[30]将逆转录重组酶聚合酶扩增结合侧流层析(reverse transcription recombinase polymerase amplification combined with lateral flow strips,RT-RPA-LFS)技术用于检测环境中的黄矮病毒,此方法在42℃条件下孵育10 min,即可在侧流层析条上观察扩增产物,且与其他主要病毒无交叉反应性。Chen等[31]利用RPA-CRISPR/Cas12a技术检测环境中的环状镰刀菌,此技术在37℃下,30 min内能够对环状镰刀菌的低浓度DNA进行特异性识别,检测结果可用肉眼来观察,该技术以其简便快速、高敏感度及结果可视化的优势,有效地降低了环状镰刀菌进入新地区的传播风险。RPA技术满足了环境中对复杂样本中病原体检测高敏感度和高特异度的需求,优良的检测降低了环境监测成本,提高了检测结果的准确性,且完全可实现现场部署,为环境中的病原体监测提供了更加丰富的选择。
由病毒、细菌和真菌病原体引起的疾病对农作物产量和品质造成严重影响,同时也对畜牧业发展和食品安全构成严重威胁。此外,这些病原体还可能引发动物疫病,进而影响肉类、奶制品等农产品的安全和市场供应[32]。面对这些复杂且多变的问题,传统的检测方法在速度、便捷性、敏感度和成本效益等方面难以满足当前的需求[33]。RPA技术以其高敏感度、低成本和快速便携等优势成为农业和畜牧业领域检测病原体的理想选择。Ren等[34]开发了一种基于RPA-CRISPR的高敏感度诊断方法,用于快速检测非洲猪瘟病毒。该方法可特异性区分该病毒与其他猪病毒核酸,且无交叉反应。此外,该方法也可用于诊断非洲猪瘟病毒临床DNA样本,对该病毒阳性和阴性样本的诊断符合率均达到100%,且敏感度与传统的qPCR方法相当甚至更高。因此,此方法极大地满足了养猪业食品检验和疾病控制的要求。Chen等[35]通过优化RT-RPA检测方法,用于检测小麦黄花叶病毒和中国小麦花叶病毒。实验验证,该方法对不同地区样本具有较高适用性,且能够有效检测单个或共感染植物中相应的病毒,表现出高度的特异性。这一优化为小麦病毒病的早期诊断、监测和鉴定提供了有力的技术支持。Sun等[36]利用RPA-CRISPR/Cas12a测定法用于鉴定两种大豆茎溃疡病病原真菌(Diaporthe aspalathi和Diaporthe caulivora),且检测时间不超过60 min。这种便携式、高特异度及现场可视化的技术有效地控制了真菌病原体在农作物中的传播。以上研究表明,RPA技术以其独特优势满足了田间和养殖现场的实时监测,大大提高了检测的灵活性和时效性,为防止病原体传播和确保粮食安全起到了至关重要的作用。
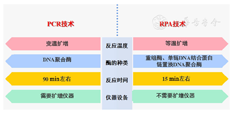
RPA技术和PCR技术的目的是一致的,都是为了在体外对靶基因进行扩增。但两种技术的原理不同,导致它们的反应过程和应用场景不同。二者之间的关系有两大特点:(1)相互独立:从技术原理的角度看它们是两种独立的、并行的技术;从技术平台的角度,它们所依托的平台和设备明显不同;从技术应用的角度他们有不同的应用场景。(2)互相补充:RPA技术在室外现场的环境应用中具有一定优势,例如对基层资源匮乏地区的现场即时即检,这与PCR技术在实验室检测形成有效互补,为复杂环境和偏远地区病原体检测提供了便利。
新型酶的开发和优化对于提高RPA技术的性能至关重要。RPA技术的核心技术是依赖于特定的酶(通常是重组酶和DNA聚合酶)来实现DNA的高效扩增。新型酶的开发和优化对于提高特异度、增强稳定性、扩大应用范围、提升扩增效率、简化操作流程、降低成本等方面意义重大。如当前重组酶蛋白在RPA试剂制备过程中,对温度的敏感性较高。一方面,可通过开发其他嗜热生物体来源的热稳定重组酶,降低对温度的敏感性以适应恒温环境。另一方面,可以利用基因工程技术方法,借助计算机对重组酶的活性部位进行需求设计,改造成在体外具有更高稳定性、重组活性更强的重组酶,提高检测敏感度及催化效率。
人工智能(artificial intelligence,AI)正在改变我们对生物医学的理解和实践方式,它不仅提高了诊断和治疗的效率,还为新技术开发和基础研究提供了新的途径和方法。对于RPA技术的开发,AI可以在3个方面提供有益的帮助。其一,通过AI算法对引物进行优化,根据预测结果自动选择最佳引物序列。一些商业软件已经开始尝试将AI引入到引物设计过程中,例如EAassay软件可在线设计RPA引物,提高了RPA引物设计的效率;PrimedRPA软件包可自动创建和过滤RPA引物和探针,通过对比多个序列来识别保守的靶标,并过滤可能与背景生物发生交叉反应的区域,旨在解决引物与探针不适配的问题,通过AI,可减少人工对引物设计的干预,提高效率和准确性。其二,借助AI大模型,模拟酶的筛选和迭代改造过程,以获得更高催化活性、更加稳定理想的酶版本。酶的改造需要对表达、稳定性、构象可塑性和催化功能等多个目标进行优化。重组酶和DNA聚合酶在使RPA技术实现高效扩增过程中扮演着关键角色。因此,通过AI有效地设计并合成这些相关酶,对RPA技术的进步将产生重大而深远的影响,这也为我们确定未来的研究方向提供了独特视角。其三,使用AI算法对RPA反应条件、模板DNA浓度、不同的引物以及最终的扩增效率进行实验前的计算机模拟分析,建立相关反应模型,以此帮助研究人员选择合适的温度、引物和反应时间,再进行真实世界的实验验证,从而节约相关的实验成本。
X技术代表先进新技术,代表对未来技术的憧憬,近年来典型的X技术如CRISPR/Cas、LFD、DMF、纳米技术、量子点技术、近红外荧光探针等。RPA技术在扩增过程的优势明显,但是样本预处理和结果检测可视化环节还存在问题,X技术的不断涌现,对于解决这两大环节存在的问题意义显著,如CRISPR/Cas系统可以用来删除不需要的基因序列或者修复目标序列上的错误,从而减少样本复杂性,提高RPA的特异度。量子点具有荧光特性,可作为标记物用于检测RPA产物,其高亮度和稳定性有助于提高检测的敏感度和分辨率。通过整合新兴X技术,可以大大提高RPA在样本预处理阶段和结果检测可视化环节的效率和准确性,降低操作难度,减少污染风险,并提升整个检测流程的自动化水平。
综合近几年RPA技术最新研究进展,RPA与多种新兴技术联合应用并实现POCT化将是该技术未来几年发展的趋势。POCT主要优势是在即采即测,RPA的特点是优良的检测性能和强大的现场适用性,二者搭配,进而可进一步扩宽其应用范围。随着分子生物学技术不断创新和发展,未来RPA技术必然能够在临床诊断、食品安全、环境监测等领域发挥更好的作用。总而言之,RPA是继PCR之后有较高实用价值的新型分子检测技术,具有巨大的发展潜力。
胡颖,张潭涛,龙雨霖,等.重组酶聚合酶扩增技术的应用新进展与思考[J].中华微生物学和免疫学杂志,2024,44(8):665-671. DOI: 10.3760/cma.j.cn112309-20240529-00180.
所有作者声明无利益冲突



























