
炎症性肠病(IBD)作为一种慢性非特异性肠道炎性疾病,其发病受免疫、基因和环境因素之间复杂的相互作用影响,肠道微生态也在IBD的发展中发挥重要作用。肠道微生态的破坏、有益肠道菌群的减少以及有害微生物的增多,包括真菌、病毒、寄生虫等的丰度改变,会导致肠道屏障的破坏,微生物的相互作用失调,以及免疫系统紊乱在内的多种生态失衡,进而导致IBD等多种疾病的发生。而以有益肠道微生物,尤其是益生菌、益生酶、益生元以及粪菌移植为代表的基于肠道微生态的治疗方法,已成为IBD有前景的治疗策略。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
炎症性肠病(inflammatory bowel disease,IBD)是一种慢性炎性疾病,发病率逐年升高,缺乏有效治疗方式[1]。其发病机制一直被广泛研究,其中肠道微生态的改变尤其是肠道菌群的变化对IBD发病的影响受到越来越多的关注。本文探讨了肠道微生态在IBD发病机制中的作用及改善肠道微生态治疗IBD的潜力,以期为IBD的治疗带来新的思考。
肠道微生态是一种有机动态的平衡状态[2],由超过1014种不同的微生物及其生存环境如肠道上皮细胞和肠道黏膜免疫系统共同构成[3]。肠道微生物主要包括细菌、病毒、真菌和寄生虫等。其中肠道菌群是核心部分,而肠上皮屏障结构及功能对系统的正常运行有重要影响。在正常情况下,肠道微生物和肠黏膜及免疫系统相互协调。肠道微生物参与营养代谢,抑制有害微生物的生长。此外,肠道微生态对于维持人体的天然免疫屏障及物理屏障功能也至关重要。当肠道微生态的稳态失衡时,肠道微生物群网络发生变化[4]。大量研究表明,肠道稳态紊乱与炎症性疾病以及代谢、心血管等疾病密切相关[2]。肠道微生态破坏会导致包括IBD在内的各种肠内外疾病的发生。因此,维持肠道微生态平衡对人类维持机体稳态及抵抗疾病至关重要。
IBD主要包括克罗恩病(Crohn's disease,CD)和溃疡性结肠炎(ulcerative colitis,UC),其发生发展可能与肠道微生物、免疫因素、环境因素和遗传易感性有关[2]。其中肠道微生态的失调可能对IBD患者的发病机制产生至关重要的影响(图1)。
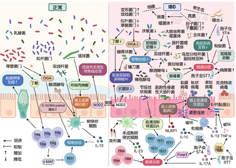

注:OGA为O-连接的β-N-乙酰氨基葡萄糖苷酶;Treg为调节性T细胞;c-MAF为细胞内肌肉腱膜肉瘤,是一种转录因子;Th17为辅助性T细胞17;NOD2为含核苷酸结合寡聚化结构域2;IL-10为白细胞介素10;TRM为组织驻留记忆T细胞;IL-23为白细胞介素23;NLRP1为含Pyrin结构域的NOD样受体1,富含亮氨酸重复序列和含Pyrin结构域3;Foxp3为叉头蛋白P3;IL-17A为白细胞介素17A;IL-1β为白细胞介素1β;TNF-α为肿瘤坏死因子α
菌群失调是IBD患者肠道微生态变化的重要特征之一[5]。健康人群肠道微生物组的主要成员厚壁菌门和拟杆菌门在IBD患者中减少,而放线菌门和变形菌门丰度升高。IBD相关肠道微生态失调的一个特征是普拉梭菌比例降低[5]。普拉梭菌是丁酸盐生产者,许多研究已提出普拉梭菌作为人类肠道健康的标志物和关键参与者,其丰度下降可能对IBD的发病产生重要影响[6,7]。此外,既往肠道微生物组的大多数临床研究均依赖粪便样本,不能代表区域特异性肠道微生物群。因此,越来越多的研究转向内镜方法来获取特定区域的样本。IBD患者肠道菌群改变的另一个特征是致病菌的丰度升高[8]。鸟型分枝杆菌副结核亚种在CD患者的肠道中被广泛检测到,研究发现其可定植于CD患者的回肠黏膜,与CD特异性相关[9]。此外,一项黏膜活检研究表明,与健康对照组相比,CD和UC患者的黏附性侵袭性大肠杆菌显著富集[10]。最近研究还表明,从IBD患者的炎症部位活检组织中分离出的具核梭菌在Caco-2细胞(肠上皮细胞)侵袭试验中较健康对照者分离出的菌株更具侵袭活性[11]。
探索肠道微生物组其他微生物(如真菌、病毒及寄生虫)作用的研究较少,然而这些微生物对IBD的发生发展也有重要的影响,并影响其药物治疗的反应性[12]。研究发现IBD患者肠道真菌群主要由担子菌门和子囊菌门构成,其中子囊菌门较为丰富,念珠菌最多,其次是棒孢酵母[12]。与正常对照组相比,IBD患者真菌菌群偏斜,担子菌与子囊菌比值升高,酿酒酵母比例降低,白色念珠菌比例增加[13]。真菌可能参与某些类型的IBD,如血清生物标志物抗酿酒酵母抗体(ASCA)阳性CD的发生[14]。许多研究发现IBD患者中白色念珠菌增加,特别是CD患者[13,15]。另一种真菌物种Malassezia restricta在部分CD患者的结肠黏膜样本中含量也升高,其可能通过抗真菌防御的胱天蛋白酶富集域家族成员9(CARD9)和Dectin-1(一种先天性真菌β-葡聚糖受体)的下游引起炎症反应[16]。真菌在IBD中的作用越来越受关注,未来的研究需要进一步探讨真菌参与IBD的具体机制,以及基于此开发新的预防和治疗策略。
IBD患者肠道微生态中的病毒群落也发生了变化。1982年的一项流行病学研究旨在结合血清学分析检测IBD患者粪便标本中的诺如病毒和轮状病毒抗原,评估疾病活动度与病毒感染证据的相关性,发现8例有肠道病毒感染血清学证据的患者中,有5例在3个月内出现症状恶化[17]。另一项流行病学研究显示UC复发和粪便中存在诺如病毒呈显著相关性[18]。然而另一项在荷兰队列中进行的前瞻性研究旨在对286例IBD患者进行每3个月一次的为期1年的随访,以检测粪便样本中的肠道病毒,结果表明,肠道病毒在基线和活动期IBD的流行率较低,疾病活动和肠道病毒感染之间没有发现流行病学联系[19]。此外,噬菌体是在细菌内寄生和复制的病毒,它们掺入细菌基因组会影响细菌基因的表达和功能。已有深度宏基因组测序报道了IBD患者和小鼠的管腔、黏膜样本中某些噬菌体种类的变化[20,21]。研究证实,IBD肠道噬菌体种群发生改变,并从有序状态转变为无序生态失调。其中UC患者黏膜噬菌体群的改变表现为有尾噬菌体的富集、噬菌体毒力增强以及肠道病毒-细菌相关性的破坏[20]。还有研究发现CD患者小肠黏膜噬菌体群出现明显的紊乱,表现为靶向病原菌噬菌体的显著缺失,以及托克特诺病毒的显著增多[22],且缺乏双歧杆菌与Lachnospiraceae主导的细菌和噬菌体之间的互惠作用。令人惊讶的是,这种情况在CD缓解期比发作期更明显,进一步表明了CD黏膜炎症的难治性和复发性[22]。
对于IBD患者肠道寄生虫相关的研究较少,其中广泛认可的是对于IBD患者蠕虫暴露的理解[23]。有研究发现由于现代高度卫生的生活方式,生活在工业化国家的人们基本失去对蠕虫的常规暴露,IBD的患病风险会大大增加[24]。"IBD卫生假说"假设减少接触传染性病原体会限制免疫系统的发育,从而导致重要免疫调节回路的功能不全,免疫系统调节受损会导致包括IBD在内的免疫介导的疾病的发展[25]。生理状态下,许多蠕虫生活在人类肠道内,可与黏膜免疫系统相互作用。宿主产生黏膜反应包括辅助性T细胞2(T helper cell 2,Th2)及其细胞因子的产生来限制蠕虫定植。寄生虫引发的Th2反应可以调节对感染的免疫反应。而未能获得这些寄生虫并经历黏膜Th2调节可能会导致Th1细胞过度活跃及CD的发生[24]。在高度卫生国家,蠕虫类生物已基本根除,因此儿童和成人缺乏相关暴露,这可能是IBD患病率增加的重要原因。而使用蠕虫治疗IBD可能具有一定的应用潜力[26],后文将进行说明。
肠道屏障是人体中最广泛和重要的屏障之一,主要由共生的微生物、黏液层和肠上皮细胞构成,将肠腔内容物和人体分隔开[27],防止病原入侵。此外,上皮细胞之间的紧密连接是肠上皮细胞屏障极其重要的组成部分[27]。紧密连接的破坏会增加肠道通透性,允许各种微生物及其产物和外来抗原等外源性物质进入黏膜和毛细血管,导致炎性相关因子的分泌并激活免疫系统,进而导致各种肠道相关疾病的发生。
肠道微生态的破坏可导致肠道屏障功能受损。肠道细菌产生的短链脂肪酸如丁酸可促进杯状细胞产生黏液,从而加强黏膜屏障功能。IBD患者肠道中产丁酸的细菌,如普拉梭菌、丁酸梭菌、罗氏菌属的数量减少[28],导致肠道黏膜屏障受损。此外,链球菌芽孢产生吲哚丙烯酸可增强肠上皮屏障功能及减轻炎症反应,而IBD患者肠道中的微生物利用黏蛋白和代谢色氨酸的能力减弱,吲哚丙烯酸的合成减少,也导致屏障功能障碍[29]。表达于肠上皮细胞的核苷酸结合寡聚化结构域蛋白2(nucleotide-binding oligomerization domain-containing protein 2,NOD2)作为细胞内细菌防御因子在共生微生物的免疫应答中发挥作用。IBD患者中NOD2基因突变会导致黏液层缺陷、屏障破坏及抗菌肽减少和致病菌增多[30]。此外,IBD患者中黏液降解细菌的总丰度显著增加[31]。健康人体内的黏液降解细菌可降解黏液黏蛋白释放产物供其他细菌使用[32],但黏液降解细菌如瘤胃球菌的急剧增加,将溶解更多的黏液并增加上皮细胞的暴露[31]。另外,除了黏液屏障,IBD肠道上皮细胞的紧密连接也被破坏。IBD伴随免疫系统的紊乱,免疫系统激活产生的细胞因子将进一步攻击肠上皮细胞的紧密连接,超过肠道屏障的自愈能力,导致IBD持续进展。例如IBD中升高的炎性因子白细胞介素(interleukin,IL)-1β可募集粒细胞浸润感染病灶,直接破坏肠上皮细胞的连接和紧密性[33]。此外,肿瘤坏死因子α(tumor necrosis factor-α,TNF-α)和IL-1β可诱导内质网应激,影响Caco-2细胞,并显著改变顶端和基底外侧膜的E-钙黏蛋白,进一步破坏肠上皮的紧密连接[34]。此外,IBD患者肠道炎症区域的含Pyrin结构域的NOD样受体1(NLRP1)炎性小体急剧增加,可诱导肠上皮细胞凋亡并进一步增加肠道通透性,促进IBD的发生发展[35]。
由于细菌的代谢产物可以作为其他共生菌的生产原料,IBD患者肠道的菌群互作破坏可进一步加重肠道微生态的失衡。IBD患者肠道中的致病共生菌显著增加[36]。同时,肠道有益的共栖细菌和产丁酸盐细菌的比例显著降低,导致以短链脂肪酸为生产原料的其他菌群随之减少,这也是IBD发展的重要因素。拟杆菌门和厚壁菌门中富含的O-连接的β-N-乙酰氨基葡萄糖苷酶(O-GlcNAcase,OGA)能够水解上皮细胞和免疫细胞中的O-GlcNAcylated蛋白,从而抑制结肠炎的发展。然而,由于细菌的减少,IBD患者的OGA水平降低[37]。近年来,科学家发现真菌也可协调共生细菌的生长,它们之间存在错综复杂的关系。UC和CD的真菌改变存在较大差异,这主要与病变部位有关,由于回肠具有产生抗菌肽和吸收胆汁酸的独特功能,回肠CD有利于真菌的生长。真菌的丰度改变也与肠道内的一些共栖细菌有关。IBD的真菌变化主要表现为担子菌门的比例增加,酿酒酵母的比例减少,酿酒酵母具有抑制黏附侵袭性大肠杆菌诱导结肠炎的能力。而肠道中双歧杆菌和布鲁氏菌的丰度与酵母呈正相关,其在IBD肠道中的丰度均下降[13]。此外,肠道病毒和细菌之间具有复杂的相互作用,肠道真核细菌可以促进病毒性肠道感染,肠道病毒可以促进细菌感染[38]。微生物组研究表明,人类诺如病毒感染可能会破坏肠道微生物群,导致变形菌数量增加和拟杆菌数量减少[39]。小鼠模型进一步证明,鼠诺如病毒甚至可以塑造微生物群,并取代无菌小鼠中共生细菌的功能[40]。未来还需要进一步的研究来阐明IBD患者体内病毒和细菌相互作用的机制,以及它们在IBD发生发展中的作用。
肠道微生态的破坏导致肠道免疫系统失调。IBD患者肠道中的变形菌门丰度升高,在大量变形菌穿过黏膜屏障后,变形菌表面的病原体相关分子模式、脂多糖和鞭毛蛋白可被树突状细胞表面的toll样受体识别[41,42],树突状细胞产生IL-23,导致局部肠道炎症反应[43]。此外,双歧杆菌及与其正相关的酿酒酵母均可促进抗炎细胞因子IL-10释放,但在IBD患者肠道中其丰度均下降[13,44]。肠道共生菌的代谢产物丙酸盐可抑制γδ T细胞产生IL-17[45]。但由于产生丙酸盐的细菌丰度降低,这种作用也被抑制,导致炎性因子增多以及炎症发生。此外,短链脂肪酸还可以诱导产生抗炎调节性T细胞(regulatory T cell,Treg)和其产生的c-MAF转录因子,后者可以维持Treg的功能并抑制Th17的激活[46]。但是因为厌氧菌的丰度降低,短链脂肪酸的产生减少,这种抗炎机制被明显抑制。不仅如此,节段丝状细菌在IBD的定植增加可以触发血清淀粉样蛋白A的分泌,促进致病性Th17细胞的分化[47]。最近研究发现,组织驻留记忆T细胞(tissue resident memory T cells,TRM)在促进IBD炎症发展中也起着重要作用[48]。虽然肠道微生物改变与TRM之间的相关机制尚不明确,但仍极有可能成为未来有效的治疗靶点。此外,寄生虫的丰度变化也可影响到IBD肠道免疫。研究表明,孢子虫ST4的预先定植通过提高有益菌的丰度、短链脂肪酸的产生以及分泌IL-10的Treg的比例,可以保护葡聚糖硫酸钠(dextran sulfate sodium,DSS)诱导的结肠炎。相反,孢子虫ST7感染通过增加病原菌的比例和诱导可分泌IL-17A和TNF-α的炎性CD4+ T细胞而加重结肠炎症[49]。
IBD患者肠道微生态改变伴随益生菌的丰度降低,而补充益生菌可有效调节肠道菌群失衡、增强肠道黏膜屏障及免疫功能,成为IBD新的治疗方法。益生菌鸡尾酒疗法对于治疗IBD也有明显益处,8种益生菌的混合物VSL#3可通过抑制高迁移率族蛋白B1(high mobility group box 1 protein,HMGB1)的释放及HMGB1介导的肠道屏障功能障碍,从而改善小鼠的肠道炎症[50]。一些有益微生物如普拉梭菌、罗伊氏乳杆菌、阿克曼菌及其产物是治疗IBD的潜在药物[51,52,53]。其中嗜黏蛋白阿克曼菌(Akkermansia muciniphila,A.muciniphila)作为明星细菌受到广泛关注,施用A.muciniphila可减轻结肠炎小鼠肠炎症状和降低组织病理学评分[54]。此外,本课题组成功分离纯化A.muciniphila外泌体蛋白—GCN5相关N-乙酰转移酶Amuc_2172,其是一种由肠道微生物产生的、可作用于宿主细胞、改善宿主细胞功能、缓解宿主疾病的酶,并将其定义为益生酶(probiotic enzyme)。益生酶Amuc_2172可通过巨胞饮作用进入细胞,并在Lys14位点上乙酰化组蛋白Histone H3(H3K14ac),促进肿瘤细胞中热休克蛋白70(HSP70)的转录和分泌及细胞毒性T淋巴细胞的激活,从而达到杀伤肿瘤的作用。益生酶Amuc_2172已被证实在结直肠癌等多种肿瘤模型中具有保护作用[55],其在IBD中的作用也值得进一步研究。此外,其他益生菌是否也会通过产生益生酶来缓解宿主疾病也值得关注。
FMT通过将健康人群粪便中的功能性细菌移植到患者的胃肠道中,重建IBD患者肠道菌群,为IBD的治疗带来新的策略。最初接受FMT治疗的UC患者在内镜和组织学上保持20年无病生存[56]。关于CD的FMT治疗,也有一些研究提示临床和内镜缓解[57,58,59,60]。总体而言,这些发现表明FMT是IBD的有效治疗方法。然而,还需要做更多的工作来评估这些患者的临床疗效以及肠道微生物群的变化,以及预防并发症的发生。
在IBD中,大多数共生有益微生物因环境恶劣而丰度降低[61]。益生元是膳食纤维补充剂,可选择性地刺激有益菌的生长及促进其活性[62]。合生元是指同时含有益生菌和益生元的混合制剂,其涉及用营养来源(益生元)为益生菌菌株提供竞争优势,以增加其定植机会[50]。在一项研究中,益生元Synergy 1与长双歧杆菌联合使用可改善UC患者活检样本中的乙状结肠镜检查评分并降低炎性因子的水平[63]。然而,如果益生菌没有成功定植,合生元可能需频繁、大剂量给药,使患者更难依从。益生元与合生元在IBD临床患者中的应用有效性及安全性仍需要更多研究来验证。
蠕虫疗法是指给患者接种特异性肠道寄生蠕虫,包括美洲钩虫[64]或猪鞭虫、鞭虫卵或幼虫[65]以及Heligmosomoides polygyrus bakeri[66]等。暴露于这些蠕虫可预防或逆转IL-10基因缺陷(IL-10-/-)小鼠的结肠炎[67]。在过去的几年里,已经开发了"药化"猪鞭虫的方法,相关的大型多中心临床试验正在进行中,以期为IBD患者带来新的疗法[68]。以上疗法示意图可参见图2。
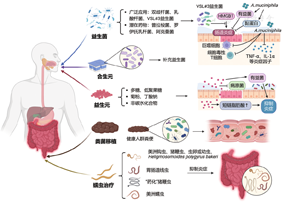

注:HMGB1为高迁移率族蛋白B1;TNF-α为肿瘤坏死因子α;IL-1α为白细胞介素1α
综上所述,随着我国IBD发病率的升高及对IBD发病及治疗的不断探索,肠道微生态在IBD发病机制以及治疗中的作用受到越来越多的关注。肠道微生态的改变导致肠道屏障的破坏,免疫系统的失调可能会促进IBD等炎性疾病的发生,而包括益生菌在内的调节肠道微生态的方法可能为IBD的治疗带来新的策略与思考。目前有益微生物治疗IBD的潜力及其有效成分广受关注。未来需要更加深入探讨微生物群与IBD相互作用的具体机制,以期提高患者的生活质量和治疗效果。
所有作者均声明不存在利益冲突
感谢上海高水平地方高校创新团队(SSMU-ZLCX20180200)对本文的支持



























