
探讨AcrAB–tolC外排泵及marA–soxS–rob调控系统对福氏志贺菌环丙沙星耐药性的影响。
连续收集45株2009至2013年分离自天津地区多家三级甲等医院肠道门诊患者粪便样本的福氏志贺菌。采用改良Kirby–Bauer纸片扩散法对菌株进行药物敏感性试验。随机选择10株环丙沙星耐药株和10株环丙沙星敏感株,分析其靶位酶基因(gyrA和parC),质粒介导的喹诺酮耐药基因(PMQR),外排泵基因(acrA和acrB)及调控基因(marA、soxS和rob)的携带情况,并进行序列分析。通过泵抑制剂羰基氰氯苯腙(CCCP)抑制试验检验外排泵基因及调控基因对菌株最低抑菌浓度(MIC)的影响,同时应用实时荧光定量PCR(RT–PCR)方法检测外排泵基因及调控基因的表达水平,并采用t检验比较这些基因在环丙沙星耐药株和敏感株中表达水平的差异。
10株环丙沙星耐药株均存在gyrA和parC突变,环丙沙星耐药株CR2同时携带qnrS1,环丙沙星耐药株CR5同时携带aac(6′)–Ib–cr基因。10株环丙沙星敏感株均未检测到外排泵基因及调控基因突变;而10株环丙沙星耐药株中,除CR10外,其余菌株均检出soxRS突变。加入CCCP后,喹诺酮类药物对10株环丙沙星耐药株的MIC均下降至原值的1/4~1/8,而对10株环丙沙星敏感株的MIC无变化或仅下降至原值的1/2。与环丙沙星敏感株相比,环丙沙星耐药株acrA、acrB、marA和soxS基因的表达水平明显升高(P<0.05或P<0.01)。
外排泵机制可能在福氏志贺菌对环丙沙星耐药中发挥重要作用,marA–soxS–rob调控系统发挥着调节泵基因表达的作用,且soxRS基因序列的改变可能是影响此调控系统的重要原因。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
志贺菌所致细菌性痢疾是影响全球公共健康卫生的重要问题。目前,喹诺酮类药物是治疗成人细菌性痢疾的一线抗菌药物。国内外有关志贺菌的耐药性监测表明,志贺菌对氟喹诺酮类抗菌药物尤其是环丙沙星的耐药率呈逐年上升趋势,以福氏志贺菌为著[1,2]。究其耐药机制,除了靶位酶基因突变和携带质粒介导的喹诺酮耐药基因(PMQR),近年来,外排机制有助于细菌对氟喹诺酮类抗菌药物的耐药性在肠杆菌科细菌中得到证实,如大肠埃希菌、肺炎克雷伯菌、沙雷氏菌等[3,4,5]。本研究针对天津地区近5年收集的对环丙沙星耐药福氏志贺菌临床菌株,在系统分析其耐药机制的基础上,探讨AcrAB–tolC外排泵及调控系统marA–soxS–rob对其耐药性的影响。
实验菌株为45株福氏志贺菌,来自2009至2013年天津地区多家三级甲等医院肠道门诊患者的粪便样本,经常规生化及血清凝集试验证实。水解酪蛋白(MH)琼脂购自上海伊华生物科技有限公司。泵抑制剂羰基氰氯苯腙(CCCP)为Sigma公司产品,纯度≥ 97%。实验所用17种抗菌药物药敏纸片购自北京天坛生物制品研究所。引物合成及PCR产物测序由北京六合华大基因技术有限公司完成。
所有菌株严格按照美国临床实验室标准化协会(CLSI)2010年的标准[6],采用改良Kirby–Bauer纸片扩散法检测。对所选10株环丙沙星耐药株及敏感对照株采用微量肉汤稀释法测定其对萘啶酸、环丙沙星、氧氟沙星、诺氟沙星及左氧氟沙星的最低抑菌浓度(MIC),质控菌株为大肠埃希菌ATCC 25922。
与上述MIC测定同时进行。在含上述抗菌药物的二倍稀释浓度的MH琼脂平板中加入CCCP至终浓度为100 μmol/L,测定泵抑制剂存在情况下抗菌药物对志贺菌的MIC,同时接种仅含等浓度泵抑制剂的MH培养基作为生长对照。比较使用泵抑制剂前后同一菌株对抗菌药物MIC的变化,MIC降至原值的1/4 (2个二倍稀释度)或更低判定为外排泵阳性株。
采用煮沸法提取细菌总DNA作为PCR反应的扩增模板。对菌株分别进行靶位酶基因gyrA、parC;质粒介导的喹诺酮耐药基因qnr,aac(6′) –Ib–cr,qepA及外排泵基因和调控基因acrR、marOR、soxRS及rob基因扩增,反应引物及特征见表1。反应体系为25 μL,其中Premix taq酶12.5 μL,ddH2O 9.5 μL,DNA模板1 μL,上下游引物各1 μL。PCR反应条件:94 ℃预变性5 min,94 ℃变性45 s,55℃退火1 min,72 ℃延伸1 min,共30个循环,72 ℃充分延伸10 min。取PCR反应产物1%琼脂糖凝胶电泳,溴化乙锭染色,凝胶系统下观察结果。对PCR反应产物测序,结果经Blast程序与GenBank数据库公布的标准菌序列进行比对。
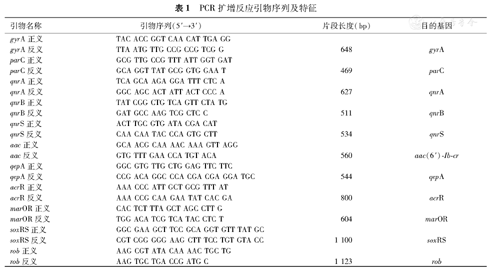
| 引物名称 | 引物序列(5′→3′) | 片段长度(bp) | 目的基因 |
|---|---|---|---|
| gyrA正义 | TAC ACC GGT CAA CAT TGA GG | ||
| gyrA反义 | TTA ATG TTG CCG CCG TCG G | 648 | gyrA |
| parC正义 | GCG TTG CCG TTT ATT GGT GAT | ||
| parC反义 | GCA GGT TAT GCG GTG GAA T | 469 | parC |
| qnrA正义 | TCA GCA AGA GGA TTT CTC A | ||
| qnrA反义 | GGC AGC ACT ATT ACT CCC A | 627 | qnrA |
| qnrB正义 | TAT CGG CTG TCA GTT CTA TG | ||
| qnrB反义 | GAT GCC AAG TCG CTC C | 511 | qnrB |
| qnrS正义 | ACT TGC GTG ATA CGA CAT | ||
| qnrS反义 | CAA CAA TAC CCA GTG CTT | 534 | qnrS |
| aac正义 | GCA ACG CAA AAC AAA GTT AGG | ||
| aac反义 | GTG TTT GAA CCA TGT ACA | 560 | aac(6′)–Ib–cr |
| qepA正义 | GGC GTG TTG CTG GAG TTC TTC | ||
| qepA反义 | CCG ACA GGC CCA CGA CGA GGA TGC | 544 | qepA |
| acrR正义 | AAA CCC ATT GCT GCG TTT AT | ||
| acrR反义 | AAA CCG CAA GAA TAT CAC GA | 800 | acrR |
| marOR正义 | CAC TCT TTA GCT AGC CTT G | ||
| marOR反义 | TGG ACA TCG TCA TAC CTC T | 604 | marOR |
| soxRS正义 | GGC GAA GCT TCC GCA GGT GTT TAT GC | ||
| soxRS反义 | CGT CGG GGG AAG CTT TCC TGT GTA CC | 1 100 | soxRS |
| rob正义 | AAG CGT ATA CAA AAC TGC TG | ||
| rob反义 | AAG TGC TGA CCG ATG C | 1 123 | rob |
RNA提取采用美国Biomiga公司的EZgene™细菌RNA小量提取试剂盒(R6616),依据产品说明操作;cDNA的合成采用天根生化科技(北京)有限公司的Quant cDNA第一链合成试剂盒并依据产品说明操作。
实时PCR检测采用美国Bio–Rad公司的IQ™ SYBR Green Supermix试剂盒。定量PCR仪为iQ5实时PCR检测系统(Bio–Rad, Hercules, CA),定量PCR扩增反应采用的引物序列见表2。用2△Ct法进行统计处理(△Ct=目的片段的Ct值–内参的Ct值),每个数值重复3次,每个结果重复计算两次。
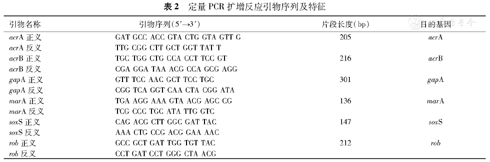
| 引物名称 | 引物序列(5′→3′) | 片段长度(bp) | 目的基因 |
|---|---|---|---|
| acrA正义 | GAT GCC ACC GTA CTG GTA GTT G | 205 | acrA |
| acrA反义 | TTG CGG CTT GCT GGT TAT T | ||
| acrB正义 | TGC TGG CTG CCA CCT TCC GT | 216 | acrB |
| acrB反义 | CGA GGA TAA ACG CCA GCG AGG | ||
| gapA正义 | GTT TCC AAC GCT TCC TGC | 301 | gapA |
| gapA反义 | CGG TCA GGT CAA CTA CGG ATA | ||
| marA正义 | TGA AGG AAA GTA ACG AGC CG | 136 | marA |
| marA反义 | TCG CCC TGC ATA TTG GTC | ||
| soxS正义 | CAG ACG CTT GGC GAT TAC | 147 | soxS |
| soxS反义 | AAA CTG CCG ACG GAA AAC | ||
| rob正义 | GCC GCT GAT TGG TGT TAC | 212 | rob |
| rob反义 | CCT GAT CCT GGG CTA ACG |
采用SPSS 11.1统计软件进行数据分析,先进行正态性检验,符合正态分布的计量数据用 ±s表示,组间比较采用t检验,P<0.05为差异有统计学意义。
±s表示,组间比较采用t检验,P<0.05为差异有统计学意义。
45株福氏志贺菌均对萘啶酸耐药,在氟喹诺酮类抗菌药物中,菌株对环丙沙星的敏感性最低,不足50%,其次为诺氟沙星和氧氟沙星,对左氧氟沙星的敏感性为75.6%;在第三代头孢菌素中,菌株对头孢他啶的敏感性最高;对氨苄西林、链霉素、复方磺胺甲噁唑和四环素的敏感性低于20%;未发现亚胺培南、头孢哌酮/舒巴坦及阿米卡星耐药株,见表3。
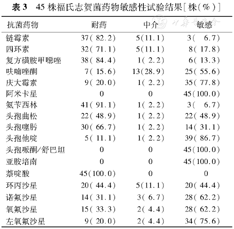
| 抗菌药物 | 耐药 | 中介 | 敏感 |
|---|---|---|---|
| 链霉素 | 37(82.2) | 5(11.1) | 3( 6.7) |
| 四环素 | 32(71.1) | 5(11.1) | 8(17.8) |
| 复方磺胺甲噁唑 | 38(84.4) | 1(2.2) | 6(13.3) |
| 呋喃唑酮 | 7(15.6) | 13(28.9) | 25(55.6) |
| 庆大霉素 | 9(20.0) | 1(2.2) | 35(77.8) |
| 阿米卡星 | 0 | 0 | 45(100.0) |
| 氨苄西林 | 41(91.1) | 1(2.2) | 3( 6.7) |
| 头孢曲松 | 22(48.9) | 1(2.2) | 22(48.9) |
| 头孢噻肟 | 30(66.7) | 1(2.2) | 14(31.1) |
| 头孢他啶 | 5(11.1) | 1(2.2) | 39(86.7) |
| 头孢哌酮/舒巴坦 | 0 | 0 | 45(100.0) |
| 亚胺培南 | 0 | 0 | 45(100.0) |
| 萘啶酸 | 45(100.0) | 0 | 0 |
| 环丙沙星 | 20(44.4) | 5(11.1) | 20(44.4) |
| 诺氟沙星 | 14(31.1) | 3(6.7) | 28(62.2) |
| 氧氟沙星 | 15(33.3) | 2(4.4) | 28(62.2) |
| 左氧氟沙星 | 9(20.0) | 2(4.4) | 34(75.6) |
随机选择10株环丙沙星耐药株(CR1~10)和10株环丙沙星敏感株(CS1~10)进行靶位酶基因gyrA、parC及PMQR基因qnr、aac(6′)–Ib–cr和qepA扩增,结果见表4。10株耐药菌全部扩增出gyrA片段和parC片段。测序结果显示,10株耐药株均出现gyrA83和parC80位点突变,7株同时存在gyrA83、87及parC80三个位点突变,3株缺乏gyrA87位点突变,其中gyrA87位点存在两种突变类型:菌株CR10由天冬氨酸变为天冬酰胺,而其他菌株在此位点由天冬氨酸变为甘氨酸。PMQR基因的扩增及测序显示菌株CR2携带qnrS1基因,菌株CR5携带aac(6′)–Ib–cr,其他菌株未出现PMQR基因扩增条带。10株敏感对照菌(CS1~10)均出现靶位酶基因gyrA83位点突变,但未出现PMQR基因扩增条带。

| 菌株 | PMQR基因突变 | 靶位酶基因突变 | 外排泵调控基因突变 |
|---|---|---|---|
| CR1 | – | gyrA(S83L,D87G) parC(S80I) | soxR (C122L) |
| CR2 | qnrS1 | gyrA (S83L) parC (S80I) | soxR (G140R) |
| CR3 | – | gyrA (S83L,D87G) parC (S80I) | soxR (E142K) |
| CR4 | – | gyrA (S83L,D87G) parC (S80I) | soxR (N154T) |
| CR5 | aac(6′)–Ib–cr | gyrA (S83L) parC (S80I) | soxR (E150G,E152D,Q153K, N154T) |
| CR6 | – | gyrA (S83L,D87G) parC (S80I) | soxR (L125R) |
| CR7 | – | gyrA (S83L,D87G) parC (S80I) | soxR (E142K) |
| CR8 | – | gyrA (S83L,D87G) parC (S80I) | soxR (N154T) |
| CR9 | – | gyrA (S83L) parC (S80I) | soxR (G140R,E142N) |
| CR10 | – | gyrA (S83L,D87N) parC (S80I) | – |
| CS1~10 | – | gyrA (S83L) | – |
注:"–".未发现突变;PMQR.质粒介导的喹诺酮类耐药基因;S.丝氨酸;L.亮氨酸;D.天冬氨酸;G.甘氨酸;I.异亮氨酸;C.半胱氨酸;R.精氨酸;E.谷氨酸;K.赖氨酸;N.天冬酰胺;T.苏氨酸;Q.谷氨酰胺;CR1~10.环丙沙星耐药株;CS1~10.环丙沙星敏感株
10株环丙沙星敏感株均未检测到外排泵基因及调控基因突变;而10株环丙沙星耐药株中,除CR10外,其余菌株均检出soxRS突变(表4):菌株CR1的soxRS第361位插入鸟嘌呤,其氨基酸序列122位由半胱氨酸变为亮氨酸,并在129位由天冬氨酸变为终止子,导致C末端26个氨基酸缺失;菌株CR2在418位由鸟嘌呤变为胞嘧啶,对应氨基酸序列140位由甘氨酸变为精氨酸;菌株CR3和CR7的soxRS 424位由鸟嘌呤变为腺嘌呤,对应氨基酸序列142位由谷氨酸变为赖氨酸;菌株CR4和CR8在457位缺失腺嘌呤,对应氨基酸序列154位由天冬酰胺变为苏氨酸;菌株CR5在449位缺失腺嘌呤,对应氨基酸150位由谷氨酸变为甘氨酸,152位由谷氨酸变为天冬氨酸,153位由谷胺酰胺变为赖氨酸,154位由天冬酰胺变为苏氨酸;菌株CR6的soxRS在375位由胸腺嘧啶变为鸟嘌呤,对应氨基酸序列125位由亮氨酸变为精氨酸;菌株CR9的419位由鸟嘌呤变为胞嘧啶,428位缺失鸟嘌呤,对应氨基酸序列140位甘氨酸变为精氨酸,142位谷氨酸变为天冬氨酸。
加入CCCP后,喹诺酮类药物对10株环丙沙星耐药株的MIC均下降至原值的1/4~1/8(表5),而对10株环丙沙星敏感株的MIC无变化或仅下降至原值的1/2(表6)。
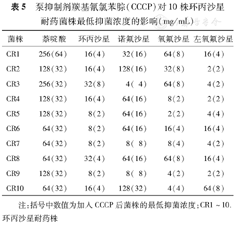
| 菌株 | 萘啶酸 | 环丙沙星 | 诺氟沙星 | 氧氟沙星 | 左氧氟沙星 |
|---|---|---|---|---|---|
| CR1 | 256(64) | 16(4) | 32(16) | 64(8) | 16(4) |
| CR2 | 128(32) | 16(4) | 128(16) | 32(8) | 2(2) |
| CR3 | 256(32) | 32(8) | 4(4) | 64(8) | 4(2) |
| CR4 | 128(32) | 16(4) | 64(16) | 8(2) | 2(2) |
| CR5 | 128(32) | 8(2) | 64(16) | 2(2) | 4(4) |
| CR6 | 64(32) | 8(2) | 64(16) | 16(4) | 16(4) |
| CR7 | 64(32) | 8(2) | 8(8) | 8(4) | 4(2) |
| CR8 | 64(32) | 32(4) | 64(16) | 64(8) | 16(4) |
| CR9 | 128(32) | 8(2) | 8(8) | 4(2) | 2(2) |
| CR10 | 64(32) | 16(4) | 128(32) | 4(4) | 64(8) |
注:括号中数值为加入CCCP后菌株的最低抑菌浓度;CR1~10.环丙沙星耐药株
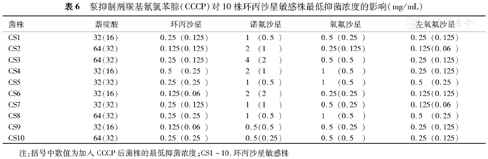
| 菌株 | 萘啶酸 | 环丙沙星 | 诺氟沙星 | 氧氟沙星 | 左氧氟沙星 |
|---|---|---|---|---|---|
| CS1 | 32(16) | 0.25 (0.125) | 1 (0.5) | 0.5 (0.25) | 0.25 (0.125) |
| CS2 | 64(32) | 0.125(0.125) | 2 (1 ) | 0.25(0.125) | 0.125(0.06) |
| CS3 | 64(32) | 0.25 (0.125) | 4 (2 ) | 0.5 (0.5 ) | 0.25 (0.125) |
| CS4 | 32(16) | 0.5 (0.25) | 2 (1 ) | 1 (0.5 ) | 0.25 (0.125) |
| CS5 | 32(32) | 0.25 (0.25) | 1 (0.5) | 1 (0.5 ) | 0.5 (0.25) |
| CS6 | 32(16) | 0.125(0.06) | 2 (2 ) | 0.25(0.25) | 0.125(0.125) |
| CS7 | 32(32) | 0.25 (0.125) | 1 (1 ) | 0.5 (0.25) | 0.125(0.06) |
| CS8 | 64(32) | 0.25 (0.25) | 1 (0.5) | 1 (0.5 ) | 0.5 (0.25) |
| CS9 | 32(16) | 0.125(0.06) | 0.5(0.5) | 0.5 (0.25) | 0.25 (0.125) |
| CS10 | 64(32) | 0.25 (0.25) | 0.5(0.25) | 0.5 (0.5 ) | 0.25 (0.125) |
注:括号中数值为加入CCCP后菌株的最低抑菌浓度;CS1~10.环丙沙星敏感株
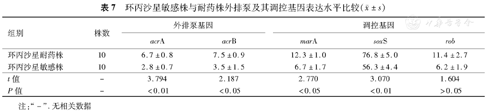
应用实时定量PCR方法检测耐药菌株外排泵基因acrA、acrB及调控基因marA、soxS、rob的相对基因表达量,结果见表7。与环丙沙星敏感株相比,环丙沙星耐药株rob基因的表达差异无统计学意义(P>0.05),其他泵基因及调控基因的表达在耐药株组中均有明显提高(均P<0.05)。
 ±s)
±s) ±s)
±s)| 组别 | 株数 | 外排泵基因 | 调控基因 | |||
|---|---|---|---|---|---|---|
| acrA | acrB | marA | soxS | rob | ||
| 环丙沙星耐药株 | 10 | 6.7±0.8 | 7.5±0.9 | 12.3±1.0 | 76.8±5.0 | 11.4±2.7 |
| 环丙沙星敏感株 | 10 | 2.8±0.7 | 3.5±1.5 | 6.7±1.7 | 56.3±4.4 | 6.2±1.9 |
| t值 | – | 3.794 | 2.187 | 2.770 | 3.070 | 1.604 |
| P值 | – | <0.01 | <0.05 | <0.05 | <0.01 | >0.05 |
注:"–".无相关数据
近年来,对氟喹诺酮类抗菌药物耐药的福氏志贺菌逐渐增多,其中对环丙沙星的耐药现象尤为突出。2013年中国CHINET耐药监测数据显示,福氏志贺菌对环丙沙星的耐药率达到45.9%[7]。本研究针对近5年天津地区收集的45株社区获得性福氏志贺菌临床株进行耐药性监测,结果显示本地区福氏志贺菌对环丙沙星的耐药现象不容乐观,其耐药率已达到44.4%,且耐药菌株均为多重耐药株,对第一代喹诺酮类药物萘啶酸完全耐药,并对其他氟喹诺酮类抗菌药物如诺氟沙星、左氧氟沙星等不同程度地产生耐药性。目前,对成人细菌性痢疾的治疗仍以氟喹诺酮类抗菌药物为主,这一耐药监测结果可用于指导本地区针对该类传染病的抗感染治疗方案的调整。
志贺菌对喹诺酮类抗菌药物的耐药机制主要包括靶位酶基因突变、PMQR基因以及外排泵活性增强等。有关靶位酶基因突变的相关研究表明,DNA解螺旋酶gyrA Ser83位点氨基酸改变可以使菌株表现出对第一代喹诺酮类抗菌药物萘啶酸的耐药,但菌株对环丙沙星等氟喹诺酮类抗菌药物产生耐药须同时存在gyrA和(或)parC多个位点突变或其他耐药机制[8]。本研究中10株对氟喹诺酮敏感的福氏志贺菌对照菌株对萘啶酸耐药,它们均出现gyrA Ser83–Leu的单个位点突变,对10株环丙沙星耐药的福氏志贺菌临床株的研究发现,所有菌株均存在gyrA83和parC80靶位酶基因突变,其中7株耐药菌还同时存在gyrA87位点突变,并出现不同的基因突变类型,再次证实靶位酶基因突变是以gyrA突变为基础的多位点突变,且突变位点具有多态性。PMQR检测显示仅有菌株CR2和CR5分别携带qnrS和aac(6′)–Ib–cr,而未发现其他qnr基因及qepA基因,提示PMQR基因并非导致耐药的主要机制。
在研究中我们发现,即使出现相同的靶位酶基因突变类型,耐药菌株对氟喹诺酮类抗菌药物的耐药表型并不完全相同,提示其他机制参与耐药的可能。自1998年Ghosh等[9]首次发现志贺菌中存在主动外排机制,并参与氟喹诺酮类抗菌药物的耐药性的研究以来,相继有研究证实这种耐药机制存在于志贺菌中[10,11]。外排泵基因的表达受其调控机制影响,主要包括局部调控和全局调控2种方式。局部调控蛋白AcrR是负性调控因子,由位于泵基因上游的acrR阻遏子基因编码,与acrAB启动子上的24个碱基对构成的反转重复序列结合,抑制自身及acrAB基因的表达。全局调控蛋白主要有MarA、Rob和SoxS等,属于AraC/XylS家族并构成marA–soxS–rob调控系统,结合于位于调节基因(如acrAB、tolC、marRAB)启动子上游的marbox序列,激活acrAB转录而提高外排泵表达。在肠杆菌科临床分离株中,此调控系统表达水平增高及调节基因acrR、marOR、rob和soxRS的基因突变可以导致acrAB操纵子的过表达,促进了细菌对氟喹诺酮类抗菌药物的耐药性[12,13,14],而此调控系统对于志贺菌的调节作用鲜见报道。本研究首先通过外排泵抑制实验初步证实了AcrAB–TolC外排泵对福氏志贺菌耐药性的影响,但是同时我们发现加入外排泵抑制剂CCCP后,耐药株的MIC并未降到敏感水平,提示志贺菌对氟喹诺酮类抗菌药物耐药仍以靶位酶基因突变为主,外排泵机制可能在其高水平耐药中起到重要作用[3]。实时定量PCR结果显示,与敏感株相比,耐药株出现了acrA、acrB的高表达,与此同时除了rob、mar及soxS基因的表达量明显增高,表明外排泵AcrAB–TolC的表达亦受此调控系统调节。为进一步探讨调控基因突变的特点,我们对10株耐药株进一步进行调控基因的序列分析,发现soxR出现了不同程度的基因突变,但并未发现acrR、marOR及rob的基因突变。SoxR是一种组成型表达的同二聚体转录调节子,它包括具有氧化还原活性的铁硫簇[2Fe–2S],这些铁–硫簇的氧化反应激活SoxR并触发了soxS基因的转录。soxS的组成型表达源于SoxR C末端的突变[15,16],但尚未发现有关志贺菌的相关研究。本试验中所检测的soxRS突变均集中于C末端,而这些突变是否导致外排泵高表达的原因尚需进一步实验研究证实。调控基因marOR未出现突变,但marA表达明显增高,表明调控因子之间存在交叉调节作用,这一作用也在其他菌株中得到证实[17]。
综上,本试验通过对福氏志贺菌环丙沙星耐药机制的系统研究,初步证实了外排泵及其调控机制在细菌耐药性中的作用。然而,临床耐药株的耐药机制复杂,同时外排泵及其调控因子的表达亦受多种因素影响,这种复杂的调控机制尚需进一步研究。
本研究得到天津医科大学总医院、天津市第一中心医院及天津市儿童医院感染科全体同事大力支持和协助,在此表示感谢。



























