
通过全基因组测序(Whole genome sequencing,WGS)分析碳青霉烯类耐药肺炎克雷伯菌(Carbapenem-resistant Klebsiella pneumonia,CRKP)的耐药基因携带情况。
收集2013年1月至2014年2月浙江中医药大学附属第二医院临床分离的30株CRKP,测定其对16种抗菌药物的敏感性,通过二代测序平台对所有菌株进行基因组测序并进行耐药基因筛选。
30株CRKP分离自8个不同临床科室,标本主要来源于痰液和尿液。药敏试验显示,30株CRKP对碳青霉烯类、头孢菌素类及β-内酰胺类/β-内酰胺酶抑制剂合剂均不敏感;对环丙沙星、庆大霉素、阿米卡星和复方磺胺甲噁唑的耐药率>70%;对多黏菌素E和替加环素的敏感率高,分别为100.0%和90.0%。经WGS检测共发现48种耐药基因。30株CRKP均携带blaKPC-2,29株(96.7%)同时携带多种超广谱β-内酰胺酶(Exterded spectrum β-lactamases,ESBLs)耐药基因;氨基糖苷类耐药基因以rmtB和aadA2为主;27株(90.0%)同时携带oqxA和oqxB喹诺酮类耐药基因;3种磷霉素耐药基因中,29株(96.7%)携带fosA,18株(60.0%)携带fosA3;23株(76.7%)CRKP携带sul1的磺胺类耐药基因;30株还同时携带不同型别的ESBLs、氨基糖苷类和磷霉素耐药基因。其中,28株(93.3%)还同时携带喹诺酮类耐药基因。所有菌株的耐药表型与耐药基因型在所测β-内酰胺类抗菌药物和多黏菌素完全符合,在氨基糖苷类和喹诺酮类抗菌药物中也存在较高的一致性。
CRKP同时携带多种抗菌药物耐药基因,呈现多重耐药表型。WGS在预测细菌耐药表型方面具有较高的应用前景。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
肺炎克雷伯菌是院内获得性感染中最为常见的致病菌之一,主要引起肺炎、败血症、尿路感染、肝脓肿及腹膜炎等。随着抗菌药物的广泛使用,尤其是碳青霉烯类抗生素的大量或不合理使用,碳青霉烯类耐药肺炎克雷伯菌(Carbapenem-resistant Klebsiella pneumonia,CRKP)呈现全球播散趋势,在我国分离率逐年攀升,已成为严重的公共卫生安全问题[1,2]。CRKP常呈现多重耐药表型,给临床治疗带来了极大的挑战。研究表明,CRKP的多重耐药表型与其同时携带多种耐药基因相关[3]。因此,该研究旨在通过全基因组测序(Whole genome sequencing,WGS)分析本院分离的CRKP的耐药基因型,了解其与耐药表型的相关性,为CRKP的临床治疗和院感防控提供科学的理论依据。
收集2013年1月至2014年2月于浙江中医药大学附属第二医院临床分离的CRKP 30株:21株来源于痰液标本,5株来源于尿液标本,2株来源于血液标本,各有1株分别来源于胃造口分泌物及盆腔引流物标本;分离自8个不同的临床科室,其中神经外科14株,重症监护室(Intensive care unit,ICU)9株,消化内科2株,呼吸内科、肛肠外科、血管外科、泌尿外科及骨科各1株。所有菌株均采用Vitek 2细菌全自动鉴定仪进行菌种及药敏鉴定。大肠埃希菌ATCC25922为药敏试验质控菌株。
LB肉汤培养基购自上海生工生物工程有限公司;Mueller-Hinton(MH)琼脂粉购自英国Oxoid公司;药敏纸片:包括美罗培南、亚胺培南、厄他培南、头孢吡肟、头孢噻肟、头孢他啶、头孢哌酮/舒巴坦、哌拉西林/他唑巴坦、氨苄西林/舒巴坦、氨苄西林、阿米卡星、庆大霉素、环丙沙星、复方磺胺甲噁唑共14种均购自Oxoid公司;替加环素和多黏菌素药敏试验粉剂购自法国梅里埃生物公司;细菌基因组DNA抽提试剂盒、产物纯化试剂盒为美国Axygen公司产品。细菌基因组测序仪为美国Illumina Hiseq 2000高通量测序仪。
采用纸片琼脂扩散法(K-B法)测定14种抗菌药物的敏感性,具体方法参照临床实验室标准化研究所(Clinical and laboratory standard institute,CLSI)2018版[4]。微量肉汤稀释法测定TGC和CST的敏感性:将待测菌株调成0.5个麦氏浊度,以1∶20稀释,为上样菌液,用MH肉汤稀释待测抗菌药物溶液,无菌96孔板的第1列加入200 μL加药的肉汤,第2~11列加入100 μL未加药的MH肉汤,从第1列吸取100 μL肉汤加入第2列,混匀后再吸取100 μL肉汤至第3列,依次进行倍比稀释,最后100 μL肉汤弃之;无菌96孔板的每孔加入待测菌液10 μL,药物及菌液上样完毕后,封好膜,置于37℃孵箱培养18~22 h观察结果。以大肠埃希菌ATCC25922和NCTC13846为质控菌株。药敏结果判断标准参照CLSI 2018版[4],其中,替加环素和多黏菌素则参照欧盟药敏试验标准(European Committee on Antimicrobial Susceptibility Testing,EUCAST)2018版[5]。
通过Illumina Hiseq 2000第二代高通量测序平台进行2×100 bp Paired-End测序,对所有菌株进行耐药基因的筛选:(1)基因组序列收到后,运用CLC Genomic Workbench 7.0软件对序列进行质量控制和拼接;(2)运用CGE网站(http://cge.cbs.dcu.dk/services/ResFinder/)的ResFinder 2.1工具,对拼接后的contigs进行耐药基因筛选,得出30株CRKP携带的所有耐药基因。
根据药物敏感试验的耐药表型与WGS检测筛查出的耐药基因型一致者,定义为两者符合;否则,均视为不符合。1.6数据分析计数资料以例(百分数)表示。
30株CRKP对美罗培南、厄他培南和亚胺培南的耐药率分别为100.0%、100.0%和96.7%;对氨苄西林、头孢吡肟、头孢噻肟、头孢他啶、头孢哌酮/舒巴坦、哌拉西林钠/他唑巴坦和氨苄西林/舒巴坦耐药率均为100.0%;对环丙沙星、庆大霉素、阿米卡星和复方磺胺甲噁唑的耐药率>70.0%;而对多黏菌素E和替加环素的敏感率高,分别为100.0%和90.0%(表1)。
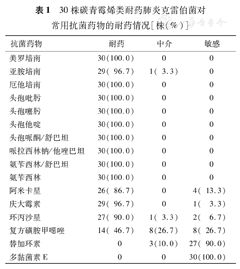
30株碳青霉烯类耐药肺炎克雷伯菌对常用抗菌药物的耐药情况[株(%)]
30株碳青霉烯类耐药肺炎克雷伯菌对常用抗菌药物的耐药情况[株(%)]
| 抗菌药物 | 耐药 | 中介 | 敏感 |
|---|---|---|---|
| 美罗培南 | 30(100.0) | 0 | 0 |
| 亚胺培南 | 29( 96.7) | 1( 3.3) | 0 |
| 厄他培南 | 30(100.0) | 0 | 0 |
| 头孢吡肟 | 30(100.0) | 0 | 0 |
| 头孢噻肟 | 30(100.0) | 0 | 0 |
| 头孢他啶 | 30(100.0) | 0 | 0 |
| 头孢哌酮/舒巴坦 | 30(100.0) | 0 | 0 |
| 哌拉西林钠/他唑巴坦 | 30(100.0) | 0 | 0 |
| 氨苄西林/舒巴坦 | 30(100.0) | 0 | 0 |
| 氨苄西林 | 30(100.0) | 0 | 0 |
| 阿米卡星 | 26( 86.7) | 0 | 4( 13.3) |
| 庆大霉素 | 29( 96.7) | 0 | 1( 3.3) |
| 环丙沙星 | 27( 90.0) | 1( 3.3) | 2( 6.7) |
| 复方磺胺甲噁唑 | 14( 46.7) | 8(26.7) | 8( 26.7) |
| 替加环素 | 0 | 3(10.0) | 27( 90.0) |
| 多黏菌素E | 0 | 0 | 30(100.0) |
30株CRKP经WGS检测,共发现48种耐药基因。10种β-内酰胺类耐药基因中,30株均携带blaKPC-2,29株(96.7%)同时携带超广谱β-内酰胺酶(Extended spectrum β-lactamases,ESBLs)耐药基因,以blaTEM-1B和blaSHV-11最多,各有23株(76.7%),15株(50.0%)携带blaCTX-M-65;11种氨基糖苷类耐药基因中,以rmtB(26株,86.7%)和aadA2(23株,76.7%)为主,其中22株(73.3%)同时携带两者;27株(90.0%)CRKP同时携带oqxA和oqxB两种喹诺酮类耐药基因;3种磷霉素耐药基因中,29株(96.7%)携带fosA,18株(60.0%)携带fosA3;磺胺类耐药基因有3种,主要基因sul1有23株(76.7%)细菌携带;氯霉素耐药基因共4种,catA2最多,有18株(60.0%)携带;另外在部分菌株中还检测到4种甲氧苄啶、大环内酯、林可胺和链霉素耐药基因,2种四环素类以及1种利福平耐药基因。30株CRKP除均携带blaKPC-2外,还同时携带不同型别的ESBLs耐药基因、氨基糖苷类耐药基因、磷霉素耐药基因。其中,28株(93.3%)还同时携带喹诺酮类耐药基因(表2)。
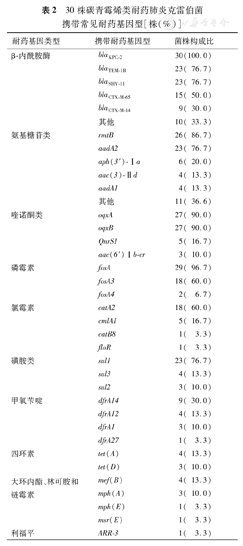
30株碳青霉烯类耐药肺炎克雷伯菌携带常见耐药基因型[株(%)]
30株碳青霉烯类耐药肺炎克雷伯菌携带常见耐药基因型[株(%)]
| 耐药基因类型 | 携带耐药基因型 | 菌株构成比 |
|---|---|---|
| β-内酰胺酶 | blaKPC-2 | 30(100.0) |
| blaTEM-1B | 23( 76.7) | |
| blaSHV-11 | 23( 76.7) | |
| blaCTX-M-65 | 15( 50.0) | |
| blaCTX-M-14 | 9( 30.0) | |
| 其他 | 10( 33.3) | |
| 氨基糖苷类 | rmtB | 26( 86.7) |
| aadA2 | 23( 76.7) | |
| aph(3′)-Ⅰa | 6( 20.0) | |
| aac(3)-Ⅱd | 4( 13.3) | |
| aadA1 | 4( 13.3) | |
| 其他 | 11( 36.6) | |
| 喹诺酮类 | oqxA | 27( 90.0) |
| oqxB | 27( 90.0) | |
| QnrS1 | 5( 16.7) | |
| aac(6′)Ⅰb-cr | 3( 10.0) | |
| 磷霉素 | fosA | 29( 96.7) |
| fosA3 | 18( 60.0) | |
| fosA4 | 2( 6.7) | |
| 氯霉素 | catA2 | 18( 60.0) |
| cmlA1 | 5( 16.7) | |
| catB8 | 1( 3.3) | |
| floR | 1( 3.3) | |
| 磺胺类 | sul1 | 23( 76.7) |
| sul3 | 4( 13.3) | |
| sul2 | 3( 10.0) | |
| 甲氧苄啶 | dfrA14 | 9( 30.0) |
| dfrA12 | 4( 13.3) | |
| dfrA1 | 3( 10.0) | |
| dfrA27 | 1( 3.3) | |
| 四环素 | tet(A) | 4( 13.3) |
| tet(D) | 3( 10.0) | |
| 大环内酯、林可胺和 | ||
| 链霉素 | mef(B) | 4( 13.3) |
| mph(A) | 3( 10.0) | |
| mph(E) | 1( 3.3) | |
| msr(E) | 1( 3.3) | |
| 利福平 | ARR-3 | 1( 3.3) |
30株CRKP的耐药表型与耐药基因型符合率结果:碳青霉烯类、头孢菌素、青霉素类、β-内酰胺类/β-内酰胺酶抑制剂合剂、多黏菌素的符合率为100.0%,氨基糖苷类为96.7%,喹诺酮类为93.3%,复方磺胺甲噁唑为56.7%。
肺炎克雷伯菌作为医院内获得性感染中的主要致病菌,常以多重耐药菌株为主,尤其对碳青霉烯类耐药形势令人堪忧。从2005至2017年,中国细菌耐药监测网(China antimicrobial surveillance network,CHINET)显示,肺炎克雷伯菌对亚胺培南和美罗培南的耐药率分别从3.0%和2.9%上升至20.9%和24.0%[2]。而这些菌株导致的感染常造成临床上无药可用。
细菌耐药性研究主要包括耐药表型和耐药基因型两方面。药敏试验是表型检测方法,但检测的抗菌药物种类有限,且有些细菌需要特殊培养环境;而基因型检测常用方法是聚合酶链反应(PCR),只能检测已知的部分耐药基因,往往不够全面。传统的检测方法虽然有助于了解细菌耐药情况,但常耗时耗力,不能深入揭示其遗传背景,各有不足之处。随着测序技术的进步,WGS已较成熟,并逐渐应用于临床[6]。据报道,WGS能准确预测金黄色葡萄球菌、大肠杆菌、肺炎克雷伯菌、沙门菌和结核分枝杆菌的耐药表型[7,8,9,10,11]。因此,基于WGS对CRKP的耐药性分析将有助于抗菌药物治疗的临床决策和医院感染的防控。
该研究结果显示,30株CRKP呈现多重耐药表型,对碳青霉烯类、三代头孢菌素、四代头孢菌素以及β-内酰胺类/β-内酰胺酶抑制剂合剂均不敏感,对喹诺酮类、氨基糖苷类表现出极高的耐药率,而对替加环素仅3株呈中介耐药,其余均敏感,多黏菌素则均敏感。基于WGS的耐药基因检测可见,30株CRKP均携带多种耐药基因,尤其是均携带blaKPC-2,可能是导致碳青霉烯类耐药的主要机制。同时,携带多种ESBLs耐药基因,已有研究报道,与头孢菌素耐药相关[12]。此外,CRKP还携带氨基糖苷类修饰酶基因、喹诺酮类耐药基因和16S rRNA甲基化酶基因,将导致临床氨基糖苷类和喹诺酮类治疗的失败,相关研究已证实[9,13]。磷霉素是目前多重耐药细菌治疗常用药物。据报道,产ESBLs大肠埃希菌对磷霉素的敏感率高达86%~100%[14]。Endimiani等[15]报道,包括对替加环素或多黏菌素不敏感的菌株在内的产KPC型酶肺炎克雷伯菌对磷霉素敏感率为87%~93%。该研究中,未进行磷霉素药敏试验检测,但从WGS检测结果中发现,30株CRKP均携带磷霉素耐药基因,主要是fosA和fosA3。可见,磷霉素在该类菌株感染治疗上可能均无效。国内有学者研究证实,携带blaKPC-2的肺炎克雷伯菌同时也携带fosA3耐药基因,而且可能是我国CRKP对磷霉素耐药的主要原因,本研究结果与其类似[16,17]。所有菌株对替加环素除3株表现中介耐药表型外,其余均敏感,检测到2种四环素类耐药基因型,均属于MFS型外排泵,但该型并不是导致替加环素耐药的主要机制。Mal系统、RND型外排泵和核糖体蛋白的基因突变则是替加环素主要耐药机制[18]。因此,3株替加环素不敏感菌株可能存在其他耐药机制。所有菌株未检测到多黏菌素耐药基因。结合多黏菌素耐药表型和耐药基因型可见,基于多黏菌素联合治疗是目前治疗CRKP的有效方案。
30株CRKP的耐药基因型与耐药表型在β-内酰胺类抗菌药物和多黏菌素完全符合,在氨基糖苷类和喹诺酮类抗菌药物中也存在较高的一致性。因此,通过菌株的耐药基因型预测其耐药表型,具有指导抗菌药物选择的潜力。但部分抗菌药物如替加环素、多黏菌素或喹诺酮类等,其耐药机制可由固有基因突变所致[19,20],而Resfinder却不能比对出突变造成的耐药基因。因此,将耐药细菌进行WGS,并通过Resfinder预测抗菌药物的药物敏感性还有一定的局限性。
综上所述,CRKP菌株同时携带多种耐药基因,呈现多重耐药表型,耐药形势十分严峻,应加强对其耐药监测,制定有效的医院感染控制措施,防止其播散流行。WGS在预测细菌耐药表型方面具有较高的应用前景,值得推广。
所有作者均声明不存在利益冲突



























