
通过回溯性研究院内的一次高毒力鲍曼不动杆菌感染暴发事件,寻找并鉴定引起此次事件的传染源及传播媒介。
收集深圳市人民医院2018年7至8月呼吸科普通病房和呼吸重症监护病房分离的9株耐碳青霉烯类鲍曼不动杆菌(Carbapenem-resistant Acinetobacter baumannii,CRAB),并采集病房环境物体表面和医务人员手部等环境标本。用Vitek 2对检出的CRAB进行菌种鉴定和药敏试验,用Illumina HiSeq 2500平台、Staramr、Ridom SeqSphere+进行全基因组测序、耐药基因检测及多位点序列分型,用IQ-Tree软件、BEAST2软件包和SCOTTI软件构建系统进化树和传播路径图。同时利用小鼠肺炎模型检测病原菌的毒力。
9株CRAB菌株均为ST2型,均携带耐碳青霉烯基因blaOXA-23,荚膜型分别为KL49、KL3、KL77和KL2。1株环境分离(可移动呼吸机屏幕)CRAB菌株为ST2型及KL49型荚膜。系统进化树分析显示环境分离株与其中5株CRAB位于同一分支,传播路径图显示这5株CRAB也位于同一条传播链。5株CRAB涉及4个致死病例,小鼠体内实验验证了引起致死感染的菌株具有高毒力表型。
高毒力鲍曼不动杆菌可在移动医疗设备表面定植,共享这些设备可能导致医院内病原菌传播。利用新型传播路径分析工具对传播事件的调查有一定的参考意义。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
目前,鲍曼不动杆菌是世界范围内与医院获得性感染相关的重要微生物之一[1]。在重症监护病房(Intensive care units,ICU),鲍曼不动杆菌占感染病原菌的20%,通常表现为呼吸机相关性肺炎和中央导管相关血流感染[2,3]。由于鲍曼不动杆菌在枕头、窗帘和其他干燥物体表面等环境中可长期定植,导致该病原菌在院内患者之间的传播很难控制,阻断其传播须遵守严格的感染防控措施[4]。至今已有许多医院报道过鲍曼不动杆菌感染的暴发事件[5]。
根据之前的研究报道,鲍曼不动杆菌一直被认为是一种低致病力的病原菌。大多数实验室标准菌株和临床分离株并不会在免疫功能完善的小鼠身上引起严重感染,即使接种大剂量细菌,也只能引发自限性肺炎,其局部复制和全身播散的能力均非常有限[6,7]。然而,有研究表明,一些临床耐碳青霉烯类鲍曼不动杆菌(Carbapenem-resistant Acinetobacter baumannii,CRAB)对具有免疫能力的小鼠具有致命性,从而揭示了该病原菌不同菌株之间的毒力差异[8,9]。更为严重的是,已有报道证实这些具有高毒力的CRAB可引起医院感染暴发,而这种暴发往往是致死性的[10]。本文报道一起由高毒力CRAB引起的院内感染暴发事件,经回溯性研究分析感染源和传播途径,最终有效阻断菌株的进一步传播。
回顾性分析深圳市人民医院2018年7至8月连续性报道的9例CRAB感染患者的临床资料。CRAB是指对亚胺培南或美罗培南等碳青霉烯类抗生素耐药的鲍曼不动杆菌[11]。该研究经深圳市人民医院伦理委员会审核通过(伦理批号:LL-KY-2022134)。
Vitek-MS、Vitek 2和配套的N335药敏卡为法国梅里埃公司产品;药敏纸片丁胺卡那和MH琼脂购自英国Oxoid公司;替加环素药敏检测试剂盒购自杭州康泰生物技术有限公司;细菌基因组DNA提取试剂盒购自天根生化科技有限公司,DNA Marker D、4S Red plus核酸染色剂、琼脂糖H、TAE缓冲液、上样缓冲液均购自生工生物工程(上海)股份有限公司;琼脂糖凝胶电泳仪、凝胶成像系统购自美国Bio-Rad公司;超微量分光光度计购自英国Biochrom公司;测序由生工生物工程(上海)股份有限公司完成。
采用全自动细菌鉴定药敏仪Vitek 2和配套的N335药敏卡进行菌株的鉴定和抗菌药物敏感性试验,结果参照2018年美国临床和实验室标准化协会(CLSI)M100-S28文件[12]标准判读。采用纸片琼脂扩散法(K-B法)测定丁胺卡那的敏感性,具体方法参照2018年CLSI M100-S28文件[12]。采用微量肉汤稀释法测定替加环素的最低抑菌浓度(MIC)值[13]。质控菌株为大肠埃希菌ATCC25922和铜绿假单胞菌ATCC27853。
对病房环境(包括高频接触的物体表面、医护人员手)进行微生物采样。按照《医院消毒卫生标准》[14]要求,使用浸有PBS的无菌棉拭子。物体表面采样方法:规则或平整表面使用棉拭子采集面积5 cm × 5 cm的区域,连续采4个区域,不规则或不平整的物体表面使用无菌棉拭子直接涂抹采样;医务人员手卫生采样方法:使用棉拭子在双手指曲面从指跟到指端来回涂抹两次,将采样好的棉拭子放置于含有中和剂的试管内保存并转运。
用十二烷基硫酸钠(SDS)法提取菌株的基因组DNA并用Qubit(Thermo Fisher,Waltham,MA,USA)进行DNA定量。构建DNA文库,在长度为350 bp的DNA片段两端特异添加index、接头和测序引物,然后上机测序,测序仪采用Illumina HiSeq 2500(Illumina,San Diego,CA,USA)。通过fastp对测序数据进行质量控制、fastq预处理、切除接头和poly G/poly X尾并筛除低质量(phred值<Q15)、低复杂度(30%)的reads[15]。使用de novo SPAdes(version 3.12.0)对处理后测序数据进行拼接[16]。接着用Staramr(version 0.2.2)对ResFinder数据库进行基因组比对,检测菌株的耐药基因[17],使用Prokka(version 1.13.3)对基因组进行注释[18],使用Ridom SeqSphere+(version 5.1.0)进行基于巴斯德分型标准的多位点序列分型(MLST)[19]。
在获得全基因组测序数据的基础上,使用IQ-Tree软件通过最大似然法构建系统进化树;使用Harvest suite(version 1.2)[20]进行核心基因组的比对及单核苷酸多态性位点(Single nucleotide polymorphism,SNPs)的提取;使用BEAST2(version 2.5.1)软件包和Structured Coalescent Transmission Tree Inference (SCOTTI)(version 1.1.1)[21]整合核心基因组SNPs及流行病学数据(出入院时间、采样时间)构建传播路径图。
小鼠肺部感染模型的建立:自湖南SJA实验动物有限公司购买SPF级6~8周的C57BL/6龄雌性小鼠,腹腔注射戊巴比妥钠(75 mg/kg)麻醉后,在直视下于气管内无创灌注鲍曼不动杆菌菌液20 μL(2.5×108和5×107个菌落形成单位(Colony-forming unit,CFU),每组10只,感染后7 d内观察小鼠存活情况,以存活率判断毒力。本研究中所有动物的护理和使用方案均按照中华人民共和国国务院批准的《实验动物管理条例》执行。
将9例患者编号为1~9。9例患者均为男性,年龄为65.9(47~84)岁。2018年7月检出1例,8月检出8例。其中呼吸科普通病房(Respiratory medicine unit,RMU)2例,呼吸重症监护病房(Respiratory intensive critical care unit,RICU)7例。共检出9株非重复性CRAB,其中8株来源于痰液,1株来源于血液标本。4例患者(病例2、4、7、9)死于CRAB感染(表1)。
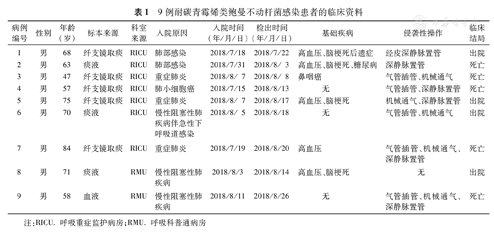
9例耐碳青霉烯类鲍曼不动杆菌感染患者的临床资料
9例耐碳青霉烯类鲍曼不动杆菌感染患者的临床资料
| 病例编号 | 性别 | 年龄(岁) | 标本来源 | 科室来源 | 入院原因 | 入院时间(年/月/日) | 检出时间(年/月/日) | 基础疾病 | 侵袭性操作 | 临床结局 |
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 男 | 68 | 纤支镜取痰 | RICU | 肺部感染 | 2018/7/18 | 2018/7/22 | 高血压、脑梗死后遗症 | 经皮深静脉置管 | 出院 |
| 2 | 男 | 63 | 痰液 | RICU | 肺部感染 | 2018/7/31 | 2018/8/ 3 | 高血压、脑梗死、糖尿病 | 深静脉置管 | 死亡 |
| 3 | 男 | 47 | 纤支镜取痰 | RICU | 重症肺炎 | 2018/8/ 7 | 2018/8/ 8 | 鼻咽癌 | 气管插管、机械通气 | 死亡 |
| 4 | 男 | 57 | 纤支镜取痰 | RICU | 肺小细胞癌 | 2018/7/15 | 2018/8/13 | 无 | 气管插管、深静脉置管 | 死亡 |
| 5 | 男 | 75 | 纤支镜取痰 | RICU | 重症肺炎 | 2018/8/ 7 | 2018/8/17 | 高血压、脑梗死 | 机械通气、深静脉置管 | 出院 |
| 6 | 男 | 70 | 痰液 | RICU | 慢性阻塞性肺疾病伴急性下呼吸道感染 | 2018/8/ 5 | 2018/8/18 | 无 | 气管插管、机械通气 | 出院 |
| 7 | 男 | 84 | 纤支镜取痰 | RICU | 重症肺炎 | 2018/7/19 | 2018/8/20 | 高血压 | 气管插管、机械通气、深静脉置管 | 死亡 |
| 8 | 男 | 71 | 痰液 | RMU | 慢性阻塞性肺疾病 | 2018/8/3 | 2018/8/14 | 高血压、脑梗死 | 无 | 出院 |
| 9 | 男 | 58 | 血液 | RMU | 慢性阻塞性肺疾病 | 2018/8/11 | 2018/8/26 | 无 | 气管插管、机械通气、深静脉置管 | 死亡 |
注:RICU.呼吸重症监护病房;RMU.呼吸科普通病房
初步怀疑为CRAB医院暴发感染。9月1日,对RMU和RICU的医师办公室及护士站、治疗室和病房环境物体表面及医务人员手进行采样,环境物体表面包括键盘鼠标、电话、冰箱、药盒、血压袖带、治疗车、隔帘、排痰机、心电监护仪、床头柜及听诊器等,共采标本30份,均未检出CRAB。
9株CRAB对美罗培南、亚胺培南均耐药;对头孢菌素类药物及加酶抑制剂均不敏感;对喹诺酮类药物左氧氟沙星和环丙沙星均耐药;对氨基糖苷类药物丁胺卡那、妥布霉素均耐药;2株对磺胺类药物复方磺胺甲噁唑敏感,7株耐药;3株对米诺环素中介,6株耐药;4株对替加环素敏感,1株中介,4株耐药;对多黏菌素均敏感(表2)。
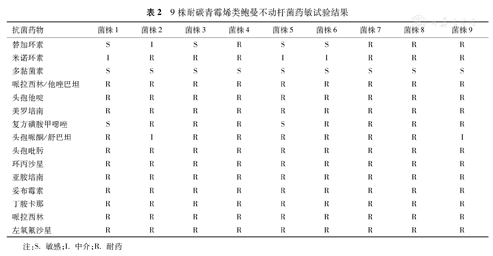
9株耐碳青霉烯类鲍曼不动杆菌药敏试验结果
9株耐碳青霉烯类鲍曼不动杆菌药敏试验结果
| 抗菌药物 | 菌株1 | 菌株2 | 菌株3 | 菌株4 | 菌株5 | 菌株6 | 菌株7 | 菌株8 | 菌株9 |
|---|---|---|---|---|---|---|---|---|---|
| 替加环素 | S | I | S | R | S | S | R | R | R |
| 米诺环素 | I | R | R | R | I | I | R | R | R |
| 多黏菌素 | S | S | S | S | S | S | S | S | S |
| 哌拉西林/他唑巴坦 | R | R | R | R | R | R | R | R | R |
| 头孢他啶 | R | R | R | R | R | R | R | R | R |
| 美罗培南 | R | R | R | R | R | R | R | R | R |
| 复方磺胺甲噁唑 | S | R | R | R | S | R | R | R | R |
| 头孢哌酮/舒巴坦 | R | I | R | R | R | R | R | R | I |
| 头孢吡肟 | R | R | R | R | R | R | R | R | R |
| 环丙沙星 | R | R | R | R | R | R | R | R | R |
| 亚胺培南 | R | R | R | R | R | R | R | R | R |
| 妥布霉素 | R | R | R | R | R | R | R | R | R |
| 丁胺卡那 | R | R | R | R | R | R | R | R | R |
| 哌拉西林 | R | R | R | R | R | R | R | R | R |
| 左氧氟沙星 | R | R | R | R | R | R | R | R | R |
注:S.敏感;I.中介;R.耐药
所有菌株均为ST2型,blaOXA-23是本研究中唯一检测到的碳青霉烯酶基因。1株荚膜型为KL3,5株为KL49,2株为KL77,1株为KL2(图1)。
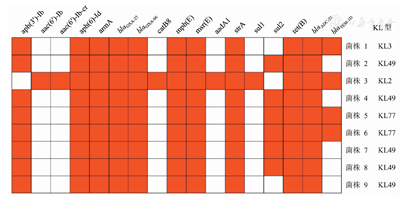

9月30日,对共享的可移动设备进行了第二次重点集中采样,包括床边纤维支气管镜、可移动呼吸机、运输担架、床边胸片机、移动呼吸机等进行了物体表面采样。培养结果显示在移动呼吸机的屏幕上分离出1株CRAB。对10株CRAB构建系统进化树后发现,环境分离株与5株CRAB位于同一分支,3株CRAB位于同一分支,另1株处于单独分支(图2)。
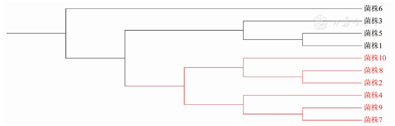

注:菌株1~9来源于患者,菌株10来源于移动呼吸机屏幕
如图3所示,共有2条传播链被鉴定出来。值得注意的是,所有死亡病例(病例2、4、7和9)均位于同一传播链中。病例2于7月31日至8月12日住在RICU,病例4于7月30日由RMU转入RICU,在病例4被转入RICU后4 d,病例8住进RMU同一床位,病例7于7月24日转入RICU,病例9于8月11至26日住在RMU,病例5于8月7至22日住在RICU,病例6于8月5至28日住在RICU。病例2和7、病例4和7、病例2和4、病例5和6在RICU住院期间存在空间交互,而病例7和8,病例7和9之间则没有空间交互。病例2被认为是传播源,病例4、8、7和9分别在病例2后接连感染,病例7的感染来源则有两种可能(病例4或8)。
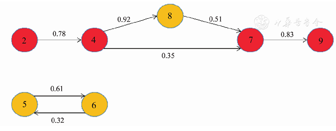

注:1~9指耐碳青霉烯类鲍曼不动杆菌感染患者,箭头上数字代表对应的传播概率,仅展示概率>0.20的传播链
基因组分析表明,参与死亡病例传播链和可移动呼吸机分离出的菌株与高毒力菌株LAC-4具有相同的荚膜型(KL49),因此选取病例2、病例6和可移动呼吸机分离株在小鼠肺部感染模型中进行了毒力检测,并使用LAC-4作为高毒力阳性对照。使用2.5×108 CFU的菌液感染小鼠,病例2、呼吸机分离株和LAC-4的7 d生存率分别为10%、10%和0,而病例6和ATCC17978的生存率则分别为70%和90%(n=10,P<0.01)(图4)。
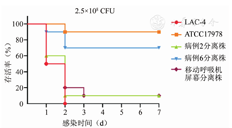

注:CFU.菌落形成单位
鲍曼不动杆菌是一种院内条件性致病菌,常引起ICU内败血症、肺炎和感染暴发事件的发生[22]。本研究从可移动医疗设备上分离得到1株CRAB,表明该病原体已成功在医院内传播和定植。
对于感染暴发的研究,最好的方式是溯源在两个宿主间是否发生了直接传播。随着全基因组测序技术的进步和生物信息学分析方法的发展,人们在结合流行病学和全基因组数据来推断暴发传播路径方面取得了一定进展[21],使得精准判断宿主之间的传播路径变得可行[21,23,24,25,26,27]。
在本研究中,启动调查的第一时间(2018年9月1日)对环境物体表面进行了无差别采样,未检出相关菌株。经全基因组测序及生物信息学分析,得到了菌株的同源性关系及传播路径图。鉴于传播路径图提示病例7和8之间存在传播,而流行病学资料显示二者分属于不同病区,没有空间上的交互联系,因此推断经由医务工作者传播的可能性较小。经调查得知,RMU和RICU位于医院的同一栋楼,共用一台床边胸片机和纤维支气管镜室。病例资料显示,所有病例感染前均拍过床边胸片,病例4、7感染前接受过纤维支气管镜检查。因此认为纤维支气管镜室内的空间交互和床边胸片机的共享可能是病例病原菌传播的原因。
基于以上发现,2018年9月30日,再次重点对多科室共享的所有移动医疗设备,包括床边纤维支气管镜、可移动呼吸机、运输担架、床边胸片机等进行了物体表面采样。从可移动呼吸机的屏幕上分离出了CRAB。全基因组测序分析后发现,呼吸机屏幕分离出的CRAB为ST2型,与RICU及RMU检出的9例菌株ST型一致;荚膜型为KL49,与病例2、4、7、8、9的荚膜型一致;系统发育树上也发现环境分离菌与病例2、4、7、8、9属于同一分支,因此推断该菌株与上述病例所在的传播链可能存在关联。
通过调查结果,推测本次感染暴发的传播源和传播途径:病例2为传播源,传播途径可能是患者的痰液污染了可移动呼吸机的屏幕,未彻底消毒的可移动呼吸机在RMU与RICU交互使用,导致在RICU患者中传播。2018年11月1日,病房在指定的密闭房间中使用过氧化氢蒸汽对所有可移动设备进行消毒[28],同时指定专人负责定期(每月)对可移动设备进行消毒。此外,ICU的移动设备不再与普通病房共用。回顾性数据显示,经过可移动设备的定期消毒管理后,2019年1至12月,RICU住院患者分离的鲍曼不动杆菌中未发现有KL49型CRAB被检出,环境物体表面样本中未检出CRAB,表明该克隆株已被成功清除。本研究使用了一种类似于结构聚类的推理工具来重建CRAB暴发事件中的传播路径。结果显示,此次暴发由若干不同的传播链组成,其中存在无空间交集的宿主间的传播链。值得重点关注的是,调查发现了可移动医疗设备在鲍曼不动杆菌传播中作为媒介的作用。一株新发的高毒力鲍曼不动杆菌在可移动医疗设备表面的成功定植,可能会导致普通病房和ICU之间在共享这些可移动设备时发生病原菌的直接传播。另外,细菌在移动设备表面的定植,增加了病原体向外部环境播散的概率以及通过医务人员引起患者之间间接传播的风险。在特殊的密闭空间内采用过氧化氢蒸汽是对可移动医疗设备消毒的有效工具,避免普通病房和ICU共享设备及定期安排人员对可移动设备进行消毒是预防此类传播的有效途径。
然而,由于本研究属回顾性调查,对曾经开展的医疗操作难以完全呈现,存在一定的局限性。由于病房内患者感染的因素较多,涉及的环境也非常多样复杂,因此环境卫生学检测的范围也可能存在疏漏,从而导致本次调查只是发现了线索,并未证实传染的源头,也未能完整还原传播的链条。
总而言之,本研究利用传播路径推断工具结合流行病学资料对一起鲍曼不动杆菌引起的院内暴发进行了分析。分析结果表明,一株高毒力菌株在院内定植和播散,可移动设备的使用不当可导致普通病房和ICU之间病原菌的传播。因此,需要定期对可移动设备进行消毒,并安排人员进行管理。此外,利用全基因组测序结合流行病学数据对于分析确定没有空间交互的患者之间的传播路径是行之有效的。
所有作者均声明不存在利益冲突



























