
随着海量多维度生物医学数据的积累,包括基因组、代谢组、脂质组、微生物组、电子医疗病历、医学影像和可穿戴设备数据以及包括深度学习在内的多模态人工智能(AI)模型算法取得的突破,使得对糖尿病精准分型和精准治疗的探索成为可能。通过对应用传统AI算法在2型糖尿病分型上取得的进展进行回顾,借鉴多模态AI模型在其他精准医学领域所得的成功经验,展望2型糖尿病精准分型中应用多模态AI模型整合多维度数据的前景。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
2型糖尿病(type 2 diabetes melitus,T2DM)作为一种在全球范围内普遍存在且日益严重的健康问题,给人们的经济和生活都带来了极重的压力与负担。多数临床医师认为T2DM是由多种因素引起的,在生理病理上属于典型的复杂疾病并且在人群中具有高度的异质性。但是,这种异质性在实际临床预防管理和治疗中并没有被充分考虑,而是对于每个T2DM患者都施以标准化的治疗方案。这种缺乏个性化的治疗方法无法使每例患者都得到有效的干预治疗效果,导致了医疗费用的浪费,甚至贻误患者病情。所以,更准确认识T2DM病理机制的多样性,实现临床上的进一步分型,是达成T2DM精准治疗的关键。目前研究人员对T2DM患者的特征描述已经不局限于简单的临床表型定义,而是尝试纳入多组学(基因、代谢、肠道菌群等)、高精度(时序)以及多结构形态(图片、文本等)的数据类型,综合这些多维度特征才能更好地描绘T2DM的全貌[1]。由于这些描述涉及海量数据及复杂数据类型,越来越多的研究人员在T2DM的研究中利用了AI算法。AI算法区别于传统计算机技术的本质是让机器学会按照人类基于经验的思维方式来处理问题,而非按照预先编程好的代码来处理问题。这需要基于海量样本数据进行特征提取和模式识别,从而对其他的样本做出有效预测。常见特征值包括文本特征、数值特征和图像特征等,AI算法的优势体现在两方面,一方面AI算法能够发现人类难以量化的特征的规律(如图像特征),另一方面,AI算法能够高效处理海量数据,并发现其中的复杂关联。
T2DM的异质性是由不同的致病分子机制造成的,并伴有不同的疾病进展和并发症。越来越多来自流行病学和基因组学的证据引发了研究人员对T2DM疾病异质性的广泛讨论与研究。近年来,流行病学的结果揭示了几个显著的疾病人群亚组,包括体型极端肥胖与极度消瘦、不同种族的遗传差异、胰岛素抵抗和胰岛素分泌不足。Færch等[2]基于常见的3种临床诊断指标组合将T2DM患者分为3类,即只根据空腹血糖浓度诊断、只根据2 h葡萄糖浓度诊断以及同时根据两种浓度进行诊断的患者,结果表明这3个亚组分别表现出不同的心脏代谢病风险。大规模全基因组关联研究(genome-wide association studies,GWAS)结果显示,虽然目前已经发现超过200个遗传变异,但每增加1个风险等位基因,其后果只是在影响糖尿病风险上表现出微妙的增加。通过相关的研究,现在可以把这些遗传变异与特殊的细胞或组织功能联系起来,而不仅仅是基于它们对T2DM这一疾病风险的影响考虑。例如,HNF1A和WFS1可影响胰岛的发育;CDKN2A会影响胰岛衰老;F14、PPARG作用于脂肪生成,而FTO作为肥胖的主要致病突变可导致胰岛素抗性的生成;KCNJ11影响胰岛功能;KLF14是脂肪组织中的基因转录调控因子,影响脂肪细胞分布和大小,这很可能是女性群体区别于男性群体腹部脂肪存储倾向的糖尿病风险差异所在[3, 4]。另外,最近对T2DM基因组和其并发症的一项研究结果也证明,根据不同单核苷酸多态性功能进行分组的遗传风险评分与糖尿病肾病和冠状动脉疾病等并发症风险之间存在关联[5]。Delahanty等[6]针对糖耐量受损的肥胖人群进行研究,比较了他们个体之间对于不同干预方式的响应程度,即研究在基因(肥胖相关的单核苷酸多态性)与环境(干预手段)的相互作用下对体重的影响,结果显示,在携带相同基因的人群中,以服用二甲双胍和改变生活方式为主要干预手段的两组人体重减少得较多,其中后者体重减少得最多,表明即使是在携带相同基因型的人群中,对于常规糖尿病治疗干预手段(药物、运动)的反应也存在不一致性。对于肠道菌群的研究结果也显现出T2DM患者在肠道菌群上的显著异质性,有着较高肠道菌群多样性和较高促产丁酸盐菌丰度的T2DM患者在血糖管理的改善上比对照组效果更显著[7]。由此可见,T2DM亚型在不同组学和流行病学观测上呈现出明显可分性,可能是受到多个平行和(或)相互作用机制以及环境暴露的影响,从而造成患者在疾病的进展和并发症类型上表现出显著的差异。近年来,随着科学科技的快速发展,已有一些利用AI模型对T2DM异质性成功解构并分型的研究结果(图1)。
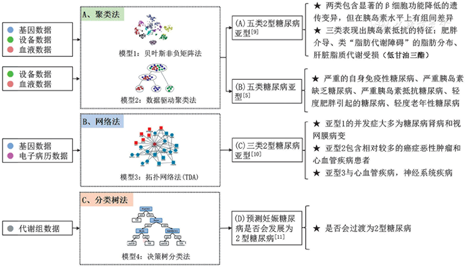

Li等[8]利用电子病历和基因型数据构建了基于拓扑的网络算法来计算T2DM患者间的相似性与差异性,该模型将T2DM分为3个亚型,其中亚型1的并发症大多为糖尿病肾病和视网膜病变,亚型2包含相对较多的癌症恶性肿瘤和心血管疾病患者,亚型3与心血管疾病、神经系统疾病、过敏和人类免疫缺陷病毒感染最为相关。另外,贝叶斯聚类算法更进一步地解构了T2DM异质性,研究者根据94个独立的T2DM基因座和47个糖尿病相关性状将T2DM分为5个大类:其中两类包含导致β细胞功能降低的遗传变异,但在胰岛素水平上有组间差异;另外3类表现出胰岛素抵抗的不同特征,肥胖介导型表现为较高体质指数、较大腰围,类“脂肪代谢障碍”型表现为低体质指数、低脂联素、低高密度脂蛋白和高甘油三酯,肝脏脂质代谢受损型表现为低甘油三酯[9]。
目前也有研究者尝试打破对常见糖尿病的传统分类,即1型糖尿病、T2DM特殊类型糖尿病和妊娠糖尿病。Ahlqvist等[5]使用聚类的算法对在成年期诊断为糖尿病的患者(包括1型糖尿病、T2DM和成年潜伏性自身免疫性糖尿病)进行了数据驱动的分型尝试,根据临床检测指标谷氨酸脱羧酶抗体、被诊断年龄、BMI、糖化血红蛋白、稳态模型评估2-β细胞功能指数以及稳态模型评估2-胰岛素抵抗指数进行聚类分析,确定了5个并在独立数据集中被验证的糖尿病患者亚群,这5个亚型包括严重自身免疫糖尿病、严重胰岛素分泌障碍糖尿病、严重胰岛素抵抗糖尿病、轻度肥胖相关糖尿病和轻度年龄相关糖尿病,其中每个亚型的患者各具有显著不同的临床特征和糖尿病并发症风险。对胰岛素最具抗性的亚组人群比其他亚组个体具有更显著的糖尿病肾病风险,而胰岛素分泌缺乏的亚组人群具有最高的视网膜病变风险,同时,各亚组人群在遗传风险上也表现出显著的组间差异[5]。而后,研究人员又把这个基于北欧人群的糖尿病分型方法(聚类法)应用在了中国和美国等独立人群数据上,并使用了除谷氨酸脱羧酶抗体外的其余5个变量将成年糖尿病聚为4个亚组,分别为轻度与年龄相关的糖尿病、轻度肥胖相关的糖尿病、严重的胰岛素缺乏型糖尿病和胰岛素抵抗型糖尿病,但这类新的分层方法只能对成年新发糖尿病提供分型,并不能对长期的并发症做出有效的分组[10]。糖尿病的临床表现是随疾病的进程而呈动态变化的,这会导致亚型诊断的转变,例如20%~50%的妊娠性糖尿病在5年内会发展为T2DM,而使用最新的代谢组学结合决策树算法鉴定21种小分子代谢物,可以提前并精准预测转变发病的情况从而提早干预,效果远远优于传统的临床空腹血糖筛查。这也更进一步证明了在妊娠性糖尿病人群中对于是否会最终转变成T2DM存在着较大的个体差异性,而借助代谢组学构建的模型能更加精准的抓取到这一异质性[11]。
以上对于糖尿病再分型的研究或是基于单一的数据类型,或是仅利用了少数经过专家先验知识挑选出来的重要生物标志物来构建的传统AI模型(表1)。这些计算模型只能为我们勾勒出简略的阶段性分型结果,并不能对糖尿病的致病机制给出更清晰的解释和与之相应的精准干预治疗方法,也没有办法为同一亚型患者在疾病进展的不同阶段做持续动态数据的建模,导致低效的干预治疗效果和大量医疗成本的浪费。目前,已有大量研究者开始尝试整合多模态多组学数据建模来提高利用现有丰富数据(从遗传变异到最终临床表型)理解疾病信息流的能力。复杂疾病的发病涉及多种危险因素,从多维分子扰动到不同细胞功能,以及组织和器官的病理变化。关注任何特定类型的数据只能提供对疾病的有限见解,这是因为影响糖尿病发展的生物过程不是一个个独立的基因或代谢物,而是以一种跨不同组学的分子级联反应和相互作用机制实现的。因此,不仅要关注传统临床表型,包括发病年龄、BMI、代谢紊乱、胰岛素、谷氨酸脱羧酶抗体、糖化血红蛋白、稳态模型评估2-β细胞功能指数和稳态模型评估2-胰岛素抵抗指数,同时更重视致病分子机制,将病因、遗传学背景、疾病进展、并发症发生情况、药物治疗反应等综合纳入分型体系才是未来糖尿病分型难题的解决之道。因此,提出一种能够整合基因组学、蛋白组学、代谢组学和临床特征及生活习惯的多模态AI辅助分型策略,希望从海量的数据中准确找到有意义的致病位点,并对其进行功能学研究,同时根据时序数据构建动态分型模型以适应不同病程的发展变化,从而使其能有效地应用于临床。事实上,McCarthy[12]在2017年提出了糖尿病的“调色板”模型,该模型强调不仅要关注分子分类学的分型,还要注重个体的主要病理生理过程和进展(如肥胖、脂肪分布、胰岛发育和功能、胰岛素敏感性)从而全面理解这一复杂疾病。

人工智能在2型糖尿病及成年发病糖尿病分型上使用的特征
人工智能在2型糖尿病及成年发病糖尿病分型上使用的特征
| 特征类别 | 特征值 |
|---|---|
| 基本信息 | 年龄、诊断年龄、性别 |
| 人体测量 | 体质指数、身高、体重、腰围、臀围、腰臀比、出生时体重、出生时身高、体脂率 |
| 生命体征 | 心率、脉搏、收缩压、舒张压、呼吸、体温 |
| 基因组 | rs2296172、rs17106184、rs10923931、rs340874、rs2820443、rs780094、rs10203174、rs243021、rs6742799 、rs13389219、rs2943641、rs1801282、rs1496653、rs2276853、rs6795735、rs11708067、rs4402960、rs6808574、rs4458523、rs6813195、rs1996546、rs702634、rs459193、rs4457053、rs9502570、rs7756992、rs3132524、rs9271775、rs4407733、rs2191349、rs864745、rs3996352、rs1182397、rs10503669、rs516946、rs7845219、rs3802177、rs10758593、rs10811661、rs17791513、rs2796441、rs505922、rs11257655、rs4746890、rs12571751、rs1111875、rs7903146、rs2421016、rs2334499、rs10840595 、rs2237892、rs5215、rs3736505、rs11227234、rs1552224、rs10830961、rs11063069、rs10842994、rs2261181、rs7961581、rs7957197、rs1106240、rs1359790、rs10146997、rs4502156、rs7178572、rs2866367、rs1371135、rs12899811、rs9939609、rs9921586、rs2925979、rs8068804、rs17676067、rs4430796、rs9891146、rs15563、rs9948462、rs12970134、rs16996148、rs8108269、rs4812829、rs17265513、rs36608、rs738409、rs3843467、rs10276674、rs2184061、rs10757282、rs231361、rs233449、rs384037、rs10219509、rs1800961 |
| 临床指标 | 丙氨酸转氨酶、天冬氨酸转氨酶、胆红素、血清肌酸酐、表皮生长因子受体、白蛋白、血红蛋白、总胆固醇、胆固醇/高密度脂蛋白、低密度脂蛋白、高密度脂蛋白、低密度脂蛋白/高密度脂蛋白、甘油三酯、尿素氮、血细胞压积、红细胞、白血球、嗜红细胞计数、嗜红细胞数百分比、嗜碱细胞计数、嗜碱细胞数百分比、淋巴细胞计数、淋巴细胞数百分比、单核细胞计数、单核细胞数百分比、中性白细胞计数、中性白细胞数百分比、血小板计数、钾、钠、钙、N3脂肪酸、N6脂肪酸、长链饱和脂肪酸、血浆磷脂脂肪酸、脂联素、尿酸盐、瘦素、谷氨酸脱羧酶抗体 |
| 血糖检测 | 空腹胰岛素、空腹血糖、餐后2 h血糖、空腹胰岛素原、胰岛素敏感性指数、糖化血红蛋白 |
相较于传统的聚类分析,由于多模态的AI模型能有效地整合来自不同组学不同空间的数据,目前已经被成功应用在癌症的临床精准医疗方案上。通过整合TCGA乳腺癌数据集中的基因表达、microRNA和DNA甲基化特征,研究人员计算了患者间的相似度(通过将不同维度的数据进行子空间合并),从而成功地发现了一组与乳腺癌患者生存率密切相关的重要功能性基因簇,并将乳腺癌在临床上进一步分为5种亚型,亚型1为三阴型乳腺癌;亚型3和亚型4均为明显的黄体酮阳性和雌激素阳性,以及不明显的表皮生长因子阴性,但有显著不同的预后;亚型2和亚型5均为不明显的黄体酮阳性和雌激素阳性,以及明显的表皮生长因子阴性,但有显著不同的预后[13]。另一组研究人员开发了一种基于卷积神经网络的深度学习的算法,将来自组织学图像和基因组的信息整合到一个统一的模型中,这两种异构数据的整合提供了单一数据无法理解的视角,并通过可视化模型的中间结果对致病机制提出新的见解,如微血管增殖对疾病发展的影响,进而对癌症的精准药物研发和精准治疗提供研究基础[14]。这些成功的研究结果突出了整合多模态数据的AI深度学习模型在处理复杂疾病、精准分型、实现精准治疗上的卓越能力。
另外,多模态AI模型在挖掘海量数据上具有一定潜力。研究者利用基于梯度增强回归的深度学习模型整合了基本的身体测量、标准化食物摄入、运动量、睡眠、血清代谢物、血糖检测和微生物组,并采用数据驱动的无偏方法成功地对任意的实验参与者个性化地预测了实时血糖水平,效果显著优于基于人群的均一化的血糖预测效果,为未来精准营养的应用探明了可行性[15]。还有学者利用相关性网络构建社群的算法,整合了基因组学和3个时间点的代谢组、蛋白质组、微生物组以及日常活动跟踪等的多模态时序多组学数据,提高了对健康状态和疾病状态的理解,特别是从健康到疾病的早期过渡状态,并尝试根据参与者的临床生物标志物状况提供个性化的行为指导,帮助参与者在疾病未发生的状态下提前改善健康状况[16]。
目前笔者尚未见利用多模态AI对T2DM进行分型的研究,大部分仍旧停留在使用传统的AI模型整合单模态数据。但是,随着近年来基因组、蛋白组、代谢组、个人体征、生活形态、临床指标等多维度数据的爆发式增长,加之多模态多组学生物银行数据的开放(UKBiobank、T2D Knowledge Portal等),都为多模态AI在T2DM的精准分型奠定了基础。精准分型在T2DM临床上的潜在应用场景可能是可以根据对个体特有的发病机制的解读从而制定有针对性的个性化治疗方案,可能的方案包括但并不仅限于:主要致病因素是胰岛功能则要注重磺酰脲类药物的治疗;对于那些以肥胖为诱因的T2DM则以体重减轻为主要干预手段;有显著胰岛素抵抗现象的患者可以通过运动来干预;能量分配机制出现显著障碍的则要以噻唑烷二酮类化合物为主要治疗药物;肠道微生物失调为病因的则可以用相应的微生物补充手段来干预治疗等。这些精准治疗方案都将显著提高医师对患者疾病进程的控制。
我们距离真正实现糖尿病的精准治疗还有很长的路要走。目前,AI模型主要被应用于对是否会从糖尿病前期转化为糖尿病的风险预测,而且,在模型中也仅纳入了一些相对晚期的风险因素,如BMI、血压、空腹血糖等。笔者在此倡议加大糖尿病多模态AI模型的研究力度,同时注重临床和随访数据的积累。具体来说,通过时序追踪一些糖尿病早期风险因素,如饮食记录、运动记录、代谢谱特征变化、肠道菌群变化、体脂等生理参数变化等,可以构建个人糖尿病多模态AI模型。该模型不仅可以用于病前预警、早期筛查诊断,也可以用于糖尿病精准分型和制定个性化干预治疗。基于模型的一些内部特征,如网络拓扑结构、参数空间分布和激活模式等,还有助于进一步加深人们对糖尿病这一复杂疾病发病机制的理解和认识。
在现有慢病管理分级诊疗制度的建设之外,结合国家倡导的“互联网+医疗健康”发展意见,我们提出了一个围绕糖尿病健康管理而建立的个人信息与服务生态平台,以更好地支撑个人糖尿病多模态AI模型的构建与应用(图2)。该平台的成功搭建,将进一步践行“主动健康”发展策略,助力“健康中国2030”美好规划的顺利实现。

所有作者均声明不存在利益冲突
1.在构建多模态AI模型过程中不需要的一项是:
A.融合处理不同形态结构的数据
B.持续地收集数据
C.广泛收集多种类型的数据
D.为患者提供的健康建议
2.传统AI模型在T2DM的应用上取得的成就不包括:
A.精准分型 B.2型糖尿病异质性分析
C.个性化持续动态分析 D.提早干预治疗
3.不属于多模态AI模型较传统模型在T2DM精准分型上的优势是:
A.提高分型精度 B.对疾病机制提供清晰的解释
C.进行疾病进展持续动态的分析
D.发现致病临床表现
4. 以下不属于传统机器学习算法的是:
A.k-mean算法 B.卷积神经网络
C.决策树算法 D.支持向量机算法
5. 为患者建立糖尿病多模态AI模型的目的不包括:
A.病前预警 B.早期筛查诊断
C.制定个性化干预治疗方案
D.收集患者的饮食运动信息



























