
基于MRI的常规T2加权成像(T2-weighted imaging,T2WI)序列,比较采用不同机器学习算法所建立的影像组学模型在卵巢上皮性肿瘤术前三分类中的诊断效能。
回顾性分析300例(良性、交界性和恶性各100例)经病理证实为卵巢上皮性肿瘤患者的术前磁共振图像,按8∶2随机划分训练集和测试集。从轴位T2WI图像上手动勾画的三维感兴趣区域中提取图像特征,并进行特征筛选。将4种特征选择方法和7种机器学习分类器两两组合,构建28个分类模型。采用曲线下面积(AUC)和准确度对所有模型的预测性能进行评估。
28种分类模型中表现最好的是递归特征消除法(recursive feature elimination,RFE)与K近邻(K nearest neighbor,KNN)分类器相结合的“RFE-KNN”模型,其测试集上良性组的AUC为0.94,交界性组的AUC为0.93,恶性组的AUC为0.96。
从常规T2WI序列中提取的定量影像组学特征所建立的RFE-KNN模型在卵巢上皮性肿瘤的术前三分类中具有良好的表现。

本刊刊出的所有论文不代表本刊编委会的观点,除非特别声明
卵巢上皮性肿瘤是卵巢肿瘤中最常见的类型,可根据其是否具有异常增殖分化与侵袭性分为良性、交界性和恶性三种类型[1],患者的治疗及预后方案都与肿瘤类型有关[2, 3, 4, 5, 6],因此,三者的术前鉴别对患者的治疗和预后具有重要意义。MRI因其无创、软组织的分辨率高的优点,对卵巢肿瘤组织病理学类型的鉴别有很大的帮助,目前已被广泛应用于卵巢肿瘤的鉴别诊断和预后评估中[7, 8, 9, 10],有报道称MRI在良、恶性卵巢上皮性肿瘤鉴别中的准确度可高达90%[11],但也有研究表明,在常规MRI上交界性与恶性卵巢上皮性肿瘤有许多相似的形态学特征,这使得鉴别诊断变得困难[12]。此外,传统的影像诊断大多依赖于影像医生主观判断,个人经验在其中起着较为重要的作用,主观影响较大[13]。
影像组学不但可以通过分析病变形状和纹理特征从而识别肉眼无法观测到的反映肿瘤特异性的影像学特征[14, 15, 16, 17],且相较于传统的影像学评估方法具有更高的诊断效能和可重复性[18, 19, 20]。目前已有研究将影像组学方法应用于卵巢肿瘤类型的鉴别诊断上,但以良性与恶性、交界性与恶性的二分类鉴别研究居多[21, 22, 23, 24, 25]。Song等[26]进行了良性、交界性和恶性卵巢肿瘤三分类的鉴别研究,但仅采用了一种机器学习算法用于建模,且病例数较少(82例)。本研究在更多数据量(300例)的基础上,采用多种机器学习分类器与多种特征筛选方法来建立影像组学分类模型,对影像组学方法在良性、交界性和恶性卵巢上皮性肿瘤鉴别问题上的可行性做进一步探索。此外,大量研究表明,不同的影像组学模型会对疾病的诊断效能产生一定的影响[27, 28, 29, 30],因此本研究拟基于MRI常规T2WI序列,评估不同影像组学模型在卵巢上皮性肿瘤术前三分类鉴别中的诊断效能。
回顾性分析重庆医科大学附属第一医院2013年1月至2020年12月术前行常规MRI检查的300例卵巢上皮性肿瘤患者,年龄17~83 (47.95±14.02)岁。其中良性、交界性和恶性卵巢上皮性肿瘤患者各100例。
纳入标准:术后经组织病理检查确诊为卵巢上皮性肿瘤的患者;既往无盆腔手术史和放化疗史;术前有包含完整卵巢肿瘤的MRI图像。
排除标准:卵巢上皮性肿瘤复发患者;伴有妊娠患者。本研究经过重庆医科大学附属第一医院伦理委员会批准(批准文号:2021-338),免除受试者知情同意。
使用GE Signa HDxt 1.5 T和3.0 T MR扫描仪,所有患者取仰卧位,采用8通道腹部线圈进行扫描,图像扫描参数见表1。MR扫描图像通过影像归档和通信系统以DICOM格式导出,并抹除患者信息。
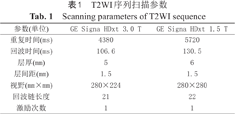
T2WI序列扫描参数
Scanning parameters of T2WI sequence
T2WI序列扫描参数
Scanning parameters of T2WI sequence
| 参数(单位) | GE Signa HDxt 3.0 T | GE Signa HDxt 1.5 T |
|---|---|---|
| 重复时间(ms) | 4380 | 5720 |
| 回波时间(ms) | 106.6 | 130.5 |
| 层厚(mm) | 5 | 6 |
| 层间距(mm) | 1.5 | 1.5 |
| 视野(mm×mm) | 280×224 | 280×280 |
| 回波链长度 | 21 | 22 |
| 激励次数 | 1 | 1 |
首先对原图像进行预处理,包括重采样、标准化和N4偏置场校正(N4 Bias Field Correction),以减少图像间的偏差,其中重采样使用双线性插值法,以保证图像体素大小的均一性和各向同性;标准化采用的是Z-score Normalization,以加快模型训练的收敛速度。再对图像进行N4偏置场校正,以解决磁共振图像采集过程中因设备抖动或患者身体的轻微活动造成的磁场分布不均匀问题。将T2WI序列上的整个肿瘤区域作为ROI,由两名具有6年以上阅片经验的影像诊断医师采用双盲法在ITK-SNAP (3.8版本)上沿肿瘤边缘手动逐层勾画,获取三维感兴趣区域(volume of interest,VOI),对有争议的病例,两人协商后达成共识。
使用python 3.7.6的影像组学包(PyRadiomics 3.0)提取图像特征,共提取7个影像组学特征簇的1288个特征,包括14个形状特征,252个直方图特征,308个灰度共生矩阵(gray level co-occurence matrix,GLCM)特征,224个灰度游程矩阵(gray level run length matrix,GLRLM)特征,224个灰度区域大小矩阵(gray level size zone matrix,GLSZM)特征,70个邻域灰度差矩阵(neighbouring gray tone difference matrix,NGTDM)特征和196个灰度相关矩阵(gray level dependence matrix,GLDM)特征。
将以上经特征提取所获得的全部原始数据进行分组标记,良性组记为“0”,交界性组记为“1”,恶性组记为“2”,采用标准化对数据集进行预处理,并对缺失值进行填充,对异常值进行平衡。以最小绝对收缩选择算子(least absolute shrinkage and selection operator,LASSO)、递归特征消除法(recursive feature elimination,RFE)、单变量特征选择法(univariate feature selection,UFS)和互信息特征选择法(mutual information,MI)为特征筛选方法,各从1288个特征中筛选出与分类相关性最高的前10个特征。
选用7种有监督机器学习分类算法:逻辑回归(logistic regression,LR)、支持向量机(support vector machine,SVM)、随机森林(random forest,RF)、K近邻(K nearest neighbor,KNN)、决策树(decision tree,DT)、高斯朴素贝叶斯(GaussianNB,GNB)和Adaboost集成分类器(Adaptive Boosting,AB)。将上述7种机器学习算法所对应的分类器与1.3.3中的4种特征选择方法两两组合,共建立28 (4×7=28)个分类预测模型,并以“特征选择方法-机器学习分类器”的模式对其进行命名,如结合RFE特征选择法和KNN分类器所建立的模型,则命名为“RFE-KNN”。将所有病例数按8∶2.随机划分为训练集和测试集,利用3次10折交叉验证对模型进行训练,并在测试集上进行验证,获取测试集的ROC曲线和分类混淆矩阵。
采用AUC和准确度对各模型的分类预测效能进行综合评价。
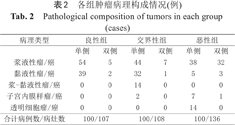
各组肿瘤病理构成情况(例)
Pathological composition of tumors in each group (cases)
各组肿瘤病理构成情况(例)
Pathological composition of tumors in each group (cases)
| 病理类型 | 良性组 | 交界性组 | 恶性组 | |||
|---|---|---|---|---|---|---|
| 单侧 | 双侧 | 单侧 | 双侧 | 单侧 | 双侧 | |
| 浆液性瘤/癌 | 54 | 5 | 44 | 7 | 38 | 32 |
| 黏液性瘤/癌 | 39 | 2 | 32 | 1 | 5 | 3 |
| 浆-黏液性瘤/癌 | 0 | 0 | 14 | 0 | 0 | 0 |
| 子宫内膜样瘤/癌 | 0 | 0 | 2 | 0 | 7 | 1 |
| 透明细胞瘤/癌 | 0 | 0 | 0 | 0 | 14 | 0 |
| 合计病例数/病灶数 | 100/107 | 100/108 | 100/136 | |||
图1为测试集上各分类模型的宏平均AUC和准确度热图,图中横坐标代表7种不同的机器学习分类器,纵坐标代表4种不同的特征选择方法,颜色越深代表数值越大。图1A显示的是28个分类模型在测试集上的宏平均AUC,AUC评分范围为0.79到0.94。其中AUC大于等于0.90的分类模型有6个,按AUC从高到低排序分别是RFE-KNN、UFS-KNN、RFE-SVM、RFE-RF、RFE-DT和LASSO-KNN,它们的AUC分别为0.94、0.92、0.91、0.91、0.90和0.90。图1B显示的是28个分类模型在测试集上的准确度,准确度评分范围为0.63到0.83,其中准确度大于0.80的分类模型有3个,按准确度从高到低分别是RFE-KNN、RFE-DT和UFS-KNN,准确度分别为0.83、0.80和0.80。结合各分类模型在测试集上的AUC和准确度,可以看出RFE-KNN模型的分类性能优于其他模型。RFE-KNN模型在测试集上的宏平均AUC和准确度分别为0.94和0.83。
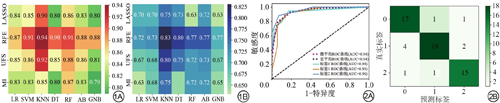

注:LASSO:最小绝对收缩选择算子;RFE:递归特征消除法;UFS:单变量特征选择法;MI:互信息特征选择法;LR:逻辑回归分类器;DT:决策树分类器;RF:随机森林分类器;AB:Adaboost集成分类器;SVM:支持向量机分类器;KNN:K近邻分类器;GNB:高斯朴素贝叶斯分类器。
表3为测试集上4种特征选择方法中的每一种方法单独与7种分类器相结合所构建的7个分类模型的AUC和准确度均值,以比较单一特征选择方法在本数据集上的分类预测性能。可见,4种特征选择方法中表现最优的为RFE,AUC均值和准确度均值分别为0.899和0.760。
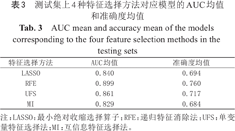
测试集上4种特征选择方法对应模型的AUC均值和准确度均值
AUC mean and accuracy mean of the models corresponding to the four feature selection methods in the testing sets
测试集上4种特征选择方法对应模型的AUC均值和准确度均值
AUC mean and accuracy mean of the models corresponding to the four feature selection methods in the testing sets
| 特征选择方法 | AUC均值 | 准确度均值 |
|---|---|---|
| LASSO | 0.840 | 0.694 |
| RFE | 0.899 | 0.760 |
| UFS | 0.861 | 0.717 |
| MI | 0.829 | 0.684 |
注:LASSO:最小绝对收缩选择算子;RFE:递归特征消除法;UFS:单变量特征选择法;MI:互信息特征选择法。
表5为7种机器学习分类器中的每一种分类器单独与4种特征选择方法相结合所构建的4个分类模型的AUC均值和准确度均值,以比较单一机器学习分类器在本数据集上的分类预测性能。可见,7种机器学习分类器中表现最优的为KNN,AUC均值和准确度均值分别为0.903和0.783。
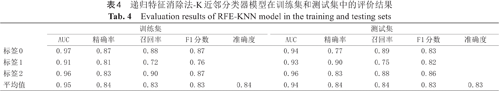
递归特征消除法-K近邻分类器模型在训练集和测试集中的评价结果
Evaluation results of RFE-KNN model in the training and testing sets
递归特征消除法-K近邻分类器模型在训练集和测试集中的评价结果
Evaluation results of RFE-KNN model in the training and testing sets
| 训练集 | 测试集 | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| AUC | 精确率 | 召回率 | F1分数 | 准确度 | AUC | 精确率 | 召回率 | F1分数 | 准确度 | ||
| 标签0 | 0.97 | 0.87 | 0.88 | 0.87 | 0.94 | 0.77 | 0.89 | 0.83 | |||
| 标签1 | 0.91 | 0.81 | 0.72 | 0.76 | 0.93 | 0.90 | 0.75 | 0.82 | |||
| 标签2 | 0.96 | 0.83 | 0.90 | 0.87 | 0.96 | 0.83 | 0.88 | 0.86 | |||
| 平均值 | 0.95 | 0.84 | 0.83 | 0.83 | 0.84 | 0.94 | 0.84 | 0.84 | 0.83 | 0.83 |

测试集上7种机器学习分类器对应模型的AUC均值和准确度均值
AUC mean and accuracy mean of the corresponding models of the seven machine learning classifiers in the testing sets
测试集上7种机器学习分类器对应模型的AUC均值和准确度均值
AUC mean and accuracy mean of the corresponding models of the seven machine learning classifiers in the testing sets
| 分类器 | AUC均值 | 准确度均值 |
|---|---|---|
| LR | 0.848 | 0.680 |
| DT | 0.838 | 0.733 |
| RF | 0.880 | 0.710 |
| AB | 0.850 | 0.735 |
| SVM | 0.863 | 0.695 |
| KNN | 0.903 | 0.783 |
| GNB | 0.820 | 0.663 |
注:LR:逻辑回归分类器;DT:决策树分类器;RF:随机森林分类器;AB:Adaboost集成分类器;SVM:支持向量机分类器;KNN:K近邻分类器;GNB:高斯朴素贝叶斯分类器。
表4为RFE-KNN模型在训练集和测试集上各项评价指标的得分情况,图中标签0代表良性肿瘤,标签1代表交界性肿瘤,标签2代表恶性肿瘤。训练集上良性组、交界性组和恶性组的AUC分别为0.97、0.91和0.96,测试集上良性组、交界性组和恶性组的AUC分别为0.94、0.93和0.96。
图2是RFE-KNN模型在测试集上的ROC曲线和混淆矩阵,图中标签0代表良性肿瘤,标签1代表交界性肿瘤,标签2代表恶性肿瘤。图2A是RFE-KNN模型在测试集上的ROC曲线图,图中展示了良性组、交界性组和恶性组的ROC曲线,及AUC分别为0.94、0.93和0.96,此外三组的宏平均AUC和微平均AUC,均为0.94。图2B是RFE-KNN模型在测试集上的分类混淆矩阵,横坐标代表预测类别,纵坐标代表真实类别,由混淆矩阵我们可以看出良性组、交界性组和恶性组发生误判的百分比分别为10.5% (2/19);25.0% (6/24),11.8% (2/17)。通过分析RFE-KNN模型在测试集上的ROC曲线和混淆矩阵,可以看出发生误判情况最多的是交界性组。
本研究针对卵巢上皮性肿瘤三分类问题,建立和验证了一种基于MRI常规T2WI序列的影像组学模型,用于鉴别良性、交界性和恶性卵巢上皮性肿瘤。在我们的研究中,28个分类模型对卵巢上皮性肿瘤类型的预测性能各不相同,AUC范围从0.79到0.94不等,整体预测性能表现良好,证明了影像组学模型在卵巢肿瘤三分类研究中具有一定的可行性,同时也验证了不同影像组学模型对同一疾病的诊断效能是有影响的。从我们的结果来看,所采用的特征选择方法中表现最好的是RFE。在既往的文献中,RFE在疾病分类和预后预测上的有效性已得到了广泛认可[31, 32, 33],在不同特征选择方法的比较方面,Wang等[34]认为,在肺部良恶性病变鉴别诊断中,RFE的表现优于t检验和LASSO,基于我们的数据集,我们发现RFE特征选择方法在卵巢上皮性肿瘤三分类中的表现优于LASSO、UFS和MI。同时,我们所选用的分类器中表现最好的是KNN,它是机器学习中最简单最常见的分类器之一,该分类器的核心主要是度量测试集和训练集样本之间的距离或相似性,因为有着良好的适应性,KNN被广泛用于大数据的分类中[35, 36, 37, 38]。在我们的实验中,KNN的表现优于其他6种分类器,证明了其在卵巢上皮性肿瘤术前三分类中的有效性和优越性。
基于我们的数据集,以RFE为特征筛选方法,KNN为分类器的RFE-KNN模型在卵巢上皮性肿瘤三分类中表现最好。Song等[26]曾采用影像组学方法来鉴别良性、交界性和恶性卵巢肿瘤,他们的研究共纳入82个病例,104个病灶,建立了一种基于动态对比增强磁共振成像(dynamic contrast-enhanced magnetic resonance imaging,DCE-MRI)的药物代谢动力学模型,他们的测试结果显示,良性组、交界性组和恶性组的AUC值分别为0.893、0.944和0.891。与他们的研究不同的是,我们的研究基于MRI常规序列T2WI,共纳入300个病例,351个病灶,采用4种特征选择方法和7种机器学习分类器,两两组合建立了28个影像组学分类模型,比较了各模型的分类预测性能,其中预测性能最好的是RFE-KNN模型。由图2A RFE-KNN模型在测试集上的ROC曲线图可见,在我们的模型中良性组(标签0)、交界性组(标签1)和恶性组(标签2)的AUC值分别为0.94、0.93和0.96,整体略优于Song等的研究。
根据我们的研究结果,交界性组发生误判的比例高于良性组和恶性组,这与临床经验是一致的。以往的研究表明,与良恶性肿瘤相比,交界性肿瘤在影像学诊断中最易发生误诊,这与肿瘤的影像表现有直接的联系,由于卵巢肿瘤的影像表现通常是重叠和非特异性的,且交界性肿瘤在影像上的宏观特征可能与良性和恶性卵巢肿瘤相似或重叠[39, 40],导致交界性肿瘤在传统影像学诊断中不易鉴别。我们的组学模型也有误判的情况,但总体来说发生误判的比例在可接受范围内,相较于传统影像学诊断,影像组学在卵巢肿瘤诊断方面仍是一个更加客观、可重复性更高的诊断方法。
我们的研究有以下几个局限,首先这是一个回顾性研究,在图像数据选择方面由于时间跨度大难免出现偏差,虽然相对以往的研究在数据量上有所提升,但总体数据量依然比较小。其次,我们的数据来源是单中心的,往后的研究还需要多中心数据来验证该方法的普适性。在未来,基于大数据的前瞻性、多中心、多序列研究将会有更广阔的应用前景。
综上所述,我们所建立的影像组学模型在良性、交界性和恶性卵巢上皮性肿瘤鉴别诊断上具有可行性。基于我们的数据集,RFE-KNN模型在卵巢上皮性肿瘤三分类鉴别诊断上表现最好,可为临床诊断卵巢上皮性肿瘤类型提供决策支持。
全部作者均声明无利益冲突。



























