
探讨使用MRI影像组学的方法,建立一个用于预测较低级别胶质瘤(lower grade gliomas, LGGs)异柠檬酸脱氢酶突变(isocitrate dehydrogenase mutation, IDH mut)合并氧6-甲基鸟嘌呤DNA甲基转移酶启动子甲基化(O6-methylguanine-DNA methyltransferase promoter methylation, MGMT meth)亚型的影像组学模型。
回顾性分析来自山西医科大学第一医院、山西省人民医院和TCGA/TCIA(The Cancer Genome Atlas和The Cancer Imaging Archive)公共数据库的158例患者的术前MRI图像、临床和基因信息。将上述三处数据合并,对所有图像进行重采样和强度归一化处理后,以7∶3的比例随机分为训练集和测试集。从术前MRI图像上提取对比增强后T1加权序列(post-contrast enhanced T1-weighted, CE-T1)和T2加权流体衰减反转恢复序列(T2-weighted fluid attenuation inversion recovery, T2-FLAIR)的1702个影像组学特征。通过单因素逻辑回归(logistic regression, LR)、最小绝对收缩和选择算法(least absolute shrinkage and selection operator, LASSO)进行有效特征筛选,使用合成少数过采样技术(synthetic minority over-sampling technique, SMOTE)对训练集少数类样本进行数据平衡。最后,使用多因素LR进行建模。通过受试者工作特征(receiver operating characteristic, ROC)曲线和校准曲线评价模型诊断效能和拟合优度,并建立列线图进行可视化风险预测。
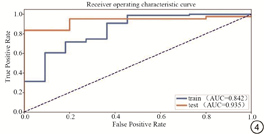
在训练集和测试集两个亚型的不同临床特征的组间比较差异无统计学意义(P>0.05)。影像组学模型在训练集和测试集的AUC(area under the curve, AUC)分别为0.842和0.935,F-Measure分别为0.965和0.942,训练集校准曲线经Hosmer-Lemeshow检验的P值为0.1393。
术前MRI影像组学模型可以预测LGGs患者IDH mut合并MGMT meth亚型,从而为LGGs患者的分子分型的精确诊断、替莫唑胺(temozolomide, TMZ)使用的决策、生存期预测提供重要的临床辅助价值。

本刊刊出的所有论文不代表本刊编委会的观点,除非特别声明
较低级别胶质瘤(lower grade gliomas, LGGs)通常指世界卫生组织(World Health Organization, WHO)分级为2、3级的胶质瘤。其中,约占全部胶质母细胞瘤8.8%~10%的继发型是由LGGs进展而来[1, 2, 3]。LGGs的精准诊断、个体化治疗和疗效预后评估均与肿瘤分子分型密切相关[2]。LGGs的异柠檬酸脱氢酶突变(isocitrate dehydrogenase mutation, IDH mut)合并氧6-甲基鸟嘌呤DNA甲基转移酶启动子甲基化(O6-methylguanine-DNA methyltransferase promoter methylation, MGMT meth)亚型的患者,在异柠檬酸脱氢酶(isocitrate dehydrogenase, IDH)、氧6-甲基鸟嘌呤DNA甲基转移酶(O6-methylguanine-DNA methyltransferase, MGMT)的所有分子亚型中具有更长的总生存期(overall survival, OS)和(或)无进展生存期(progression free surviva, PFS)[4, 5],并从替莫唑胺(temozolomide, TMZ)的治疗中获益[6]。因此,该分子分型的准确预测具有重要的临床价值。
目前,胶质瘤IDH和MGMT分子标记物获得需采用侵入性的、有创的病理活组织检查或手术切除方式得以实现,而基于常规MRI检查的影像组学方法,相比前者,具有无创性、及时性和价格合理等优点[7, 8]。MRI检查可以无创地获取活体内深埋于组织或器官内部肿瘤的静态结构、动态功能的多维度和多参数的图像,利用计算机图像处理和大数据挖掘的影像组学方法,发掘肉眼无法观察到的纹理、小波等高维度影像特征,从而获得深层次的映射肿瘤分子标记物的影像组学标记物[9, 10, 11, 12]。
然而,应用影像组学的方法对脑胶质瘤分子生物标记物的研究多集中在IDH、MGMT等单一分子上[13, 14]。在多个肿瘤分子标记物结合作为联合预测因子的研究中,也是根据2016年WHO中枢神经系统肿瘤的分子分型,集中在IDH合并1p/19q基因型的研究中[15, 16]。目前,基于常规MRI的影像组学方法在胶质瘤的IDH合并MGMT状态的分子亚型的研究却很少涉及。Zhang等[17]使用自动机器学习的放射组学方法,对胶质瘤(WHO 2~4级)IDH mut和MGMT meth共同发生进行预测研究。这项研究的病例中,WHO 4级胶质瘤占所有被纳入研究的胶质瘤的51.8%,且该研究中作为对照组的亚型包含更广。因此,该研究的影像组学模型的分类性能更侧重于WHO 4级的胶质瘤。而本研究的对象为LGGs,即WHO 2、3级胶质瘤,且定义的两个分子亚型更具针对性。由于LGGs的IDH mut合并MGMT meth作为联合的预测因子,预示着良好的OS和(或)PFS,并能从TMZ治疗中获益。因此,本研究探讨使用基于MRI的影像组学的方法,建立一个用于预测LGGs的IDH mut合并MGMT meth亚型的预测模型,从而为脑胶质瘤的精确诊断、TMZ的临床决策及生存期预测提供重要的辅助价值。
本研究回顾性分析来自山西医科大学第一医院(The First Hospital of Shanxi Medical University, FHSXMU)、山西省人民医院(Shanxi Provincial People' Hospital, SPPH),以及TCGA/TCIA(The Cancer Genome Atlas和The Cancer Imaging Archive)公共数据库(TCGA中包含了胶质瘤患者的临床和基因信息,网址为:https://www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga,TCIA中为与TCGA对应的患者影像数据,网址为:https://www.cancerimagingarchive.net)的447例患者数据。FHSXMU和SPPH数据收集通过山西医科大学伦理委员会批准,免除受试者知情同意,批准文号:2019LL101,收集日期为2011年10月至2020年7月。TCGA/TCIA数据为公用数据库资源,数据上传和下载已获得华盛顿大学医学院伦理委员会批准,免除受试者知情同意,批准文号:201108194,TCGA/TCIA数据收集日期为2011年3月至2019年10月。本研究的纳入标准:(1)符合2021新版WHO中枢神经系统肿瘤分级为2、3级的胶质瘤患者;(2)术前MRI图像包括对比增强后T1加权序列(post-contrast enhanced T1-weighted, CE-T1)、T2加权流体衰减反转恢复序列(T2-weighted fluid attenuation inversion recovery, T2-FLAIR),并且序列完整、图像清晰;(3)完整的IDH和MGMT分子信息及临床信息。最终,158例患者符合纳入标准,其中TCGA/TCIA数据集86例,FHSXMU和SPPH数据集72例。
FHSXMU数据采用8通道阵列线圈的3.0 T Scanner(Signa HDxt, GE Healthcare, USA)进行MRI扫描,SPPH数据采用8通道阵列线圈的3.0 T Scanner(Discovery 750, GE Healthcare, USA)进行MRI扫描。采集协议统一采用CE-T1序列(TR/TE:195 ms/4.76 ms;FOV:240 mm×240 mm;层厚/层间距:5.0 mm/1.5 mm;矩阵:256×256)和T2-FLAIR序列(TR/TE:8000 ms/95 ms;FOV:240 mm×240 mm;层厚/层间距:5.0 mm/1.5 mm;矩阵:256×256)。注射0.1 mmol/kg钆螯合对比剂(Omniscan, GE Healthcare, Ireland)后获得CE-T1图像。
由于TCGA/TCIA数据库的图像来自不同的MRI设备和采集协议,并且与FHSXMU数据和SPPH数据的MRI设备和采集协议亦存在差异,为使不同设备采集的图像具有空间分辨率和灰度强度的可比性,我们对所有MRI图像进行重采样,从而消除不同型号MRI设备造成空间分辨率不一致的干扰,并通过最大最小值的归一化方法对图像强度进行归一化处理。此过程使用Python 3.10.4版(https://www.python.org/)的SimpleITK库实现。
对于TCGA/TCIA数据集,从数据库中下载获得IDH mut状态和MGMT meth状态的数据。对于FHSXMU和SPPH数据,IDH mut状态通过Sanger测序确定,采用Simlex OUP® FFPE DNA核酸提取试剂盒提取DNA,应用Chromas序列分析软件判定IDH状态。MGMT meth状态通过焦磷酸测序进行评估,使用BisulFlash™ DNA修饰试剂盒(Epigentek, USA)对提取的DNA进行修饰,获得MGMT启动子内10个CpG位点数据,平均甲基化≥8%,则认为肿瘤存在甲基化,未甲基化病例平均甲基化<8%[18]。
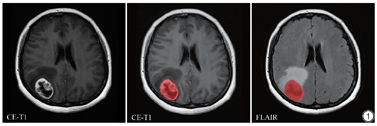
首先将T2-FLAIR图像通过FSL软件(http://fsl.fMRIb.ox.ac.uk/fsl/fslwiki/FSL)采用仿射变换与相应的CE-T1图像进行配准。肿瘤感兴趣区(region of interest, ROI)的勾画利用ITK-SNAP软件(http://www.itksnap.org)进行。由两名年资在10年以上的放射科主治医生进行双盲手动勾画,重叠区作为肿瘤的感兴趣区。并最终由一名年资超过20年的放射科主任医生审验。在CE-T1 MRI的图像上勾画出ROI,然后配准到T2-FLAIR图像上(图1)。

从CE-T1和T2-FLAIR图像中提取肿瘤ROI的1702个影像组学特征,每个MRI序列提取851个特征,包括:18个一阶直方图特征,14个形状特征,75个纹理特征[包括24个灰度共生矩阵(gray level co-occurence matrix, GLCM)特征,14个灰度依赖矩阵(gray level dependence matrix, GLDM)特征,16个灰度运行长度矩阵(gray level run length matrix, GLRLM)特征,16个灰度大小区域矩阵(gray level size zone matrix, GLSZM)特征,5个邻域灰度依赖矩阵(neighbouring gray tone difference matrix, NGTDM)特征],以及通过小波滤波后MRI图像的一阶特征和纹理特征得到的744个小波特征。影像组学特征提取采用开源软件FAE(http://github.com/salan668/FAE)实现,该软件基于PyRadiomics软件包(https://github.com/Radiomics/pyradiomics)。
根据IDH mut状态下MGMT启动子的是否发生甲基化,将LGGs分为IDH mut合并MGMT meth亚型、IDH mut合并氧6-甲基鸟嘌呤DNA甲基转移酶启动子非甲基化(O6-methylguanine-DNA methyltransferase promoter unmethylation, MGMT unmeth)亚型(该亚型简写为:IDH mut合并MGMT unmeth亚型)。由于两种分子亚型的构成比明显不均衡,为提高模型泛用性,将FHSXMU数据、SPPH数据和TCGA/TCIA数据进行整合,然后以7∶3比例随机分为训练集和测试集。统计分析采用R软件4.1.2版本(www.R-project.org)和SPSS软件23.0版本(https://www.ibm.com/analytics/spss-statistics-software)实现。
由于本研究两个分子亚型数据构成比差异明显,进行组间比较时,使用皮尔逊卡方检验或Fisher's确切检验。连续变量使用中位数和四分位数间距表示,组间比较使用Mann-Whitney U检验,双侧P<0.05表示差异具有统计学意义。
首先,使用Z-score变换对训练集的影像组学特征进行标准化,单因素逻辑回归(logistic regression, LR)进行特征初筛,P<0.05表示特征具有统计学意义。然后使用最小绝对收缩和选择算法(least absolute shrinkage and selection operator, LASSO)回归进一步进行特征选择,通过10-fold交叉验证选择最佳参数λ,在其值最小时选择系数非零的影像组学特征。由于数据集的构成比差异明显,为提高训练模型的准确性,使用合成少数过采样技术(synthetic minority over-sampling technique, SMOTE)方法对训练集少数类样本进行平衡化,再采用多因素LR进行建模。通过训练集和测试集的受试者工作特征(receiver operating characteristic curve, ROC)曲线以及校准曲线,对模型性能和拟合优度进行检验。绘制列线图进行可视化风险预测。
FHSXMU数据、SPPH数据和TCGA/TCIA数据合并后数据集的两个分子亚型在训练集和测试集的临床特征统计描述如表1所示。两种分子亚型在训练集和测试集的年龄、性别和肿瘤分级的P值均大于0.05,说明两种分子亚型在训练集和测试集的不同临床特征的组间比较差异均无统计学意义。
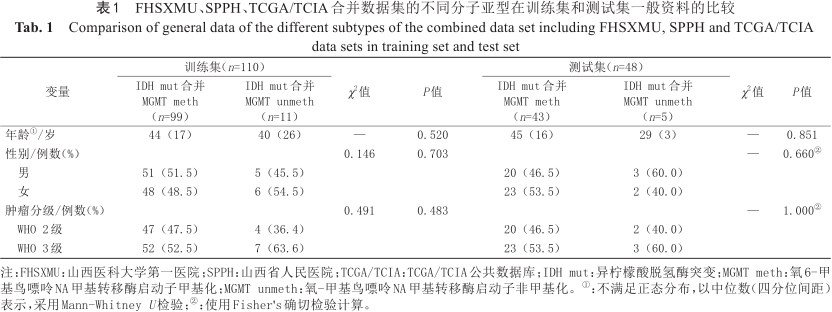
FHSXMU、SPPH、TCGA/TCIA合并数据集的不同分子亚型在训练集和测试集一般资料的比较
Comparison of general data of the different subtypes of the combined data set including FHSXMU, SPPH and TCGA/TCIA data sets in training set and test set
FHSXMU、SPPH、TCGA/TCIA合并数据集的不同分子亚型在训练集和测试集一般资料的比较
Comparison of general data of the different subtypes of the combined data set including FHSXMU, SPPH and TCGA/TCIA data sets in training set and test set
| 变量 | 训练集(n=110) | χ2值 | P值 | 测试集(n=48) | χ2值 | P值 | ||
|---|---|---|---|---|---|---|---|---|
IDH mut合并MGMT meth (n=99) | IDH mut合并MGMT unmeth (n=11) | IDH mut合并MGMT meth (n=43) | IDH mut合并MGMT unmeth (n=5) | |||||
| 年龄①/岁 | 44(17) | 40(26) | — | 0.520 | 45(16) | 29(3) | — | 0.851 |
| 性别/例数(%) | 0.146 | 0.703 | — | 0.660② | ||||
| 男 | 51(51.5) | 5(45.5) | 20(46.5) | 3(60.0) | ||||
| 女 | 48(48.5) | 6(54.5) | 23(53.5) | 2(40.0) | ||||
| 肿瘤分级/例数(%) | 0.491 | 0.483 | — | 1.000② | ||||
| WHO 2级 | 47(47.5) | 4(36.4) | 20(46.5) | 2(40.0) | ||||
| WHO 3级 | 52(52.5) | 7(63.6) | 23(53.5) | 3(60.0) | ||||
注:FHSXMU:山西医科大学第一医院;SPPH:山西省人民医院;TCGA/TCIA:TCGA/TCIA公共数据库;IDH mut:异柠檬酸脱氢酶突变;MGMT meth:氧6-甲基鸟嘌呤NA甲基转移酶启动子甲基化;MGMT unmeth:氧-甲基鸟嘌呤NA甲基转移酶启动子非甲基化。①:不满足正态分布,以中位数(四分位间距)表示,采用Mann-Whitney U检验;②:使用Fisher's确切检验计算。
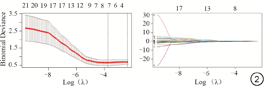
训练集经过单因素LR,仅CE-T1序列保留了21个放射组特征,T2-FLAIR序列为0。为了选择最佳的影像组学特征和解决过拟合问题,采用LASSO算法。最终,在λ值最小时,有7个系数不为零的特征被保留(图2)。经SMOTE数据平衡(阳性和阴性样本例数为99∶99),使用多因素LR建立影像组学模型(图3)。



影像组学模型在训练集和测试集的曲线下面积(area under the curve, AUC)值分别为0.842和0.935(图4)。在训练集的准确率为93.6%、精确率为94.2%、召回率为98.9%、F-Measure为0.965;在测试集的准确率为89.6%、精确率为93.1%、召回率为95.3%、F-Measure为0.942(表2)。

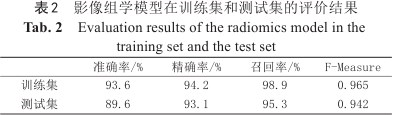
影像组学模型在训练集和测试集的评价结果
Evaluation results of the radiomics model in the training set and the test set
影像组学模型在训练集和测试集的评价结果
Evaluation results of the radiomics model in the training set and the test set
| 准确率/% | 精确率/% | 召回率/% | F-Measure | |
|---|---|---|---|---|
| 训练集 | 93.6 | 94.2 | 98.9 | 0.965 |
| 测试集 | 89.6 | 93.1 | 95.3 | 0.942 |
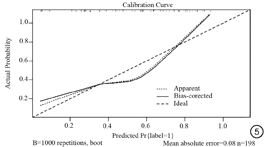
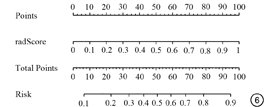
训练集的校准曲线显示了影像组学模型和实际结果有着较好的一致性,经Hosmer-Lemeshow检验的P值为0.1393(图5),测试集因为数据过少、且构成比的差异显著,R语言的glm和lrm函数无法拟合模型,因此无法生成校准曲线。根据模型计算的影像组学评分(radiomics score, Radscore),我们还绘制了列线图用于风险预测(图6)。


本研究探讨使用基于常规MRI的无创影像组学方法,建立一个用于预测LGGs的IDH mut合并MGMT meth亚型的影像组学模型,结果显示模型具有良好的预测性能(训练集和测试集的AUC分别为0.842和0.935)。本研究在LGGs层面,使用基于常规MRI的影像组学方法,预测IDH mut合并MGMT meth分子亚型为国内外首次被提出。由于LGGs的IDH mut合并MGMT meth作为联合的预测因子,预示着良好的OS和(或)PFS,并能从TMZ治疗中获益。因此,该亚型的准确预测,为LGGs患者分子分型的精确诊断、TMZ的临床决策及生存期预测提供重要的临床辅助价值。
使用影像组学的方法对胶质瘤IDH是否发生突变已经进行了广泛的研究,Wang等[19]通过基于MRI动态增强扫描序列和扩散加权序列,探讨了使用影像组学的方法对胶质瘤IDH状态的改善价值(AUC在训练集和测试集分别为0.939和0.880)。Manikis等[20]也探讨了基于动态磁化率对比MRI的影像组学方法对IDH状态的增益价值(AUC在训练集和独立的验证集的分别为0.678和0.667)。本研究在LGGs的IDH mut的基础上,结合MGMT启动子是否发生甲基化,将其进行分层,并获得良好的效果(AUC在训练集和测试集分别为0.842和0.935)。而胶质瘤IDH mut状态下,MGMT meth较MGMT unmeth有着更长的中位OS,且可以从TMZ的治疗中获益[5]。对于使用影像组学在胶质瘤MGMT meth研究中,Lohmann等[21]通过基于正电子发射断层显像(PET)/MRI的影像组学方法,预测了脑胶质瘤MGMT meth状态(最高的准确度为83%,而本研究为93.6%)。Huang等[22]通过一篇Meta分析证明了影像组学在研究胶质瘤MGMT meth的价值,并计算了多个模型的综合AUC值(训练集和独立的验证集分别为0.91和0.88)。而仅具有MGMT meth的单一分子标记物的胶质瘤,只能表明它可以从TMZ的治疗中获益。本研究中,LGGs的IDH mut合并MGMT meth亚型同时具有更好的生存期,以及从TMZ治疗获益的优点。
在胶质瘤多个分子生物标记物结合的影像组学研究中,Arita等[23]运用影像组学的方法,构建LGGs包含IDH合并TERT基因共同突变亚型的3种亚型的预测模型,并检验其性能。Fan等[24]使用影像组学的方法,构建了WHO 2级胶质瘤IDH mut下是否发生1p/19q共丢失的预测模型。以上研究证实了使用影像组学的方法在IDH mut状态合并其他肿瘤分子标记物的可行性。Zhang等[17]使用自动机器学习的放射组学方法,对胶质瘤IDH mut和MGMT meth共同发生进行预测研究(多个模型的平均AUC=0.951)。与本研究相比,该研究包含了WHO 2~4的三个级别的胶质瘤,适用范围更广。然而,该研究中,作为对照组的其他亚型包含了IDH mut合并MGMT unmeth亚型、IDH野生型合并MGMT meth和IDH野生型合并MGMT unmeth三种亚型,且IDH mut合并MGMT unmeth亚型仅为一例,同时,WHO 4级的胶质瘤占全部胶质瘤的51.8%。而本研究着重于WHO 2、3级的胶质瘤,并以IDH mut合并MGMT unmeth亚型作为对照组,同时,为解决两个亚型的构成比不均衡问题,我们使用了SMOTE算法进行数据平衡化,最终的影像组学模型亦具备良好的预测性能。
为解决数据构成比问题,我们引入了SMOTE算法进行数据平衡化,其优点是:相较于过采样对于原始数据随机抽取而言,该算法是通过少数样本中任意单个样本与其临近的k个样本(通常默认为5)进行插值,从而提高分类器泛化能力,同时,与只对多数类进行欠采样相比,该方法可以实现更好的分类器性能[25, 26]。当然,SMOTE也有缺点,如由于k近邻的选择而产生的噪声和样本复制问题[27]。然而,样本信息能否真实地反映总体,是模型好坏的关键。SMOTE算法在一定程度上解决了样本均衡问题,这也体现在本研究中的影像组学模型具有较好的性能。
本研究存在以下局限性:(1)由于对照组(即IDH-mut合并MGMT unmeth亚型)样本数量过少导致的构成比不均衡,尽管通过SMOTE算法得以数据平衡化,但毕竟不是真实样本的反映;(2)缺乏独立的外部验证集;(3)由于构成比的显著差异,导致临床变量统计学不显著,未能实现影像组学-临床结合模型的建立。因此,本研究还需要获得更多的少数类样本,在保证少数类样本足够丰度的前提下,进一步实现影像组学模型对真实总体的反映。同时,使用独立外部验证集进行检验可以进一步验证影像组学模型的普遍适用性。最后,足够的少数类样本的获得也可能使临床变量具统计学有意义,使影像组学-临床结合模型的实现成为可能,从而进一步提高模型的性能。
综上所述,本研究是通过基于术前MRI的影像组学方法,构建了一个用于预测LGGs的IDH mut合并MGMT meth亚型的影像组学模型。结果表明该模型具有良好预测性能,从而为LGGs患者的分子亚型的精确诊断、化疗药物TMZ使用的决策、患者OS和(或)PFS的评估提供重要的临床辅助价值。
National Natural Science Foundation of China (No. U21A20386, 81971592, 81971593).
全体作者均声明无利益冲突。



























