
构建基于卷积神经网络的儿童病毒性脑炎MRI分类与早期诊断模型,探讨其对儿童病毒性脑炎早期诊断、精准治疗和改善患儿预后的价值。
收集浙江大学医学院附属儿童医院2020至2022年期间颅脑MRI影像数据1077例,其中病毒性脑炎患儿577例,非病毒性脑炎儿童500例。运用卷积神经网络中的Squeeze-and-Excitation Residual Networks(SE-ResNet)模型构建儿童病毒性脑炎MRI分类与早期诊断模型并与Convolutional Block Attention Module Residual Networks(CBAM-ResNet)、Mobile Networks(MobileNet)、Residual Networks(ResNet)、Shuffle Networks(ShuffleNet)模型进行了对比。
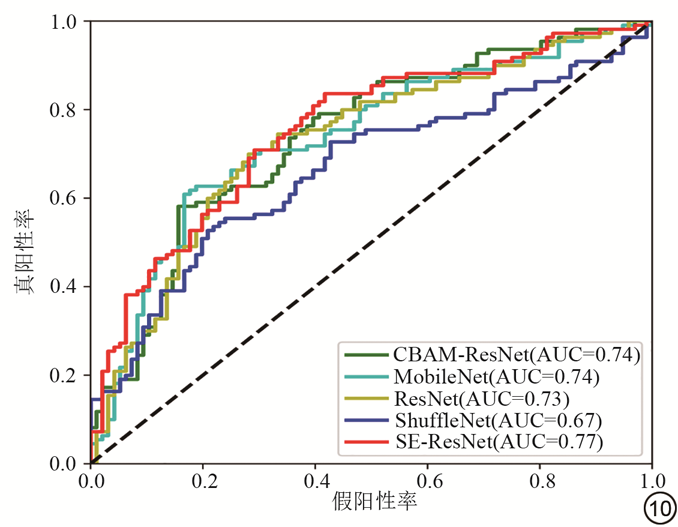
所有模型在训练集上都达到了收敛。SE-ResNet、CBAM-ResNet、MobileNet和ShuffleNet模型在训练集训练100轮后准确率都达到90%以上,而只有CBAM-ResNet模型和本研究选用的SE-ResNet模型在验证集上同样取得了90%以上的准确率。在测试集上,CBAM-ResNet具有最高的准确率73.91%,ResNet具有最高的召回率75.45%,但只有本文所用SE-ResNet模型在准确率和召回率都达到较高水平,并且取得最好的F1得分和曲线下面积(area under the curve, AUC)值:准确率为70.83%,召回率为72.73%,AUC为0.77,F1得分为0.7183。
运用人工智能技术结合MRI实现儿童病毒性脑炎早期诊断是可行的,本研究为进一步实现全面的儿童脑炎早期诊断、精准治疗和改善脑炎患儿预后提供了理论和应用基础。

本刊刊出的所有论文不代表本刊编委会的观点,除非特别声明
病毒性脑炎(viral encephalitis, VE)是由脑实质炎症引起的神经系统疾病,临床症状主要表现为头疼、发热、意识障碍等,其在儿童中的发病率显著高于成人[1, 2]。儿童VE,尤其是早期阶段具有临床表现多样、病情发展迅速、实验室阳性检测率低等特点,给临床早期诊断和精准治疗带来了巨大挑战,从而使部分患儿发展成为重症患者[3]。重症脑炎常常导致患儿遗留不可逆的神经系统后遗症(如:脑积水、癫痫、智力低下、肢体臃肿等),甚至危及患儿生命(约有8.4%的死亡率)[4]。现阶段脑脊液检测是脑炎诊断的“金标准”,但其依赖腰椎穿刺技术且耗时较长,给患儿带来痛苦和创伤[5]。因此,运用新的方法和无创技术实现儿童VE早期精准诊断和治疗并改善患儿预后,具有重要的临床价值。
MRI因其安全、无创、组织分辨率高等优点更适合辅助临床医生进行脑部疾病的诊断[6, 7, 8, 9]。此外,人工智能技术的蓬勃发展为基于无创MRI影像的疾病早期诊断和分类方法提供了崭新的思路[10, 11, 12, 13, 14]。POLONI等[15]研究者利用海马不对称属性进行MRI影像分类,成功在阿尔茨海默病(Alzheimer's disease, AD)患者的数据集上取得了82.59%的准确率。LATIF[16]团队运用卷积神经网络和增强模糊算法实现了脑肿瘤患者MRI影像的分割和分类,为脑肿瘤诊断提供了一条新的途径。在国内,来自常州大学人工智能实验室的GU等[17]科研人员在脑肿瘤MRI分类上也取到了突破,提出的一种基于局部约束卷积字典学习的影像分类方法在临床验证数据集中获得了超过97%的准确率。然而,基于MRI影像的大脑疾病与人工智能技术结合的研究主要集中在AD和脑肿瘤等疾病,针对儿童VE的分类和早期诊断研究较少。在儿童VE早期诊断研究中陈立晔提出脑电图具有重要的意义,但其特异性不高,在诊断困难时建议继续予以MRI检查[18]。也有研究者认为MRI在儿童VE的诊断中具有重要价值,但因其MRI影像特征与缺血卒中、低级别胶质瘤、线粒体脑病等相似,故对VE临床表现不典型的患儿诊断困难[19, 20, 21, 22]。针对上诉问题本研究创新性地提出了将基于卷积神经网络的Squeeze-and-Excitation Residual Networks(SE-ResNet)模型应用于儿童VE的MRI影像分类和早期诊断,为实现儿童VE的早期发现和精准治疗提供了理论和应用基础。
本文所提出的方法流程如图1所示,共有以下六步:(1)收集相关患儿资料;(2)通过纳入标准进行数据的筛选;(3)对原始MRI影像数据进行预处理;(4)对数据进行分组;(5)建立SE-ResNet分类模型;(6)利用测试集对模型性能进行验证。


本研究遵守《赫尔辛基宣言》,经浙江大学医学院附属儿童医院伦理委员会批准(〔伦〕审编号:2020-IRB-058)并免除患儿及患儿家属知情同意。回顾性纳入我院2020至2022年期间进行颅脑MRI检查的相关儿童患者资料。纳入标准:(1)年龄1个月~16岁;(2)完成颅脑MRI检查;(3)MRI影像资料完善;(4)既往无严重神经系统疾患史。排除标准:(1)既往有脑外伤或颅脑手术病史;(2)出院时诊断不明确。
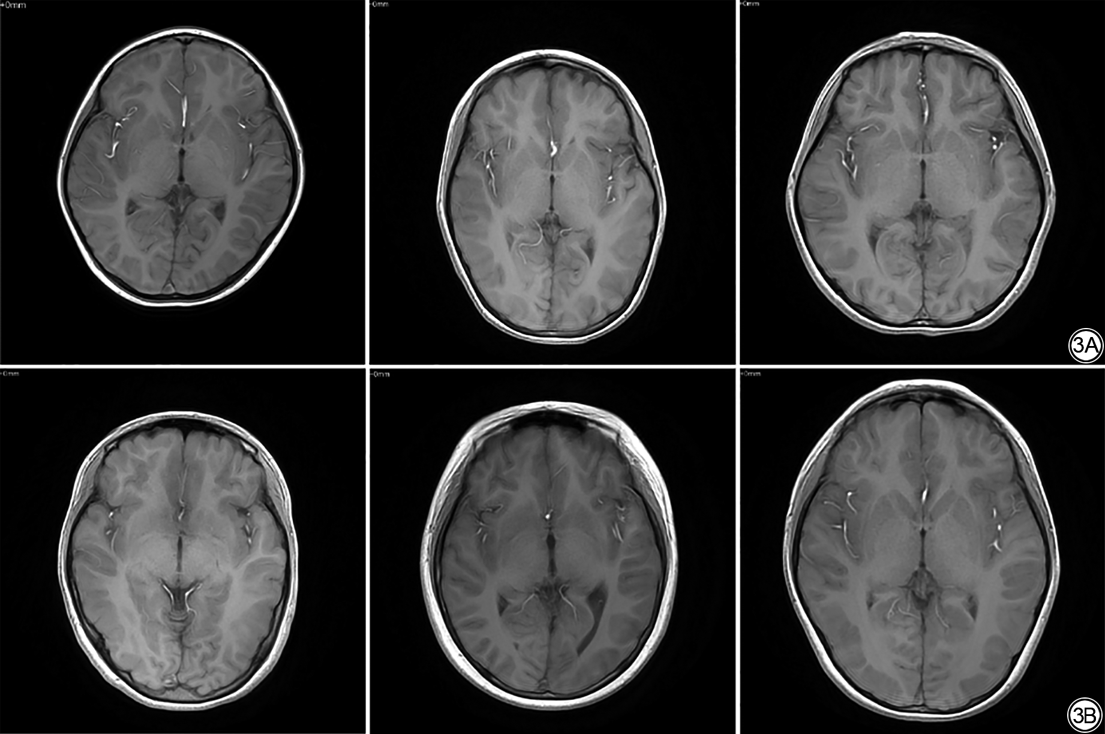
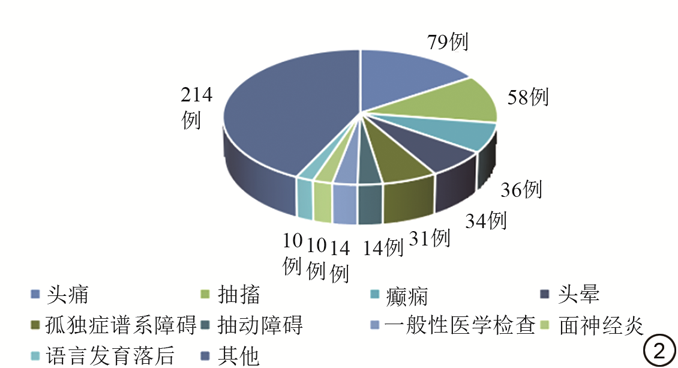
最终1077例纳入研究,男583例,女494例,年龄(5.94±3.85)岁(范围1个月~16岁)。其中经神经内科临床确诊为VE的患儿577例,男312例,女265例,年龄(5.57±3.78)岁(范围1个月~15.67岁)。非VE儿童500例,男271例,女229例,年龄(6.36±3.88)岁(范围1个月~16.17岁)。非VE儿童的MRI检查原因人数分布如图2所示。儿童VE的主要诊断标准为:头痛、发热、嗜睡、认知/行为改变等精神状态异常;出现局部或弥漫性脑炎症状;脑脊液检测结果异常;CT、MRI等脑影像学结果提示异常等并排除由创伤、代谢混乱、肿瘤等非感染性病因引起的疾病[23, 24]。本文是基于临床轴位Fast Field Echo(FFE)T1WI序列图像进行研究,VE患儿与非VE患儿T1WI序列图像如图3所示。


MRI影像数据来源于患儿MRI平扫检查结果,使用来自荷兰飞利浦公司3 T MRI(Achieva; Philips Healthcare)进行扫描检查。轴位FFE T1WI序列扫描参数为:翻转角80°,TR 191.68 ms,TE 2.13 ms,扫描矩阵 512×512,FOV 230 mm×230 mm,层厚6 mm,层间隔 1 mm。
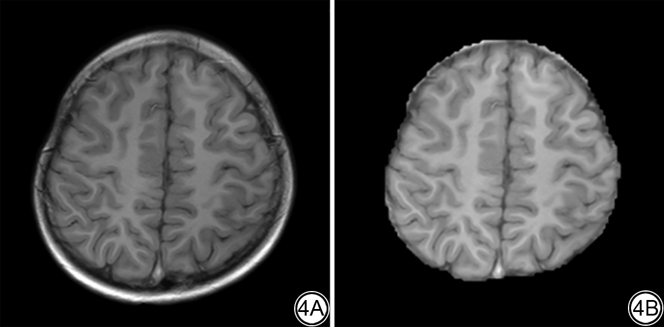
患儿因大脑发育不同导致实际扫描过程中存在扫描层数不一致的情况,从而无法满足模型单一输入尺寸的要求。本研究选取所有案例中最具有代表性的数量作为标准,对于未达到此层数的案例,对其首尾通过复制进行补充,以达到模型的输入要求。在本研究中,所有案例的层数统一为18,输入大小为512×512×18。然后通过HD-BET网络对脑部区域进行了分割,分割后的区域如图4所示[25]。最后对数据进行了归一化(0均值1方差)。

随机选取70%的数据作为训练集,10%的数据作为验证集,20%的数据作为测试集。训练集共有754例,其中确诊为VE的患儿404例,非VE儿童350例;验证集共有108例,其中确诊为VE的患儿58例,非VE儿童50例;测试集共有215例,其中确诊为VE的患儿115例,非VE儿童100例。
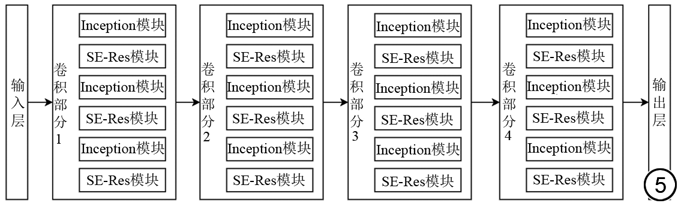
本文选用卷积神经网络中的SE-ResNet深度学习模型。其包含四个卷积部分,每部分由三个包含Inception模块、SE(Squeeze-and-Excitation)-Res(Residual)模块模块的子模块组组成,其基本结构如图5所示:

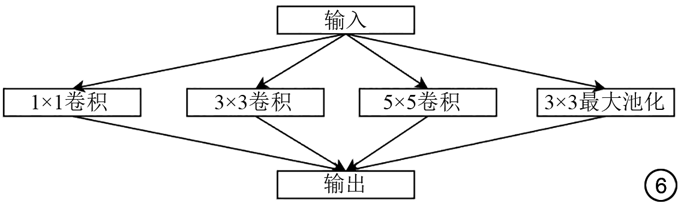
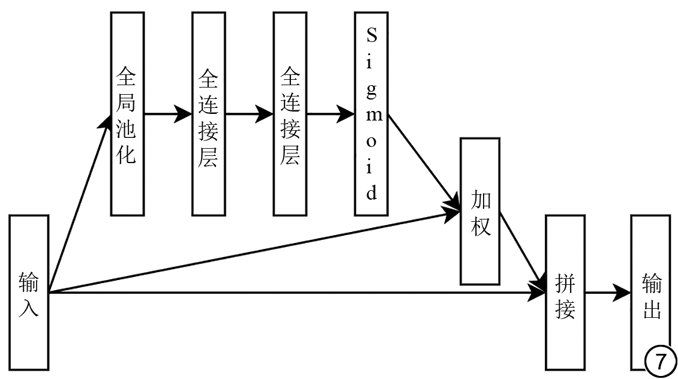
在子模块组中,首先通过Inception模块获取不同尺度的特征数据。如图6所示,对于输入X,Inception模块设计了三个尺寸分别是1×1、3×3、5×5的卷积核以及一个大小为3×3的池化核,即。输入X通过此层可以得到四个不同尺度的特征,然后通过拼接获得此模块的输出并接入到SE-Res模块,从而对Inception子模块获取的特征进行加权筛选。如图7所示,SE-Res模块首先用1×1大小的全局池化层获得同等大小的特征矩阵,之后用1/16的通道数进行全连接卷积,获得特征矩阵,接着用通道数的卷积层进行卷积,获得特征矩阵,然后用sigmoid激活函数将权重归一化到0~1之间,获得特征矩阵,接着用F4进行加权,获得加权特征,将两个特征矩阵进行拼接得到最后的特征矩阵。最终分类预测结果通过全连接卷积层输出。


本文所运用的SE-ResNet模型,与Convolutional Block Attention Module Residual Networks (CBAM-ResNet)、Mobile Networks (MobileNet)、Residual Networks (ResNet)、Shuffle Networks (ShuffleNet)模型进行了对比,所有模型都是基于PyTorch实现,模型的训练、验证、测试在Ubuntu 16.04服务器上完成[26, 27, 28, 29]。服务器的配置如下:CPU Intel E5-1650 3.50 GHz,内存64G DDR4,显卡为RTX 2080Ti。在训练时,所有模型的批次大小为32,输入大小为20×512×512,模型选择Adam作为优化器,初始学习率为0.001,训练次数为100轮,选用交叉熵作为损失函数。基于ResNet的模型均选用ResNet50作为基础网络。全部模型在包含了225例儿童MRI影像数据(VE患儿115例,非VE儿童100例)的测试集上进行了验证,并通过准确率、召回率、受试者工作特征(receiver operating characteristic, ROC)曲线及其曲线下面积(area under curve, AUC)和F1评分等指标对模型进行评价。
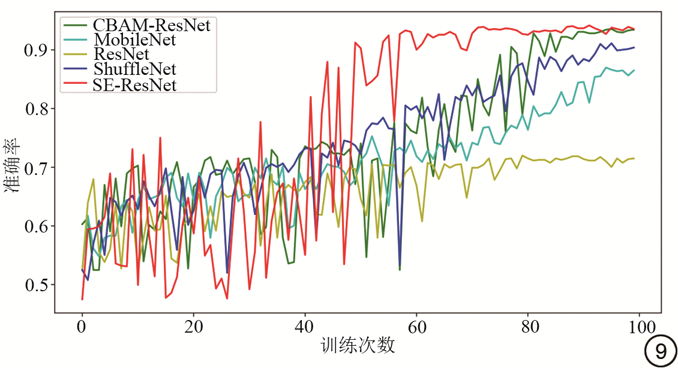
如图8所示,所有模型在训练集上都达到了收敛。如图9所示,SE-ResNet、CBAM-ResNet、MobileNet和ShuffleNet模型在训练集训练100轮后准确率都达到90%以上,而只有CBAM-ResNet模型和本研究选用的SE-ResNet模型在验证集上同样取得了90%以上的准确率,说明各模型在训练集上都达实现了收敛,并在验证集上表现为一直得到提升。


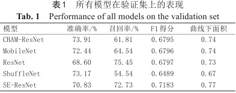
如表1、图10所示,在测试集上,CBAM-ResNet具有最高的准确率73.91%,ResNet具有最高的召回率75.45%,但只有本研究所用方法在准确率和召回率都达到较高水平,并且取得最好的F1得分和AUC:准确率为70.83%,召回率为72.73%,AUC为0.77,F1得分为0.7183。Delong检验结果表明本文所用模型与其他4个模型在正确率上存在显著性差异(P<0.05)。因此本文所用SE-ResNet模型相对其他模型,更适合用于儿童VE分类任务。
所有模型在验证集上的表现
Performance of all models on the validation set
所有模型在验证集上的表现
Performance of all models on the validation set
| 模型 | 准确率/% | 召回率/% | F1得分 | 曲线下面积 |
|---|---|---|---|---|
| CBAM-ResNet | 73.91 | 61.81 | 0.6795 | 0.74 |
| MobileNet | 72.44 | 64.54 | 0.6796 | 0.74 |
| ResNet | 68.60 | 75.45 | 0.6797 | 0.73 |
| ShuffleNet | 73.17 | 54.54 | 0.6489 | 0.67 |
| SE-ResNet | 70.83 | 72.73 | 0.7183 | 0.77 |

SE-ResNet在测试集上各年龄段的表现
Performance of SE-ResNet models on the test set at all ages
SE-ResNet在测试集上各年龄段的表现
Performance of SE-ResNet models on the test set at all ages
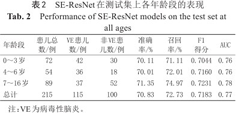
| 年龄段 | 患儿总数/例 | VE患儿数/例 | 非VE患儿数/例 | 准确率/% | 召回率/% | F1得分 | AUC |
|---|---|---|---|---|---|---|---|
| 0~3岁 | 72 | 42 | 30 | 70.11 | 71.11 | 0.7044 | 0.76 |
| 4~6岁 | 54 | 36 | 18 | 70.01 | 72.01 | 0.7160 | 0.76 |
| 7~16岁 | 89 | 37 | 52 | 71.35 | 74.97 | 0.7231 | 0.78 |
| 总计 | 215 | 115 | 100 | 70.83 | 72.73 | 0.7183 | 0.77 |
注:VE为病毒性脑炎。
为证明模型对不同年龄段儿童的表现差异性,我们将测试集分成了0~3、4~6、7~16岁三组。SE-ResNet模型在测试集上各年龄段的表现如表2所示,模型在各年龄段上表现相差不大,说明模型可以通用于各年龄段。
最后选取100例患儿(儿童VE和非儿童VE各50例)的相关MRI影像作为待测图像进行试验,将模型分类结果与专业人员影像判断结果、专业人员在模型辅助下的影像诊断结果进行对比。试验结果显示,医生影像判断的准确率与召回率分别是53.00%与54.00%,远低于模型的准确率72.00%与召回率78.00%,但是医生在模型辅助下的诊断结果要比模型的性能要好,准确率与召回率分别达到75.00%与84.00%,说明模型对于医生的判断具有较大的提升,具有较高的临床应用价值。
本文创新性地提出一种非侵入性的儿童VE分类模型,可以基于儿童患者的MR图像中自动分辨儿童VE和非儿童VE患者。该分类模型是包含862例的患儿的数据集上进行训练开发,并在包含215例患儿的数据集上进行了验证,结果表明本文方法获得了最佳的综合性能(准确率70.83%,召回率72.73%,AUC为0.77,F1为0.7183),也证明了利用SE-ResNet区分儿童VE与非VE的MRI影像是可行的。据我们所知,这是首批使用MR图像进行儿童VE诊断的研究之一,虽然准确率低于作为金标准的脑脊液检查,但是证明了非侵入性诊断的可能性。
儿童VE是一种常见中枢神经系统感染性疾病,具有高致残率和死亡率的特点,常常难以治愈[1]。现阶段儿童脑炎的诊断主要依赖临床症状、实验室检查(脑脊液检查)、影像学和脑电图检测等方法[24]。然而脑炎患儿早期临床表现不典型,影像学和脑电图检测则只有重症时才能明显观察到病变区域,作为脑炎诊断金标准的脑脊液检查是一种耗时较长的有创技术手段[5],如图3所示,VE患儿与非VE患儿在影像学上表现并无明显差异。这些局限性妨碍了缺乏神经内科专科医生的基层医院对于患儿在发病早期阶段的精准诊断和及时干预,延误了治疗时机,导致患儿致残致死率增加[30]。
本研究采用MR图像进行儿童VE的自动诊断,并以不同的方式验证了我们的算法。据我们所知,对儿童脑炎非侵入性诊断的研究较少。研究结果表明,深度学习算法可以执行此任务。我们的目标不仅是建立一个儿童VE诊断模型,而且还探索该模型在协助神经放射科医生方面的临床效用。本研究进行了3次测试来评估分类模型在不同情况下的性能。第一个试验在患儿的数据集上对分类模型进行了验证,以避免统计偏差。该测试的目的是将SE-ResNet分类模型的性能与常用模型的性能进行比较。第二个试验将测试集分成了三个年龄段其目的是验证模型在儿童各年龄段的表现差异。第三个试验是在包含100例患儿的数据集上进行验证,其目的是调查人类评估者是否可以在分类模型的帮助下提高其诊断准确性。这些实验相互补充,表明了分类模型在临床环境中应用的实用性。
在评估分类模型用于儿童VE自动诊断的过程中,分类模型在测试数据集中都取得了较好的性能。这些结果表明,分类模型在诊断儿童VE方面可能可以为临床提供一条新的探索路径。此外,分类模型还拥有比人工判读更高的诊断效率,一定程度上节省了人力物力。
SE-ResNet模型在测试集上各年龄段的表现相差不大,说明模型可以通用于各年龄段,这可能与图像预处理时首先去除了头骨并进行了偏移校正,使模型只关注于分割出来的头部区域,从而避免了不同年龄段患儿脑部发育不一致对模型的影响有关。
分类模型辅助评估试验中,神经放射科医生、分类模型以及神经放射科医生在分类模型的帮助下分别诊断儿童VE。通过这种方式,神经放射科医生可以使用分类模型结果作为参考,以尝试提高整体诊断准确性。研究结果表明,分类模型可以整合到临床工作流程中,并帮助神经放射科医生做出更准确的诊断。开发这种分类模型的核心目标是帮助神经放射科医生更准确地进行诊断。当然,有时分类模型系统可能会误导神经放射科医生做出错误的诊断。我们与医生一起调查了这些诊断,医生的反馈是他们对自己的诊断没有信心,因为图像中没有足够的信息或独有的特征。试验表明,本文所提出的分类模型可以辅助医生判断儿童是否患有VE并给予及时干预,改善VE患儿预后,此方法无需对患儿进行腰椎穿刺,避免了患儿的创伤,也极大地提升了诊断效率。
SE-ResNet是一种结合了SENet(Squeeze-and-Excitation Networks)和ResNe(Residual Networks)的图像分类模型[28, 31]。SENet的核心思想在于提出了具有自注意力机制的SE模块,SE模块通过对不同特征通道权重的学习,从而对特征矩阵进行加权,起到了自我筛选特征矩阵的功能。ResNet是一种基于VGGNet的网络模型,其核心在于通过残差模块解决了深度学习模型因网络过深导致的梯度消失或爆炸问题。HU等[31]采用SENet block和ResNeX结合而成的SE-ResNet,在 ILSVRC 2017的图像分类任务中拿到第一。目前基于SE-ResNet的深度学习模型被广泛应用于肺结节、乳腺、心脏、骨龄等医学领域,在医学图像分类方向表现优越[32, 33, 34, 35]。因此本文选择SE-ResNet网络模型,并在其基础上加入了Inception模块,来提升网络对不同尺度特征的学习能力[36, 37, 38]。在测试集上,本文模型与CBAM-ResNet、MobileNet、ResNet和ShuffleNet模型进行了对比实验,结果也证明了SE-ResNet区分儿童VE与非VE患儿MR影像是可行的。
需要指出的是,本研究尚存在不足之处:(1)本研究的数据均来自同一家儿童医院,在外部数据集中尚未得到验证。今后的工作中,我们会利用区域医疗中心的优势,收集不同医疗机构的儿童脑炎数据并在此过程中不断迭代优化模型。(2)模型分类和诊断结果只在儿童VE检测中有较高的准确率,在其他类型脑炎的分类结果一般。运用MRI影像增强技术获得更高质量的临床序列MRI影像并用于儿童脑炎的诊断研究中以期获得全面的儿童脑炎分类诊断模型是我们今后研究的目标。
综上所述,人工智能技术的发展为传统疾病的诊断研究开辟了崭新的道路。本研究运用SE-ResNet网络构建了基于MRI影像的儿童VE早期诊断和分类模型。结果表明基于无创技术的儿童VE早期诊断是可行的,为进一步实现全面的儿童脑炎早期诊断、精准治疗和改善脑炎患儿预后提供了理论和应用基础。
黄坚, 余卓, 徐璐, 等. 基于卷积神经网络的儿童病毒性脑炎磁共振影像分类与早期诊断研究[J]. 磁共振成像, 2023, 14(1): 54-60.
HUANG J, YU Z, XU L, et al. Classification and early diagnosis of children viral encephalitis on MRI images based on convolutional neural network[J]. Chin J Magn Reson Imaging, 2023, 14(1): 54-60.
National Key R&D Program of China (No. 2019YFE0126200); National Natural Science Foundation of China (No. 62076218).
全体作者均声明无利益冲突。



























