
探讨无创产前筛查(NIPS)在胎儿染色体非整倍体异常(CAA)及全基因组拷贝数变异(CNV)筛查中的临床应用价值。
选择2018年1月至2019年12月,广西壮族自治区60余家医疗机构采集血液样本后送至本院完成NIPS的50 975例单胎孕妇为研究对象。其分娩年龄为13~54岁,高龄孕妇(分娩时年龄≥35岁)为17 216例(33.77%),血样标本采集时,孕龄为10~36孕周。根据血清学筛查结果、孕妇年龄、妊娠史/家族史,将其分为高风险组[n=22 852,血清学筛查结果高风险(+)、高龄孕妇(+)、不良孕产史/家族史者纳入(+),至少满足其中一项者],中风险组[n=4 584,血清学筛查结果中风险/临界风险(+)、胎儿超声检查软指标异常(+),至少满足二者之一者]和低风险组(n=23 539,无上述风险因素者)。对于NIPS提示高风险者,采用介入性产前诊断,进行胎儿G显带染色体核型分析及染色体微阵列分析(CMA)。本研究经广西壮族自治区妇幼保健院伦理委员会批准(审批文号:桂妇保院医伦审[2020]9-2号)。
①50 975例接受NIPS孕妇中,702例NIPS筛查结果显示高风险,高风险率为1.38%。其中21-、18-、13-三体高风险率分别为0.34%(175/50 975)、0.13%(67/50 975)和0.12%(63/50 975),性染色体非整倍体异常(SCA)高风险率为0.40%(205/50 975),罕见染色体数目异常(RCA)高风险率为0.19%(96/50 975),基因组拷贝数变异(CNV)高风险率为0.19%(96/50 975)。②在702例NIPS高风险者中,对555例(79.06%)孕妇进行胎儿染色体核型分析和(或)CMA,93例(16.76%)拒绝进行产前诊断,42例(7.57%)因为流产或已引产而未进行产前诊断,NIPS结果为高风险孕妇的失访率为1.71%(12/702)。555例接受介入性产前诊断的胎儿中,271例阴性胎儿的NIPS和介入性产前诊断结果基本一致,284例假阳性中,16例胎儿的介入性产前诊断结果为染色体异常,但与NIPS高风险结果不一致。③21-三体、18-三体、13-三体、SCA、RCA和CNV的阳性预测值分别为85.09%(137/161)、57.14%(28/49)、16.67%(9/54)、42.31%(66/156)、5.56%(4/72)和42.86%(27/63)。④16例经介入性产前诊断未检测到NIPS所提示的高风险结果,却检测到其他异常结果。其中,4例21-三体高风险胎儿中,3例检测到CNV,1例为21号染色体复杂的结构变异;1例18-三体高风险胎儿中,染色体核型分析结果提示为21-三体;2例13-三体高风险胎儿中,1例为染色体结构变异的嵌合体,另1例为CNV;3例SCA高风险和2例RCA高风险胎儿中,均检测到其他染色体的CNV;4例CNV高风险胎儿中,1例检测到47,XYY,3例为其他染色体的CNV异常。
NIPS对于21-、18-、13-三体等常见CAA的筛查具有较高临床实际应用价值。NIPS对于性染色体单体、RCA及CNV的筛查阳性预测值偏低,但是结合产前超声等相关检查,可为遗传咨询和进一步介入性产前诊断提供依据。

未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计,除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。本刊为电子期刊,以网刊形式出版。
胎儿染色体异常是导致出生缺陷的一个主要因素,包括染色体非整倍体异常(chromosome aneuploidy abnormality, CAA),如21-、18-及13-三体综合征(trisomy syndrome,TS),性染色体异常,其他染色体数目异常及染色体结构变异等[1]。随着孕妇外周血中胎儿游离DNA(cell free DNA,cf-DNA)的发现及高通量测序技术(high-throughput sequencing)的快速发展,直接分析母体外周血中胎儿cf-DNA的无创产前筛查(non-invasive prenatal screening,NIPS)成为产前筛查胎儿染色体异常的新方向[2]。目前,NIPS以其无创性、高通量、高敏感度和高特异度等优势,于胎儿21-、18-、13-TS及性染色体非整倍体(sex chromosome aneuploidy,SCA)产前筛查中,得到广泛应用[3,4]。NIPS还可发现胎儿罕见染色体数目异常(rare chromosomal abnormality,RCA)及全基因组拷贝数变异(copy number variation,CNV)[5]。尽管许多实验室已将NIPS用于胎儿RCA筛查,但其准确性、特异度等仍有待进一步研究证实。本研究通过回顾性分析2018年1月至2019年12月广西壮族自治区妇幼保健院50 975例单胎孕妇的NIPS结果,探讨NIPS对广西地区胎儿CAA及CNV筛查中的临床应用价值。现将研究结果报道如下。
选择2018年1月至2019年12月,广西壮族自治区60余家医疗机构采集血液样本后送检至本院完成NIPS的50 975例单胎孕妇为研究对象,血液标本采集时,孕龄为10~36孕周,其分娩年龄为13~54岁,其中高龄孕妇(分娩时年龄≥35岁)为17 216例(33.77%)。根据血清学筛查结果、孕妇年龄、妊娠史/家族史,将其分为高风险组[n=22 852,血清学筛查结果高风险(+)、高龄孕妇(+)、不良孕产史/家族史者纳入(+),至少满足其中一项者],中风险组[n=4 584,血清学筛查结果中风险/临界风险(+)、胎儿超声检查软指标异常(+),至少满足二者之一者]和低风险组(n=23 539,无上述风险因素者)。本研究遵循的程序符合广西壮族自治区妇幼保健院医学科研与临床试验伦理委员会批准(审批文号:桂妇保院医伦审[2020]9-2号),并与所有受试者签署临床研究知情同意书。
本研究纳入标准:具有进行NIPS适应证,具体包括以下孕妇。①血清学筛查结果胎儿染色体异常者,②产前超声筛查提示胎儿异常者,③年龄≥35岁者,④具有异常妊娠史者,⑤其他要求进行NIPS者。排除标准:①曾接受移植手术及干细胞治疗者,②过去4周内曾接受免疫治疗和(或)在1年内输注异体血液制品者。
本研究血清学筛查高风险是指21-三体风险值≥1/270和(或)18-三体风险值≥1/380;中风险/临界风险是指21-三体风险值为1/1 000~1/270和(或)18-三体风险值为1/1 000~1/350[6]。
抽取孕妇外周肘静脉血5 mL,及时分离血浆后提取血浆cf-DNA,通过酶反应、加入接头修饰DNA片段,采用聚合酶链式反应(polymerase chain reaction,PCR)扩增制备DNA文库,经Qubit质量控制和定量后,采用高通量基因测序仪BGISEQ-500(华大基因)进行胎儿基因测序。通过生物信息分析,根据检测结果的Z值,得出胎儿染色体异常的风险,Z值在—3.0~3.0为正常范围,定义为低风险,而超过此范围则定义为高风险[7]。
①介入性产前诊断方法:对NIPS结果为高风险者,经详细的遗传咨询后,根据孕妇及其家属意愿,自愿进行介入性产前诊断,包括胎儿G显带染色体核型分析和染色体微阵列分析(chromosome microarray analysis,CMA)。②标本采集:对于中孕期孕妇,于孕龄为16~22孕周时,在超声引导下经腹壁羊膜腔穿刺术,抽取羊水30 mL,其中20 mL用于胎儿G显带染色体核型分析,10 mL用于胎儿CMA。对于晚孕期孕妇,则于孕龄为24~35孕周时,在超声引导下经腹壁脐静脉穿刺术,抽取脐带血1 mL,其中0.5 mL用于胎儿G显带染色体核型分析,0.5 mL用于胎儿CMA。③胎儿G显带染色体核型分析:将穿刺获取的羊水,1 400 r/min离心8 min后,去掉上清液,剩余2 mL的沉淀混匀后,无菌接种于2.5 mL羊水培养基,置于37 ℃、5% CO2培养箱培养7~9 d;获取的脐血标本,混匀后取0.5 mL在无菌条件下接种于外周血培养基中,置于37 ℃、5%CO2培养箱中培养3 d。培养后的细胞以秋水仙素阻断细胞有丝分裂,获取中期分裂象细胞,收获制片后进行胎儿G显带染色体核型分析。每份标本计数20个胎儿中期分裂象细胞,分析5个核型,若出现染色体嵌合,则计数分析至50个胎儿中期分裂象细胞。参照《人类细胞遗传学国际命名体制(2016版)》(International System for Human Cytogenetic Nomenclature-2016),确定胎儿细胞染色体核型。④CMA:对于穿刺获取的羊水,1 500 r/min离心15 min后,保留200 μL沉淀进行DNA提取;对于脐血标本,颠倒混匀后,取200 μL进行DNA提取。采用基因组DNA提取试剂盒(QI Aamp DNA Blood Mini Kit,批号为51106,德国QIAGEN公司)提取羊水、脐血样本中胎儿基因组DNA,采用Nanodrop2000分光光度计(美国Thermo Fisher公司)测定胎儿DNA浓度及纯度。根据HumanCytoSNP-12芯片检测的标准操作流程(美国Illumina公司)对胎儿DNA进行全基因组扩增、片段化、杂交、荧光染色及扫描,扫描获得的数据采用Karyostudio 1.4.3软件(美国Illumina公司)分析。根据国内相关指南[8,9],通过与正常人基因组变异数据库(Database of Genomic Variants,DGV),International Standards for Cytogenomic Arrays(ISCA)(http://www.iscaconsortium.org/),在线人类孟德尔遗传数据库(Online Mendelian Inheritance in Man,OMIM) (https://omim.org/),DECIPHER (https://decipher.sanger.ac.uk),ClinVar(https://www.ncbi.nlm.nih.gov/clinvar/)等数据库进行比对分析,判断所检出胎儿CNV致病性。根据相关指南和评估标准[8,10],胎儿CNV的致病性可被分为5类:致病性CNV、可能致病性CNV、临床意义不明CNV、可能良性CNV及良性CNV。
采用SPSS 22.0统计学软件进行统计学分析,采用Ssize软件,确定满足本研究统计检验的最小样本量。对于本研究计数资料,如高风险率、假阳性率、敏感度、特异度、阳性预测值等,采用百分比(%)表示。所有统计学检验采用双侧检验,以P<0.05表示差异有统计学意义。
对50 975例孕妇进行NIPS结果显示,胎儿CAA和CNV高风险者为702例(1.38%),其中SCA高风险者所占比例最高为29.20%(205/702),其次是21-TS高风险所占比例为24.93%(175/702),而RCA、CNV、18-及13-TS高风险所占比例分别为13.68%(96/702)、13.68%(96/702)及9.54%(67/702)及9.00%(63/702)。
3组孕妇NIPS结果阳性率整体比较,差异有统计学意义(χ2=23.61,P<0.001),进一步两两比较结果显示,高风险组孕妇NIPS胎儿阳性率为1.65%(377/22 852),显著高于低风险组的1.13%(265/23 539),并且差异有统计学意义(χ2=23.325,P<0.001)。但是,高、低风险组分别和中风险组的NIPS结果胎儿阳性率分别比较,差异均无统计学意义(χ2=2.830、1.126,P=0.090、0.289)。此外,高风险组中,血清学筛查高风险人群的NIPS胎儿阳性率(1.93%, 63/3 271)显著高于不良孕产史/家族史者(1.14%, 27/2 365),并且差异有统计学意义(χ2=5.374,P=0.020),但高龄孕妇分别与血清学筛查高风险者和不良孕产史/家族史者的阳性率比较,差异均无统计学意义(χ2=1.098、3.638,P=0.295、0.056)。3组孕妇NIPS筛查情况比较,见表1。
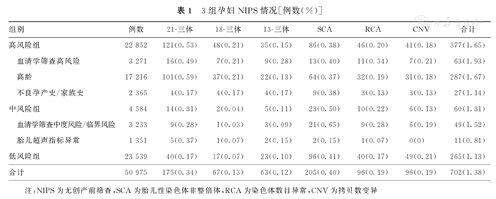
3组孕妇NIPS情况[例数(%)]
3组孕妇NIPS情况[例数(%)]
| 组别 | 例数 | 21-三体 | 18-三体 | 13-三体 | SCA | RCA | CNV | 合计 | |
|---|---|---|---|---|---|---|---|---|---|
| 高风险组 | 22 852 | 121(0.53) | 48(0.21) | 35(0.15) | 86(0.38) | 46(0.20) | 41(0.18) | 377(1.65) | |
| 血清学筛查高风险 | 3 271 | 16(0.49) | 7(0.21) | 9(0.28) | 13(0.40) | 11(0.34) | 7(0.21) | 63(1.93) | |
| 高龄 | 17 216 | 101(0.59) | 37(0.21) | 22(0.13) | 64(0.37) | 32(0.19) | 31(0.18) | 287(1.67) | |
| 不良孕产史/家族史 | 2 365 | 4(0.17) | 4(0.17) | 4(0.17) | 9(0.38) | 3(0.13) | 3(0.13) | 27(1.14) | |
| 中风险组 | 4 584 | 14(0.31) | 2(0.04) | 5(0.11) | 23(0.50) | 10(0.22) | 6(0.13) | 60(1.31) | |
| 血清学筛查中度风险/临界风险 | 3 233 | 9(0.28) | 1(0.03) | 3(0.09) | 21(0.65) | 9(0.28) | 6(0.19) | 49(1.52) | |
| 胎儿超声指标异常 | 1 351 | 5(0.37) | 1(0.07) | 2(0.15) | 2(0.15) | 1(0.07) | 0(0) | 11(0.81) | |
| 低风险组 | 23 539 | 40(0.17) | 17(0.07) | 23(0.10) | 96(0.41) | 40(0.17) | 49(0.21) | 265(1.13) | |
| 合计 | 50 975 | 175(0.34) | 67(0.13) | 63(0.12) | 205(0.40) | 96(0.19) | 96(0.19) | 702(1.38) | |
注:NIPS为无创产前筛查,SCA为胎儿性染色体非整倍体,RCA为染色体数目异常,CNV为拷贝数变异
本研究702例NIPS筛查高风险者中,555例孕妇(79.06%)进行介入性产前诊断染色体核型分析和(或)CMA,93例(16.76%)拒绝进行介入性产前诊断,42例(7.57%)因为流产或已引产而未进行介入性产前诊断,12例失访(1.71%)。555例胎儿中,最终确诊为271例。介入性产前诊断对于21-、18-、13-TS等常见CAA及不同类型SCA的检测效力较高,其敏感度均为100.00%,特异度均达到99.9%以上。此外,NIPS筛查低风险的50 273例均呈阴性(表2)。
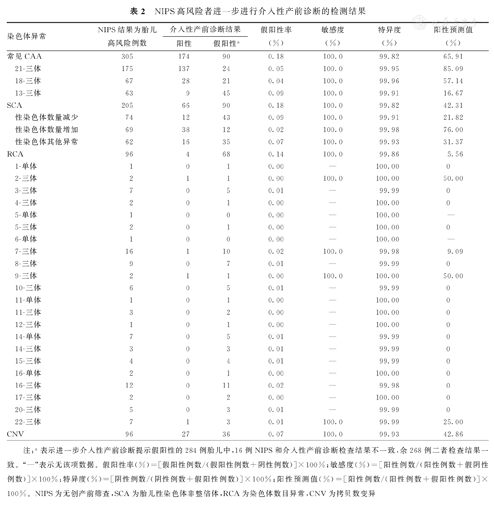
NIPS高风险者进一步进行介入性产前诊断的检测结果
NIPS高风险者进一步进行介入性产前诊断的检测结果
| 染色体异常 | NIPS结果为胎儿高风险例数 | 介入性产前诊断结果 | 假阳性率(%) | 敏感度(%) | 特异度(%) | 阳性预测值(%) | ||
|---|---|---|---|---|---|---|---|---|
| 阳性 | 假阳性a | |||||||
| 常见CAA | 305 | 174 | 90 | 0.18 | 100.0 | 99.82 | 65.91 | |
| 21-三体 | 175 | 137 | 24 | 0.05 | 100.0 | 99.95 | 85.09 | |
| 18-三体 | 67 | 28 | 21 | 0.04 | 100.0 | 99.96 | 57.14 | |
| 13-三体 | 63 | 9 | 45 | 0.09 | 100.0 | 99.91 | 16.67 | |
| SCA | 205 | 66 | 90 | 0.18 | 100.0 | 99.82 | 42.31 | |
| 性染色体数量减少 | 74 | 12 | 43 | 0.09 | 100.0 | 99.91 | 21.82 | |
| 性染色体数量增加 | 69 | 38 | 12 | 0.02 | 100.0 | 99.98 | 76.00 | |
| 性染色体其他异常 | 62 | 16 | 35 | 0.07 | 100.0 | 99.93 | 31.37 | |
| RCA | 96 | 4 | 68 | 0.14 | 100.0 | 99.86 | 5.56 | |
| 1-单体 | 1 | 0 | 1 | 0.00 | — | 100.00 | 0 | |
| 2-三体 | 2 | 1 | 1 | 0.00 | 100.0 | 100.00 | 50.00 | |
| 3-三体 | 7 | 0 | 5 | 0.01 | — | 99.99 | 0 | |
| 4-三体 | 2 | 0 | 1 | 0.00 | — | 100.00 | 0 | |
| 5-单体 | 1 | 0 | 0 | 0.00 | — | 100.00 | — | |
| 5-三体 | 2 | 0 | 1 | 0.00 | — | 100.00 | 0 | |
| 6-单体 | 1 | 0 | 0 | 0.00 | — | 100.00 | — | |
| 7-三体 | 16 | 1 | 10 | 0.02 | 100.0 | 99.98 | 9.09 | |
| 8-三体 | 9 | 0 | 7 | 0.01 | — | 99.99 | 0 | |
| 9-三体 | 2 | 1 | 1 | 0.00 | 100.0 | 100.00 | 50.00 | |
| 10-三体 | 6 | 0 | 5 | 0.01 | — | 99.99 | 0 | |
| 11-单体 | 1 | 0 | 1 | 0.00 | — | 100.00 | 0 | |
| 11-三体 | 3 | 0 | 2 | 0.00 | — | 100.00 | 0 | |
| 12-三体 | 1 | 0 | 1 | 0.00 | — | 100.00 | 0 | |
| 14-单体 | 7 | 0 | 5 | 0.01 | — | 99.99 | 0 | |
| 14-三体 | 3 | 0 | 3 | 0.01 | — | 99.99 | 0 | |
| 15-三体 | 4 | 0 | 4 | 0.01 | — | 99.99 | 0 | |
| 16-单体 | 2 | 0 | 1 | 0.00 | — | 100.00 | 0 | |
| 16-三体 | 12 | 0 | 11 | 0.02 | — | 99.98 | 0 | |
| 17-三体 | 2 | 0 | 2 | 0.00 | — | 100.00 | 0 | |
| 20-三体 | 5 | 0 | 3 | 0.01 | — | 99.99 | 0 | |
| 22-三体 | 7 | 1 | 3 | 0.01 | 100.0 | 99.99 | 25.00 | |
| CNV | 96 | 27 | 36 | 0.07 | 100.0 | 99.93 | 42.86 | |
注:a表示进一步介入性产前诊断提示假阳性的284例胎儿中,16例NIPS和介入性产前诊断检查结果不一致,余268例二者检查结果一致。"—"表示无该项数据。假阳性率(%)=[假阳性例数/(假阳性例数+阴性例数)]×100%;敏感度(%)=[阳性例数/(阳性例数+假阴性例数)]×100%;特异度(%)=[阴性例数/(阴性例数+假阳性例数)]×100%;阳性预测值(%)=[阳性例数/(阳性例数+假阳性例数)]×100%。NIPS为无创产前筛查,SCA为胎儿性染色体非整倍体,RCA为染色体数目异常,CNV为拷贝数变异
555例胎儿进行介入性产前诊断的具体检测结果如下:①264例常见CAA胎儿中,常见CAA共确诊174例,总体阳性预测值为65.91%(174/264),4例(2.30%,4/174)确诊为嵌合体(3例21-三体嵌合和1例18-三体嵌合)。21、18、13-TS高风险胎儿分别为161、49、54例,对其染色体核型分析和(或)CMA结果显示,确诊21、18、13-TS高风险分别为137、28、9例,阳性预测值分别为85.09%(137/161)、57.14%(28/49)、16.67%(9/54)。②对于胎儿SCA进行介入性产前诊断,共确诊66例,性染色体数目减少、性染色体数目增加和性染色体其他异常分别确诊12、38、16例,其阳性预测值分别为21.82%(12/55)、76.00%(38/50)和31.37%(16/51)。胎儿SCA的性染色体数目减少和性染色体数目增加的阳性预测值比较,差异有统计学意义(χ2=30.825,P<0.001)。同时,66例SCA胎儿中,13例为嵌合体(19.7%),除1例为47,XXY[38]/46,XY[12]外,其余12例均为45,X与其他染色体核型嵌合体。对常见CAA(21-、18-、13-TS)总体阳性预测值(65.91%,174/264),显著高于对胎儿SCA的42.31%(66/156),并且差异有统计学意义(χ2=22.303,P<0.001)。③ NIPS筛查出96例胎儿RCA高风险者,RCA阳性率为0.19%(96/50 975),72例接受介入性产前诊断胎儿中,4例胎儿结果RCA呈阳性,RCA阳性预测值为5.56%(4/72)。这4例胎儿的染色体检测结果均为嵌合体,分别为47,XN,+2[4]/46,XN[48];47,XN,+7[4]/46,XN[46];47,XN,+9[5]/46,XN[45];47,XN,+22[4]/46,XN[36]。④NIPS共筛查出CNV高风险为96例,CNV阳性率为0.19%(96/50 975)。经介入性产前诊断后,CNV呈阳性为27例,CNV阳性预测值为42.86%。
284例介入性产前诊断假阳性胎儿中,16例的介入性产前诊断结果与NIPS不一致,见表3。其中,4例(No.1~4) 21-三体高风险胎儿中,CMA发现3例(No.1~3)为染色体部分片段CNV,染色体核型分析发现1例(No.4)为21号染色体复杂的结构变异;1例(No.5)18-三体高风险胎儿中,染色体核型分析结果提示为21-三体;2例(No.6~7)13-三体高风险胎儿中,染色体核型分析发现1例(No.6)为染色体结构变异的嵌合体,另1例(No.7)经CMA检测为CNV;3例(No.8~10) SCA高风险和2例(No.11~12) RCA高风险胎儿中,CMA检测到其他染色体的CNV;4例(No.11~12)CNV高风险胎儿中,经CMA检测发现,1例为47,XYY,3例为其他染色体的CNV异常。16例胎儿的染色体异常结果中,9例胎儿的染色体异常为致病性或可能致病,7例胎儿的CNV被评估为临床意义不明。
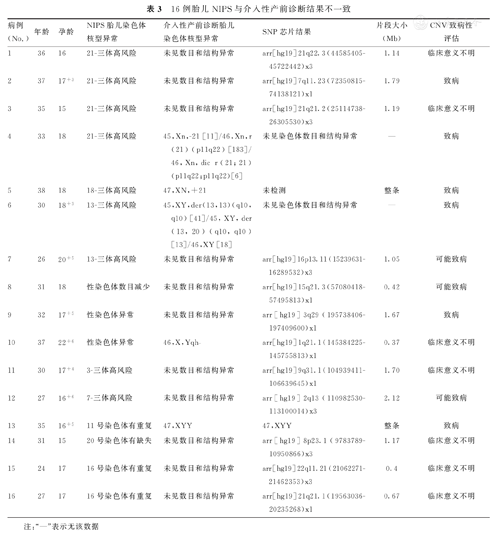
16例胎儿NIPS与介入性产前诊断结果不一致
16例胎儿NIPS与介入性产前诊断结果不一致
| 病例(No.) | 年龄 | 孕龄 | NIPS胎儿染色体核型异常 | 介入性产前诊断胎儿染色体核型异常 | SNP芯片结果 | 片段大小(Mb) | CNV致病性评估 |
|---|---|---|---|---|---|---|---|
| 1 | 36 | 16 | 21-三体高风险 | 未见数目和结构异常 | arr[hg19]21q22.3(44585405-45722442)x3 | 1.14 | 临床意义不明 |
| 2 | 37 | 17+3 | 21-三体高风险 | 未见数目和结构异常 | arr[hg19]7q11.23(72350815-74138121)x1 | 1.79 | 致病 |
| 3 | 35 | 15 | 21-三体高风险 | 未见数目和结构异常 | arr[hg19]21q21.2(25114738-26305530)x3 | 1.19 | 临床意义不明 |
| 4 | 33 | 18 | 21-三体高风险 | 45,Xn,-21[11]/46,Xn,r(21)(p11q22)[183]/46,Xn,dic r(21;21)(p11q22;p11q22)[6] | 未见染色体数目和结构异常 | — | 致病 |
| 5 | 38 | 18 | 18-三体高风险 | 47,XN,+21 | 未检测 | 整条 | 致病 |
| 6 | 30 | 18+3 | 13-三体高风险 | 45,XY,der(13,13)(q10,q10)[41]/45,XY,der(13,20)(q10,q10)[13]/46,XY[18] | 未见染色体数目和结构异常 | — | 致病 |
| 7 | 26 | 20+5 | 13-三体高风险 | 未见数目和结构异常 | arr[hg19]16p13.11(15239631-16289532)x3 | 1.05 | 可能致病 |
| 8 | 31 | 18 | 性染色体数目减少 | 未见数目和结构异常 | arr[hg19]15q21.3(57080418-57495813)x1 | 0.42 | 可能致病 |
| 9 | 32 | 17+5 | 性染色体异常 | 未见数目和结构异常 | arr[hg19]3q29(195738406-197409600)x1 | 1.67 | 致病 |
| 10 | 37 | 22+6 | 性染色体异常 | 46,X,Yqh- | arr[hg19]1q21.1(145384225-145755813)x1 | 0.37 | 临床意义不明 |
| 11 | 30 | 17+4 | 3-三体高风险 | 未见数目和结构异常 | arr[hg19]9q31.1(104939411-106639645)x1 | 1.70 | 临床意义不明 |
| 12 | 27 | 16+6 | 7-三体高风险 | 未见数目和结构异常 | arr[hg19]2q13(110982530-113100014)x3 | 2.12 | 可能致病 |
| 13 | 35 | 16+5 | 11号染色体有重复 | 47,XYY | 47,XYY | 整条 | 致病 |
| 14 | 31 | 15 | 20号染色体有缺失 | 未见数目和结构异常 | arr[hg19]8p23.1(9783789-10950866)x3 | 1.17 | 临床意义不明 |
| 15 | 24 | 17 | 16号染色体有重复 | 未见数目和结构异常 | arr[hg19]22q11.21(21062271-21462353)x3 | 0.4 | 临床意义不明 |
| 16 | 27 | 17 | 16号染色体有重复 | 未见数目和结构异常 | arr[hg19]21q21.1(19563036-20235268)x1 | 0.67 | 临床意义不明 |
注:"—"表示无该数据
由于配子形成时发生减数分裂错误,以及有丝分裂错误,可引起胎儿CAA和结构变异等染色体异常,从而导致胚胎生长停滞、着床失败或早孕期自然流产,如果早孕期未得到介入性产前诊断,1.0%~1.7%染色体异常胎儿可继续生长发育至出生[11,12]。小于1岁的先天性畸形患儿中,约15%由染色体异常所致,约25%染色体异常胎儿死于围生期[13]。因此,减少该类患儿的出生,是降低出生缺陷发生率的关键。
NIPS作为一种"近似于诊断"的筛查方法,在21-、18-、13-三体中的筛查效率已得到临床医师和患者的认可[14]。本研究通过对广西地区50 975例孕妇进行NIPS结果显示,总阳性率为1.38%(702/50 975),与文献报道结果相似[15,16]。同时,NIPS对于常见CAA(21-、18-、13-三体)检测敏感度均达100%,特异度亦均>99.9%。本研究结果还发现,在由血清学筛查高风险、高龄及不良孕产史组成的高风险组中,其筛查阳性率高于低风险组和中风险组。因此,对于血清学筛查提示高风险、高龄和有不良孕产史的孕妇,采用NIPS进行检测,可减少介入性产前诊断,从而降低流产率和医疗资源浪费。同时,与介入性产前诊断相比,NIPS具有较高的敏感度和特异度,可作为介入性产前诊断过程中的有效辅助手段。
阳性预测值是指在筛查阳性人群中,真正患病者所占比例,是除敏感度和特异度外,评价筛查手段检测效力的重要指标之一[17]。本研究中,21-、18-TS的阳性预测值均较高,分别为85.09%(137/161)和57.14%(28/49),和既往文献报道结果相似[15,16,17],但是13-TS的阳性预测值略低(16.67%,9/54),可能是由于13-TS胎儿的受精卵容易形成胎儿自救机制,胎儿染色体核型正常,胎盘出现正常和异常的染色体嵌合体,即限制性胎盘嵌合体所致[18]。
尽管NIPS对于21-、18-、13-TS的筛查准确性得到广泛认可,但是对于胎儿SCA检出的临床价值以及是否应该将SCA作为目标检测疾病尚存争议。本研究对156例NIPS提示疑似SCA胎儿进行介入性产前诊断,异常者为66例,阳性预测值为42.31%(66/156)。本研究结果还发现,NIPS对于不同类型SCA检出的阳性预测值存在差异,对于性染色体增加的阳性预测值(76.00%)远高于性染色体减少(21.82%),并且差异有统计学意义(χ2=30.825,P<0.001),与之前的研究结果一致[16,19]。另外,66例SCA胎儿中,13例为性染色体嵌合体,嵌合体也是影响产前筛查和介入性产前诊断结果准确性的重要因素之一[20,21]。因此,对于NIPS提示为SCA胎儿进行染色体核型分析时,尽量采用荧光原位杂交或基于单核苷酸多态性(single nucleotide polymorphism,SNP)的CMA检测,排除性染色体嵌合体。目前,由于NIPS对于SCA筛查的假阳性率较高,国家药品监督管理局尚未将SCA筛查列入NIPS的目标疾病,但是因为SCA的发病率高达1/500[22],且随着样本量累积和NIPS技术的不断发展,未来SCA也许亦将成为NIPS,筛查的目标疾病之一。
本研究NIPS共筛查出96例RCA高风险孕妇,阳性检出率为0.19%(96/50 975),低于Pertile等[23]报道的0.43%,其可能的原因是本研究纳入孕妇不仅包含高风险人群,也纳入了低风险人群,导致阳性率有所下降。96例RCA高风险胎儿中,7号染色体三体所占比例最大,为16例,和其他研究结果一致[16,24],其中11例进行介入性产前诊断,仅1例确诊为7-三体的嵌合体(47,XN,+7[4]/46,XN[46])。其他RCA高风险胎儿经介入性产前诊断,只有2-、9-和22-三体中各1例为相应染色体的嵌合三体。本研究NIPS对于RCA的阳性预测值仅为5.56%(4/72),其可能的原因是:①除13、18和21号染色体外,其他常染色体数目异常往往导致胎儿早期自然流产;②限制性胎盘嵌合所致。鉴于RCA的阳性预测值较低,且嵌合型的染色体数目异常的临床表型无法评估,因此,美国医学遗传学和基因组学会(American College of Medical Genetics and Genomics,ACMG)不推荐将NIPS用于13、18和21号染色体以外的胎儿CAA的筛查,对于NIPS的非目标疾病,需通过超声密切监测胎儿情况,再考虑介入性产前诊断。
CNV在人类表型变异或疾病中具有重要作用,其在人群中发生率为0.1%~0.3%[24]。本研究纳入的50 975例样本中,NIPS共检出96例CNV高风险胎儿,阳性率为0.19%(96/50 975),与文献报道[25]相近。目前对于CNV的诊断方法主要包括染色体核型分析和CMA,但这2种方法都为侵入性操作,如绒毛膜绒毛取样、羊膜腔穿刺术、脐血取样等方法采集胎儿细胞进行基因分析,有导致流产、感染等并发症的风险,从而降低患者检测依从性。因此,NIPS在CNV检测中的可行性受到广泛关注和研究。本研究中,进行介入性产前诊断的63例CNV高风险孕妇中,异常为27例,NIPS对于CNV筛查的阳性预测值为42.86%,说明NIPS对胎儿基因组的缺失/重复的检测具有普遍和实用的价值,并且可纳入检测胎儿非整倍性无创性产前筛查方案。Liang等[26]进行的1项大样本量研究结果显示,如果对生物信息分析流程进行一定改进,可以显著提高NIPS对于胎儿CNV的检出率,使其成为胎儿CNV的一线筛查技术。
需注意的是,本研究中通过染色体核型分析和CMA对NIPS高风险胎儿进行介入性产前诊断,发现16例检测到非高风险目标染色体或区域的异常结果,其中9例被评估为可能致病性或致病性染色体异常。因此,在进行介入性产前诊断时,建议进行全基因组分析,而不只是分析NIPS提示高风险的目标区域[27,28,29,30]。此外,由于CMA不仅可检出CNV,也可检测单亲二倍体,还可检测一定比例嵌合体,因此建议尽可能选择基于SNP的基因芯片进行CMA[31,32]。
NIPS对于常见胎儿CAA的临床诊断价值较高,但也存在一定的假阳性率,需要进一步采用染色体核型分析和(或)CMA等介入性产前诊断手段确诊。对于性染色体单体、RCA和CNV,NIPS的筛查阳性预测值较低,但结合产前超声检查结果,可为遗传咨询和进一步介入性产前诊断提供依据。随着二代测序技术的进一步发展和检测成本的降低,NIPS可作为所有胎儿CAA和全基因组CNV介入性产前诊断的一种初步筛查方法,将会在早孕期应用更加广泛和有效。
所有作者均声明不存在利益冲突



























