
采用两样本孟德尔随机化方法(Mendelian randomization,MR)研究血清辅酶Q10(coenzyme Q10,CoQ10)水平对阿尔茨海默病(Alzheimer's disease,AD)及帕金森病(Parkinson's disease,PD)的因果关系。
对全基因关联研究(genome-wide association study,GWAS)的汇总数据进行分析。所采用的GWAS汇总数据均来源于欧洲人群。利用与血清CoQ10水平显著相关的单核苷酸多态性作为工具变量,分别用逆方差加权法、MR-Egger回归和加权中位数法行两样本孟德尔随机化分析,以比值比(odds ratio,OR)评价血清CoQ10与AD及PD的因果效应。
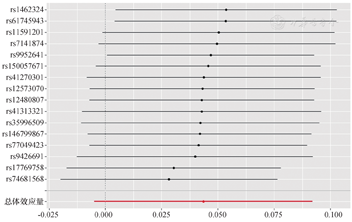
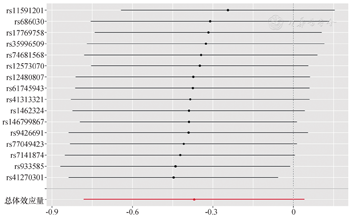
逆方差加权法结果显示血清CoQ10与AD(OR=1.140,95%CI:0.874~1.486,P=0.334)及PD(OR=0.691,95%CI:0.458~1.042,P=0.078)均不存在显著相关性。Q检验及MR-Egger回归结果表明不存在异质性及基因的水平多效性(P>0.05)。
从基因层面而言,目前MR研究不支持血清CoQ10与AD及PD存在显著的因果关联,但仍需要进一步的研究来更全面地评估它们之间的相关性。

本刊版权归人民卫生出版社有限公司所有。任何机构或个人欲转发本刊图文,请与本刊联系。凡未经授权而转载、摘编本刊文章者,本刊将保留追究其法律责任的权利。
随着全球老龄化趋势不断增加,阿尔茨海默病(Alzheimer's disease,AD)、帕金森病(Parkinson's disease,PD)等神经退行性疾病(neurodegenerative diseases,ND)因其高发病率、高死亡率已成为世界范围内最严重的公共卫生问题,带来极大的社会经济负担[1]。ND发病与年龄密切相关。随着大脑衰老,线粒体功能不良导致大量活性氧(reactive oxygen species,ROS)自由基的产生以及能量代谢异常,产生氧化应激,是ND发病的一个重要机制[2]。因此,研究者一直积极探索抗氧化剂对相关疾病的保护作用。辅酶Q10(coenzyme Q10,CoQ10)作为一种重要的天然抗氧化剂及自由基清除剂,是维持神经元、神经胶质细胞线粒体功能和能量代谢的重要物质[3]。相关研究结果显示CoQ10可以在AD、PD进展过程中增强机体抗氧化水平,维持线粒体功能,保护神经细胞[4,5]。但由于既往临床试验样本量小、随访周期短等问题,补充CoQ10是否对AD、PD患者存在保护作用仍有争议。此外,CoQ10也有潜力作为一种反应氧化应激状态的生物标志物来预测疾病风险[6,7]。因此,研究CoQ10与AD、PD的相关性对于疾病的治疗和预测均有价值。
孟德尔随机化(Mendelian randomization,MR)是近年来兴起的一种新的推断因果效应的方法。该方法以遗传变异作为工具变量,基于工具变量与暴露因素强关联探讨暴露与结局之间的因果关系[8]。本研究将通过两样本孟德尔随机化方法,探讨血清CoQ10水平与AD、PD的因果关联。
本研究采取两样本孟德尔随机化方法,以与血清CoQ10浓度相关的单核苷酸多态性(single nucleotide polymorphism,SNP)位点作为工具变量,探究血清CoQ10浓度与AD、PD发病的因果关系。
在本MR研究中,所选取的工作变量需满足以下3个假设:①遗传变异与血清CoQ10的浓度相关;②遗传变异不得与影响AD或PD的任何混杂因素相关,不得与暴露-结局通路上的混杂因素相关;③遗传变异只通过血清CoQ10影响结局[9](图1)。
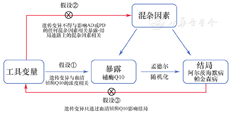

暴露方面,血清CoQ10的遗传数据来源一个全基因组关联研究(genome-wide association study,GWAS)的荟萃分析。该研究纳入两个基于德国人群展开的独立的横断面队列研究,受试者共1 300例。相关数据已公开发表,可在GWAS Catalog网站(https://www.ebi.ac.uk/gwas/home)上下载。
在AD患病相关的遗传变异选择方面,我们使用了来自国际阿尔茨海默病基因组学项目(International Genomics of Alzheimer's Project,IGAP)的GWAS研究meta分析[10]。该研究共纳入63 926受试者(21 982例AD病例和41 944例健康对照),全部基于欧洲人群。受试者通过尸检或应用临床诊断标准确诊AD。PD方面,本研究采用国际帕金森病遗传协会(International Parkinson's Disease Genomics Consortium,IPDGC)发表的大型GWAS荟萃分析[11]。该研究同样基于欧洲人口展开,共纳入482 730例受试者,其中包括33 674例PD病例和449 056例健康对照。为了避免由于人群分层现象发生偏倚,我们所选取的遗传数据全部来源于欧洲人口。
我们通过筛选GWAS数据,纳入与血清CoQ10显著相关的SNP。基于血清CoQ10水平荟萃分析结果,此次纳入相关性满足P<5×10-6的SNP作为工具变量[12];并且为了避免SNP存在连锁不平衡(linkage disequilibrium,LD)对分析结果产生影响,设置参数r2阈值为0.001,距离为10 000kb[13]。为了保证工具变量与内生变量间具有强关联,避免弱工具变量偏倚发生,我们分别计算了每一个SNP的R2,代表由工具变量SNP解释的变异所占的比例;F统计量,用于评估工具变量的强度[14,15]。
此外,为了纳入的SNP仅通过暴露与结局相关,我们通过PhenoScanner(V2)数据库筛选与SNP显著相关的表型,并通过既往发表的MR研究判断这些表型是否为AD或PD的混杂因素。若是,则将涉及的SNP剔除。最终纳入19个SNP进行筛选,相关SNP的基本特征见表1。
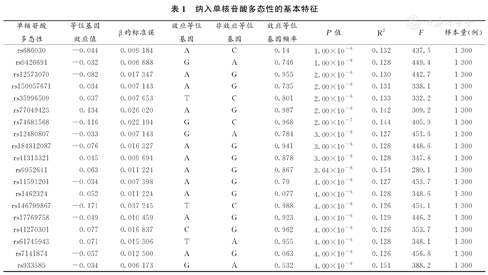
纳入单核苷酸多态性的基本特征
纳入单核苷酸多态性的基本特征
| 单核苷酸多态性 | 等位基因效应值 | β的标准误 | 效应等位基因 | 非效应等位基因 | 效应等位基因频率 | P值 | R2 | F | 样本量(例) |
|---|---|---|---|---|---|---|---|---|---|
| rs686030 | -0.044 | 0.009 184 | A | C | 0.14 | 1.00×10-6 | 0.132 | 437.5 | 1 300 |
| rs9426691 | -0.032 | 0.006 888 | G | A | 0.746 | 1.00×10-8 | 0.128 | 449.4 | 1 300 |
| rs12573070 | -0.082 | 0.017 347 | A | G | 0.955 | 2.00×10-6 | 0.130 | 442.7 | 1 300 |
| rs150057671 | 0.034 | 0.007 143 | A | G | 0.735 | 2.00×10-6 | 0.131 | 338.1 | 1 300 |
| rs35996509 | 0.037 | 0.007 653 | T | C | 0.801 | 2.00×10-6 | 0.133 | 332.2 | 1 300 |
| rs77049423 | 0.134 | 0.026 020 | A | G | 0.987 | 2.00×10-6 | 0.142 | 309.2 | 1 300 |
| rs74681568 | -0.116 | 0.022 194 | G | C | 0.968 | 2.00×10-7 | 0.144 | 405.9 | 1 300 |
| rs12480807 | -0.033 | 0.007 143 | G | A | 0.784 | 3.00×10-6 | 0.127 | 451.6 | 1 300 |
| rs184812087 | -0.076 | 0.016 327 | A | G | 0.941 | 3.00×10-6 | 0.128 | 448.6 | 1 300 |
| rs41313321 | 0.045 | 0.009 694 | A | G | 0.878 | 3.00×10-6 | 0.128 | 347.8 | 1 300 |
| rs9952641 | 0.063 | 0.011 224 | A | G | 0.867 | 3.64×10-8 | 0.154 | 280.1 | 1 300 |
| rs11591201 | -0.034 | 0.007 398 | A | G | 0.79 | 4.00×10-6 | 0.127 | 453.7 | 1 300 |
| rs1462324 | 0.052 | 0.011 224 | A | G | 0.077 | 4.00×10-6 | 0.128 | 348.6 | 1 300 |
| rs146799867 | -0.171 | 0.037 245 | T | C | 0.988 | 4.00×10-6 | 0.126 | 454.1 | 1 300 |
| rs17769758 | -0.049 | 0.010 459 | A | G | 0.923 | 4.00×10-6 | 0.129 | 446.2 | 1 300 |
| rs41270301 | 0.077 | 0.016 837 | C | G | 0.962 | 4.00×10-6 | 0.126 | 353.7 | 1 300 |
| rs61745943 | 0.071 | 0.015 306 | T | A | 0.955 | 4.00×10-6 | 0.128 | 348.1 | 1 300 |
| rs7141874 | -0.057 | 0.012 500 | A | G | 0.063 | 4.00×10-6 | 0.126 | 456.8 | 1 300 |
| rs933585 | -0.034 | 0.006 173 | G | A | 0.532 | 4.00×10-6 | 0.151 | 388.2 | 1 300 |
本MR研究主要采用逆方差加权法(inverse-variance weighted,IVW)探究血清CoQ10浓度与PD、AD之间的因果关系。同时为了保证统计结果的鲁棒性,我们同时采用加权中值估计法(weighted median estimator,WME)以及基于Egger回归的孟德尔随机化法(Mendelian randomization-egger regression,MR-Egger)对统计结果行敏感性分析。IVW被认为是MR汇总数据的标准方法[16]。该方法使用Wald比值法对每个纳入的工具SNP进行因果效应估计值,然后进行加权汇总分析[17]。加权中值估计法仅要求至少50%由遗传变异贡献的权重是有效的即可进行统计计算[18]。MR-Egger回归则可发现并校正多效性问题,其要求所纳入的工具变量满足InSIDE假设(instrument strength independent of direct effect,InSIDE),即假设工具-暴露和工具-结果的关联是独立的[19]。
本MR分析以比值比(odds ratio,OR)为效应值,取95%可信区间(confidence interval,CI)。当P<0.05时认为差异有统计学意义。此外,我们基于α=0.05,应用mRND(https://shiny.cnsgeno-mics.com/mRnd/)在线工具计算分别MR分析的统计效力,以验证结果的可靠性。
敏感性方面,本研究通过IVW及MR-Egger回归分别计算Cochran's Q统计量,若P>0.05则代表没有明显异质性。同时,我们还通过留一法逐个剔除纳入SNP,观察是否对分析结果产生影响,并绘制森林图,若剔除某个SNP后,得到P>0.05则认为SNP不会对结果造成显著影响[17]。多效性方面,我们同时采用MR-Egger回归的截距项及孟德尔随机多效性残差和及异常值(Mendelian randomization pleiotropy residual sum and outlier,MR-PRESSO)检验纳入SNP的水平多效性。在MR-Egger回归中,若截距趋于0,则可认为不存在水平多效性。在MR-PRESSO检验中,不仅可以计算出所纳入工具变量的水平程度,还可将异常值剔除后计算暴露与结局间的效应量,并对校正前后的结果进行检验[20]。
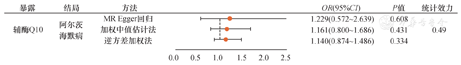
经PhenoScanner数据库筛选,我们发现rs686030与AD混杂因素高密度脂蛋白显著相关(P=4.29×10-27),予以剔除[21]。rs184812087存在连锁不平衡,rs41270301、rs61745943、rs74681568作为具有中等等位频率的回文序列在行MR分析时删除。最终纳入14个SNP行MR分析。IVW结果显示血清CoQ10浓度与AD患病风险无显著相关性(OR=1.140,95%CI:0.874~1.486,P=0.334)。WME和MR-Egger结果也显示血清CoQ10浓度与AD发病无关(P>0.05)。具体结果见图2。

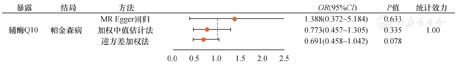
经PhenoScanner数据库检索,我们未发现SNP与PD混杂因素相关。rs41270301、rs150057671、rs9952641因具有中等等位频率的回文序列在行统计分析时予以剔除,最终纳入16个SNP。在血清CoQ10浓度与PD发病的MR分析中,所纳入的3种MR计算模型均显示二者没有相关性(P>0.05)。IVW方式示OR=0.691,95%CI:0.458~1.042,P=0.078。具体结果见图3。

敏感性分析方面,经Cochran's Q检验,各工具变量之间未见显著异质性(P>0.05)。留一法显示在分别剔除所纳入的SNP后,统计结果无显著差异(P>0.05)。具体结果详见图4、图5。多效性方面,MR-Egger回归示各组截距接近0,且P>0.05。MR-PRESSO检验亦未发现纳入的SNP对于AD及PD存在潜在多效性或存在异常值,具体结果见表2。


MR-Egger及MR-PRESSO多效性检验结果
MR-Egger及MR-PRESSO多效性检验结果
| 暴露 | 结局 | MR-Egger | MR-PRESSO | |||
|---|---|---|---|---|---|---|
| 截距 | 标准误 | P值 | RSSobs | P值 | ||
| 辅酶Q10 | 阿尔茨海默病 | -0.003 | 0.016 | 0.841 | 13.756 | 0.731 |
| 帕金森病 | -0.031 | 0.029 | 0.293 | 19.501 | 0.322 | |
注:RSSobs,MR-PRESSO globe检验。
探究暴露因素与神经退行性疾病之间的因果关系是疾病预防、早期诊断和改善治疗的关键。虽然随机对照试验(randomized controlled trial,RCT)是一种观察因果关系的理想手段,但受限于伦理和实际条件,标准RCT的开展较为困难。观察性研究也可用于观察因果关系,但容易受到潜在混杂和反向因果关系的影响,很难被认作是强有力的因果证据[22]。基因遗传学的快速发展,促使MR在医学研究中广泛应用。由于配子形成时遵循随机分配的孟德尔遗传定律,基因与结局的关联关系不会受到环境、后天疾病等混杂因素的影响,MR被认为是一种天然的RCT研究。此外,随着生物信息技术的发展,遗传变异的测量精度不断提高,这也大大降低了研究中由于测量误差可能带来的偏倚[23]。因此,使用遗传工具作为替代物来评估暴露与结局之间的关联,可以产生较传统观察性研究更可靠的因果证据[24]。本次MR研究发现,血清CoQ10水平与AD及PD发病之间不存在显著相关性。该结果在IVW、加权中值法和MR-Egger回归中均保持一致。本研究纳入的工具变量均经PhenoScanner数据库筛选,其F统计量均大于10,所采用的结局数据来源于3个大型GWAS研究,工具变量间未见明显的异质性或多效性,上述条件保证了该MR研究结果的稳健性。
Bustos等[25]测定了44例AD患者的循环CoQ10水平,并经胆固醇水平校正,发现其与正常对照组水平基本相同。但由于所设计的临床研究样本量较小,结论仍有待进一步验证。一项meta分析显示,PD患者的血清或血浆CoQ10水平相比对照组无明显差异[26]。但由于所纳入的研究设计和对象均有较大差异,研究间异质性明显,因此该meta分析结果可靠性有待加强。本两样本MR从遗传层面提供了相关证据,支持了血清CoQ10浓度与AD、PD发病无明显相关性。因此,以血清CoQ10作为AD、PD相关生物标志物,预测疾病进展的应用效果有限。临床治疗方面,许多前期研究与本研究结果一致。Galasko等[27]纳入70例轻度至中度AD患者口服CoQ10治疗16周,β-淀粉样蛋白和tau蛋白水平无显著改善,且因随访时间较短,研究没有汇报认知功能变化。PD方面,一项RCT研究发现早期PD患者补充CoQ10对于延缓病情进展无明显改善[28]。究其原因,可能是由于CoQ10自身水溶性差,生物利用度有限,不能透过血脑屏障,从而难以修复脑内的氧化应激损伤[29]。有研究者通过将CoQ10与亲水胶束结合制备出水溶性CoQ10剂型,其生物利用度较传统剂型显著提升,并在动物实验中表现出对AD、PD病理改变具有一定的保护作用[30,31,32]。但由于相关临床研究较少,水溶性CoQ10能否通过血脑屏障、有效改善脑内代谢状态仍不明确。未来还需要多中心、大样本的临床试验证明其治疗AD、PD的有效性与安全性。
本研究也存在一定的局限性。CoQ10在人体内的分布并不均匀,更多的CoQ10集中在氧化还原反应更频繁的器官,如大脑和肺[33]。因此外周CoQ10水平不能完全反映大脑的代谢状态。有研究指出脑脊液中的CoQ10浓度相比于血清,能够更直观地反映大脑内的状态[34]。此外,本次暴露仅探讨血清总CoQ10的水平,但CoQ10在体内有两种存在形式,分别为还原型(泛醌)和氧化型(泛醇)。正常情况下,CoQ10在这两种形式中保持动态平衡[35]。但是由于AD、PD患者体内持续处于氧化应激损伤状态,CoQ10的氧化还原状态失衡,有研究指出PD患者氧化型CoQ10/总CoQ10水平上升,代表其体内的氧化应激水平增高[36]。因此,探究泛醌或泛醇水平与AD、PD之间的因果效应可能具有更强的临床指导意义。但未来还需要更多的大样本MR研究和临床试验论证其因果联系。
综上,从基因层面而言,血清CoQ10水平与AD及PD无明显因果关联。利用其作为预测AD、PD的生物标志物可能效果不佳。未来仍需开展规范化、大样本的临床试验及相关MR研究,深入探讨CoQ10在大脑局部的代谢水平和其氧化还原状态改变对AD、PD的预防及治疗的潜在效果和临床意义。



























