
版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
慢性淋巴细胞白血病(CLL)是西方国家成年人群中最常见的白血病类型,其特点是外周血成熟CD5阳性B淋巴细胞扩增[1]。具有不同遗传学特征的CLL患者的病程有明显异质性。
近年来,多种生物学指标被证实可影响CLL预后[2]。其中,免疫球蛋白重链可变区(IGHV)基因的突变状态是与CLL预后最为相关的预后因素之一[3,4]。通过荧光原位杂交(FISH)检测到的重现性遗传学异常,如染色体11q和17p缺失,已用于定义患者危险度分组[5,6]。但少有研究揭示IGHV基因突变状态与细胞遗传学之间的相关性[7,8]。中国CLL发病率低于西方国家,且发病年龄较小,侵袭程度更高,某些与预后相关的基因突变,如ATM、SF3B1、NOTCH1、MYD88和TP53等,其发生率在东西方国家中存在差异[9],提示中国CLL患者的疾病特征有可能与其独特的遗传学背景相关。本研究对多中心数据进行归纳总结,旨在进一步探索中国CLL患者IGHV基因突变特征及与预后的相关性。
回顾性分析湖南省及天津市多家中心的CLL患者资料。所有患者均签署知情同意书。本研究获中南大学湘雅二医院伦理委员会批准,伦理批号为(2021)伦审【临研】第(K067)号。诊断基于患者临床特征及实验室检查,符合国际CLL工作组指南所发布的诊断标准[10]。
应用流式细胞术检测免疫表型,包括CD5,CD23、FMC7、CD22、CD25、CD19、CD20、CD10、CD11c及膜免疫球蛋白κ轻链、λ轻链等抗原表达。
行骨髓FISH检测细胞遗传学异常,包括11q22.3(ATM)缺失、+12q15(MDM2)、13q14(RB1)缺失、17p13(TP53)缺失及CCND1/IGH易位。10例具有正常核型和非血液系统恶性疾病的患者作为正常对照。阈值定义为正常对照的平均值+3×标准差,各探针阳性阈值如下:+12:7.5%,TP53缺失:5.0%,RB1或ATM缺失:6.5%,IgH易位:4.5%。
应用多重PCR法检测CLL患者IGHV片段使用情况及突变状态[11,12]。将免疫球蛋白重链(IgH)序列同源性<98%定义为体细胞突变,而IgH序列同源性≥98%定义为未发生体细胞突变[13]。
通过查阅患者电子病历及纸质病历确认患者住院治疗情况,对患者进行电话随访,随访时间截至2020年3月。治疗间隔时间(TTFT)定义为确诊之日起至达治疗指征开始首次治疗、死亡或末次随访(尚未达到治疗指征患者)的时间。
采用SPSS 19.0软件进行统计学分析。计数资料的组间比较应用χ2检验或Fisher精确检验,计量资料的组间比较采用Mann-Whitney非参数检验。采用Kaplan-Meier法及Log-rank检验计算预期TTFT并绘制生存曲线。P<0.05为差异有统计学意义。
287例患者中,男204例,女83例。中位年龄为58(26~86)岁。伴有淋巴结肿大的患者占74.2%,伴肝、脾肿大的患者分别占6.4%和43.5%。中位淋巴细胞绝对计数为21.75(0.6~367.61)×109/L,中位HGB水平为126(36~177)g/L,中位PLT为148(9~370)×109/L。26%的患者乳酸脱氢酶(LDH)水平升高,87.6%的患者伴β2-微球蛋白(β2-MG)水平升高。在行FISH及基因突变检测的232例患者中,最常见的遗传学异常为染色体13q缺失(23.3%),其后依次为12号染色体三体(22.9%)、染色体17p缺失/TP53基因突变(15.8%)、染色体11q缺失(14.9%)、SF3B1突变(7.7%)、MYD88L265P突变(7.6%)及BIRC3突变(5.3%)。初诊时仅有14.6%的患者处于Rai 0期,其余患者均处于进展期,Ⅰ、Ⅱ、Ⅲ、Ⅳ期占比分别为31.9%、21.7%、12.6%和19.3%。
287例患者中,IGHV基因为突变状态者占66.2%(190/287),突变中位值为5.6%(0~22.0%)。7个VH基因家族中最常见的VH基因家族为VH3(44.0%),其后依次为VH4(30.5%)、VH1(17.7%)、VH2(3.2%)、VH5(1.8%)、VH6(1.8%)、VH7(1.1%)。使用频率在5%以上的VH基因片段包括VH4-34(11.7%)、VH4-39 (8.5%)、VH3-30(8.2%)、VH3-23(6.7%)、VH1-69(6.0%)及VH3-70(5.3%)。使用VH3-21基因片段的患者占2.8%。7个基因家族中IGHV基因突变率呈不均一分布(图1A)。在使用VH3基因家族的患者中,78.2%的患者发生体细胞突变,突变率显著高于其他基因家族(P<0.001)。而在使用VH1基因家族的患者中,突变率仅为36%,显著低于其他基因家族(P<0.001)。进一步分析常见VH基因片段中的突变情况,突变率最高的基因片段为VH4-34(90.9%),显著高于其他基因片段(P=0.001)。VH4-39和VH1-69片段的突变率分别为41.7%和17.6%,显著低于其他基因片段(P=0.009、P<0.001)(图1B)。
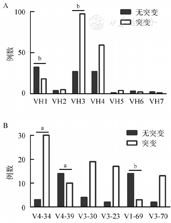

A:使用不同VH基因家族的慢性淋巴细胞白血病(CLL)患者中IGHV基因突变情况;B:使用不同VH基因片段的CLL患者中IGHV基因突变情况。aP<0.01;bP<0.001
将患者按照IGHV基因有无突变分为两组,两组间临床特征的比较见表1。IGHV为突变状态的患者相较于IGHV无突变者合并染色体13q缺失的比例更高(28.2%对13.2%,P=0.015)。而TP53基因缺失或突变比例在IGHV无突变组更常见(P<0.001)。IGHV无突变组患者中有28.6%伴有TP53基因缺失或突变,而突变组中仅9.6%的患者伴有TP53基因缺失或突变。两组患者的中位年龄、性别比、临床分期、血细胞计数、β2-MG水平、LDH水平之间差异均无统计学意义(P值均>0.05)。另外,使用VH4-34片段的患者中均不伴有TP53基因缺失或突变,显著低于使用其他基因片段的患者(0对15.8%,P=0.026)。
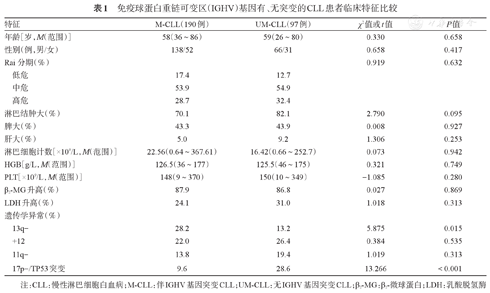
免疫球蛋白重链可变区(IGHV)基因有、无突变的CLL患者临床特征比较
免疫球蛋白重链可变区(IGHV)基因有、无突变的CLL患者临床特征比较
| 特征 | M-CLL(190例) | UM-CLL(97例) | χ2值或t值 | P值 | |
|---|---|---|---|---|---|
| 年龄[岁,M(范围)] | 58(36~86) | 59(26~80) | 0.330 | 0.658 | |
| 性别(例,男/女) | 138/52 | 66/31 | 0.658 | 0.417 | |
| Rai分期(%) | 0.919 | 0.632 | |||
| 低危 | 17.4 | 12.7 | |||
| 中危 | 53.9 | 54.9 | |||
| 高危 | 28.7 | 32.4 | |||
| 淋巴结肿大(%) | 70.1 | 82.1 | 2.790 | 0.095 | |
| 脾大(%) | 43.3 | 43.9 | 0.008 | 0.927 | |
| 肝大(%) | 5.0 | 9.2 | 1.306 | 0.253 | |
| 淋巴细胞计数[×109/L,M(范围)] | 22.56(0.64~367.61) | 16.42(0.66~252.7) | 0.073 | 0.942 | |
| HGB[g/L,M(范围)] | 126.5(36~177) | 125.5(46~175) | 0.321 | 0.749 | |
| PLT[×109/L,M(范围)] | 148(9~370) | 150(10~349) | -1.085 | 0.280 | |
| β2-MG升高(%) | 87.9 | 86.8 | 0.027 | 0.869 | |
| LDH升高(%) | 24.1 | 31.0 | 1.018 | 0.313 | |
| 遗传学异常(%) | |||||
| 13q- | 28.2 | 13.2 | 5.875 | 0.015 | |
| +12 | 22.0 | 26.4 | 0.384 | 0.535 | |
| 11q- | 13.8 | 19.4 | 1.019 | 0.313 | |
| 17p-/TP53突变 | 9.6 | 28.6 | 13.266 | <0.001 | |
注:CLL:慢性淋巴细胞白血病;M-CLL:伴IGHV基因突变CLL;UM-CLL:无IGHV基因突变CLL;β2-MG:β2-微球蛋白;LDH:乳酸脱氢酶
中位随访时间为21个月,234例患者中有159例达到治疗指征并启动治疗。Rai低危组(Rai 0期)患者中位TTFT为36.0个月,而中危组(Rai Ⅰ~Ⅱ期)和高危组(Rai Ⅲ~Ⅳ期)患者的中位TTFT分别为20.0个月和12.0个月(P=0.020)。同时,IGHV无突变(16.0个月对36.0个月,P=0.001)、TP53基因异常(19.0个月对25.0个月,P=0.025)和LDH水平升高(12.0个月对25.0个月,P=0.017)也是显著影响TTFT的预后不良因素。MYD88基因突变和SF3B1基因突变虽然在CLL患者中较常见,但对TTFT无显著影响。
进一步分析IGHV突变状态在不同临床分期亚组患者中的预后意义,在Rai低危组患者中,IGHV基因无突变者较突变者TTFT显著缩短[35.0(95% CI 15.6~54.4)个月对103.2 (95% CI 76.7~129.7)个月,P=0.002](图2A)。然而在Rai中高危组患者中,IGHV基因突变状态则对TTFT无显著影响[32.8(95% CI 22.6~43.1)个月对62.4(95% CI 46.3~78.5)个月,P=0.102](图2B)。
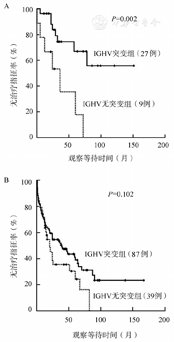

A:Rai危险度分层为低危组的CLL患者中IGHV基因突变状态对TTFT的影响;B:Rai危险度分层为中高危组的CLL患者中IGHV基因突变状态对TTFT的影响
有不少研究探讨了IGHV基因突变状态对CLL患者的预后的影响,但不同种族的CLL患者使用的VH基因片段存在差异。对于亚洲CLL人群的IGHV基因使用情况及对预后的影响还需要更深入的研究。在西方人群中,最常见的基因家族为VH1、VH3和VH4[14,15,16,17]。本研究中,VH3基因家族使用比例更高(44.0%),其次为VH4基因家族(30.5%)和VH1基因家族(17.7%),其余基因家族使用率较低,这与中国的另一研究相近[18]。VH1基因家族的使用率显著低于西方CLL人群(和美国[17]相比,P<0.001;和意大利[19]相比,P=0.030)。
VH基因片段的使用情况在东西方CLL人群中也存在差异。VH1-69是在西方CLL人群中最常见的基因片段,在美国CLL人群中为11%~18%[17, 20],在欧洲CLL人群中则为10.7%~11.4%[16,21,22],而在本研究中表达VH1-69的患者仅为6.0%。表达VH4-34、VH4-39、VH3-30及VH3-23的患者总和占本组患者的35%,而在英国及斯堪的纳维亚CLL人群中,这些基因片段的表达比例总和仅为约20%[16,22,23]。VH4-34为本组患者中最常见的基因片段,与其他东亚CLL人群的研究一致[9,24],但在中东则以VH3-7基因片段最为常见[25]。
在西方CLL人群中,IGHV突变率约为50%[26],本研究中IGHV基因突变比例更高(66.0%),与其他亚洲CLL的研究相近[18,27]。突变率最高的基因家族为VH3和VH4基因家族,与国外报道一致[17,28]。表达VH4-34基因片段的患者体细胞突变比例更高[16,27],提示患者预后更好。本研究中,表达VH4-34基因片段的患者体细胞突变比例高达90.9%,同时,无患者伴有染色体17p缺失或TP53基因突变。由于染色体17p缺失或TP53基因突变在IGHV基因无突变的CLL患者中更常见,我们推测这可能是表达VH4-34基因片段的患者中出现TP53基因异常的比例偏低的原因之一。
与VH4-34基因片段不同,大部分表达VH4-39和VH1-69基因片段的患者体细胞突变比例较低。以往认为,在亚洲CLL人群中,VH1-69基因片段的使用比例很低[18,24],但在本研究中,VH1-69的使用比例排在第5位。VH1-69的表达被认为与不良预后相关,在东西方人群中均有报道[29,30]。本研究中VH1-69的使用比例显著低于瑞典[22](P<0.001)和美国[17](P=0.040),而与其他国家的研究相比则差异无统计学意义。VH4-39基因片段发生体细胞突变比例偏低(41.7%),提示预后更差,本研究中VH4-39基因片段使用比例显著高于国外研究(和英国[16]相比,P=0.044;和意大利[19]相比,P=0.002)。
VH3基因家族中的多数基因片段突变比例较高,如VH3-30、VH3-23和VH3-7的突变率均超过80%。其他研究也曾报道VH3-7基因片段常见于IGHV基因有突变的CLL患者[16,17,23]。虽然VH3-21基因片段突变比例也较高,但有研究提示表达VH3-21基因片段者无论突变与否,均预后不良[31]。本研究中VH3-21基因片段的使用率与多数研究相近,但显著低于日本[24]和瑞典CLL人群[22]。
IGHV基因突变状态在CLL患者中的预后价值已非常明确,即伴IGHV突变的患者预后优于无突变者[2, 32]。本研究中IGHV突变状态仅在Rai低危组患者中显著影响TTFT,而在Rai中高危患者组中,IGHV突变状态则对TTFT无显著影响。表明进展期患者开始启动治疗的间隔时间不受IGHV突变状态的影响。
IGHV基因片段重排在不同地域和不同人种之间存在差异,即使在亚洲人群中,VH基因片段的使用情况也不尽相同,其潜在机制也许和IGHV胚系基因构成以及环境因素相关[21]。不同人群之间除表达不同的IGHV基因片段之外,互补决定区3(CDR3)的序列也存在差异,提示病原体或自身抗原在CLL的发病过程中扮演重要角色[33],遗憾的是由于本研究中患者CDR3序列信息不完善而无法分析。综上,本研究进一步揭示了中国CLL人群和西方CLL人群之间IGHV片段的表达差异,为CLL起源和发病机制提供更多理论依据。


























