
构建人工突变的噬菌体展示文库并与天然噬菌体展示文库序列对比,进而对人工突变的噬菌体展示文库的质量进行科学评价,为纳米抗体的进一步改造提供鉴。
对结合人卵泡刺激素受体(FSHR)的纳米抗体的抗原互补决定区3(CDR3)进行NNY定点饱和突变,合成VHH06-ΔCDR3随机突变DNA。将突变DNA序列连接到载体pMECS上,构建VHH06-ΔCDR3突变噬菌体展示文库。通过DNA测序和分析,比较该文库与FSHR免疫的VHH噬菌体展示文库的多样性和CDR3区的氨基酸分布。采用FSHR对文库进行6轮亲和筛选,测定筛选后克隆的富集程度。
按照NNY突变原则,合成了长度为16个氨基酸的CDR3随机突变基因库。成功构建了库容量为7.36×108 cfu/ml的VHH06-CDR3随机突变噬菌体展示文库。多克隆与单克隆phage酶联免疫吸附实验结果显示,经过6轮筛选,输出噬菌体与FSHR的结合明显得到富集,但获得的克隆与FSHR没有明显结合。
VHH06-ΔCDR3随机突变噬菌体展示文库尽管具有序列多样性,但由于CDR3缺乏功能多样性,导致其在亲和筛选中不利于获得目标抗体。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
噬菌体展示技术在抗体工程中发挥着重要作用,通过将外源多肽和蛋白编码序列整合到噬菌体PIII基因中,以融合蛋白的形式在噬菌体表面表达,并通过结合子富集亲和筛选,在体外即可获得抗原特异的抗体[1]。噬菌体展示技术的一大优势是不必采用天然来源的抗体基因序列构建文库,因此一方面可绕过动物免疫步骤,从而可节约宝贵的抗原蛋白,另一方面也可避开某些抗原(如毒蛋白)不能进行动物免疫的限制[2]。因此,噬菌体展示技术常被用来构建非免疫的天然来源抗体展示文库。此外,还可采用合成的方法获得非天然来源合成抗体基因展示文库,在对现有抗体进行工程改造以提高其亲和力和其他理化性质方面具有重要应用价值[3,4]。
天然抗体库中的克隆源自动物B淋巴细胞胚系基因VDJ重排[V(D)J recombinase]和进入外周淋巴器官生发中心的体细胞突变。在这些过程中,尽管大部分突变是随机的,但在抗原的选择压力下,最终成熟的B细胞产生的抗体基因,尤其是在V基因上具有特定序列组合[5]。Knappik等[6]分析了人抗体库的基因结构、序列组成、文库多样性和胚系基因的使用,发现7个VH和7个VL胚系基因家族涵盖了超过95%的人抗体库基因组成;并根据抗体可变区的结构设计,人工构建了第一个合成抗体库,获得亲和力较高的抗体。目前,人工合成的噬菌体展示文库主要采用单点突变(one-pot)及抗原互补决定区(complementarity determining region,CDR)随机突变等方法。尽管多数人工合成的噬菌体展示文库均有较高的库容(~109),显示出较高的序列多样性,但对其序列的功能多样性了解较少。对突变文库中的可变区序列,特别是对CDR3序列与天然抗体文库CDR3序列相关的系统分析,有助于设计合理的抗体文库,对于抗体工程改造和发现新的抗体均具有重要应用价值。
为了系统分析CDR3序列在突变文库和天然文库的功能多样性,本研究中采用源自骆驼的重链可变区抗体06(variable region of heavy-chain antibodies 06,VHH06)是一种小分子纳米抗体[7],其相对分子质量仅为15 ku,稳定性高、免疫原性低、更容易接近靶抗原的裂缝或隐藏抗原表位[8,9],且组织穿透能力强,易于表达等[10,11],可广泛应用于疾病的诊断与治疗和分子检测(如食品污染物检测)等领域[12,13]。此外,由于纳米抗体结构简单,只具有一个CDR3结构,在抗体结构与功能研究中是一个良好的模型分子。
卵泡刺激素受体(follicle-stimulating hormone receptor,FSHR)是一种重要的促性腺激素FSH结合的受体膜蛋白。近期研究结果表明,FSHR在多种肿瘤血管内皮细胞表面特异表达[14]。前期,笔者团队对新疆双峰驼免疫FSHR蛋白构建了VHH免疫文库,并从中筛选获得了一株具有一定FSHR结合能力的纳米抗体VHH06[15],但该抗体与FSHR结合的亲和力较差。本研究中,对VHH06的CDR3区进行突变构建VHH06-ΔCDR3突变噬菌体展示抗体库,对VHH06的CDR3序列进行分子进化,并以VHH06作为模型,分析CDR3区序列在突变文库和天然文库的功能多样性,为纳米抗体的基因工程改造奠定一定基础。
ExTaq酶、dNTP Mix、T4 DNA连接酶、pstⅠ和notⅠ限制性内切酶(日本TAKARA公司),质粒小提试剂盒、聚合酶链式反应(polymerase chain reaction,PCR)产物纯化试剂盒、DNA胶回收试剂盒购(美国Omega公司),DNA Marker(大连宝生物工程有限公司产品),胰蛋白胨、酵母提取物(美国Sigma公司),引物(生工生物工程股份有限公司),M13K07辅助噬菌体(美国NEB公司),TG1菌种、DH5α-pMECS噬菌粒(新疆生物资源基因工程重点实验室),酶联免疫吸附实验(enzyme-linked immuno sorbent assay,ELISA)试剂盒(美国Abcam公司)。
VHH06-ΔCDR3随机突变的目的基因由苏州金唯智生物科技有限公司合成,对VHH06的CDR3区,按照NNY(N=A/T/C/G,Y=C/T)的原则进行随机突变,形成人工突变抗体基因库。
噬菌粒载体pMECS和目的基因在37 ℃、4 h条件下,使用pstⅠ和notⅠ限制性内切酶同时进行双酶切;载体和基因按照质量比为1∶3,16 ℃过夜进行连接;将连接产物和感受态细胞按照一定比例混合均匀,转化大肠杆菌TG1,涂布于含50 μg/ml氨苄抗生素和2%葡萄糖的2YT固体培养基培养板上,37 ℃过夜培养。次日,计单克隆菌落数,计算库容量;随机挑选平板上的克隆,其中30个克隆进行菌液PCR,验证文库的重组率;另外30个克隆进行测序,了解文库的多样性。最后,使用细胞刮子刮下所有克隆,加入甘油,存于-80 ℃。
采用亲和淘洗技术,对VHH06-CDR3随机突变噬菌体展示文库进行特异性筛选(抗原FSHR)。筛选过程酶标板包被靶蛋白抗原FSHR234,每孔100 μl,质量浓度为30 μg/ml,4 ℃过夜。次日磷酸缓冲液(phosphate buffer saline,PBS)洗1次,2%牛血清白蛋白(bovine serum albumin,BSA)封闭(37 ℃、2 h)。PBS洗3次,每孔加入100 μl滴度为1×1011 cfu/ml的噬菌体文库(37 ℃、2 h)。PBST洗涤(洗涤次数随筛选次数增加),摸索得每孔加入100 μl Buffer E进行洗脱,立即用每孔6 μl的中和液调节至pH=7.0。洗脱液转移到新鲜对数期的TG1中,混匀,37 ℃下静止感染30 min。取10 μl用于测定噬菌体输出滴度,其余以2 000× g离心10 min,将剩菌体吹匀涂板,37 ℃培养过夜。次日用3 ml 2-YT液体培养基收集菌落,以质量分数为15%的甘油保存。
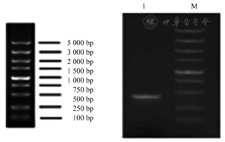
在VHH06原始序列基础之上,对其CDR3区的另外15个氨基酸[105位氨基酸保留为Ala丙氨酸(A)]进行定点饱和突变,得到纯化的DNA分子5 μg,片段大小约450 bp(图1)。

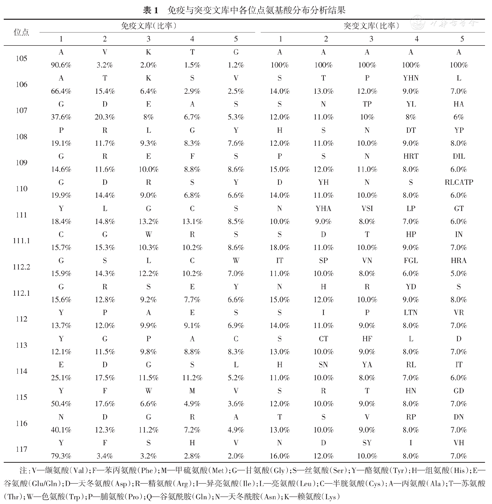
将合成的线性DNA分子亚克隆至载体pUC57形成重组子,随机挑选VHH06-ΔCDR3(NNY)16文库中100条克隆氨基酸进行测序,对其CDR3按照国际免疫遗传学数据库(international Im Muno Gene Tics database, IMGT)注释,分析105~117位点上各氨基酸的分布情况(图2)。为更好的评价VHH06-ΔCDR3(NNY)16突变文库的多样性,同时对天然纳米抗体文库中CDR3区有同样长度的52 226条高通量序列进行了各位点上氨基酸分布情况分析(图2A),并列出对应位点上出现频率最高的5个氨基酸(表1)。结果表明,对于天然免疫文库,其105~117位点上出现频率最高的氨基酸分别是Ala(90.6%)、Ala(66.4%)、Gly(37.6%)、Pro(19.1%)、Gly(14.6%)、Gly(19.9%)、Tyr(18.4%)、Cys(15.7%)、Gly(15.9%)、Gly(15.6%)、Tyr(13.7%)、Tyr(12.1%)、Glu(25.1%)、Tyr(50.4%)、Asn(40.1%)和Tyr(79.3%)。从氨基酸的分布可以看出,相对较保守的氨基酸为105、106位的Ala,107位的Gly,115、117位的Tyr以及116位的Asn,分布于CDR3区的两侧区域,这些氨基酸为疏水氨基酸和不带电荷的亲水性氨基酸。而对于VHH06-ΔCDR3突变文库,其105位保留为Ala;106~117位氨基酸分布较平均,位点无保守氨基酸。
免疫与突变文库中各位点氨基酸分布分析结果
免疫与突变文库中各位点氨基酸分布分析结果
| 位点 | 免疫文库(比率) | 突变文库(比率) | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 1 | 2 | 3 | 4 | 5 | |
| 105 | A | V | K | T | G | A | A | A | A | A |
| 90.6% | 3.2% | 2.0% | 1.5% | 1.2% | 100% | 100% | 100% | 100% | 100% | |
| 106 | A | T | K | S | V | S | T | P | YHN | L |
| 66.4% | 15.4% | 6.4% | 2.9% | 2.5% | 14.0% | 13.0% | 12.0% | 9.0% | 7.0% | |
| 107 | G | D | E | A | S | S | N | TP | YL | HA |
| 37.6% | 20.3% | 8% | 6.7% | 5.3% | 12.0% | 11.0% | 10% | 8% | 6% | |
| 108 | P | R | L | G | Y | H | S | N | DT | YP |
| 19.1% | 11.7% | 9.3% | 8.3% | 7.6% | 12.0% | 11.0% | 10.0% | 9.0% | 8.0% | |
| 109 | G | R | E | F | S | P | S | N | HRT | DIL |
| 14.6% | 11.6% | 10.0% | 8.8% | 8.6% | 15.0% | 12.0% | 11.0% | 8.0% | 6.0% | |
| 110 | G | D | R | S | Y | D | YH | N | S | RLCATP |
| 19.9% | 14.4% | 9.0% | 6.8% | 6.6% | 14.0% | 11.0% | 10.0% | 8.0% | 6.0% | |
| 111 | Y | L | G | C | S | N | YHA | VSI | LP | GT |
| 18.4% | 14.8% | 13.2% | 13.1% | 8.5% | 10.0% | 9.0% | 8.0% | 7.0% | 6.0% | |
| 111.1 | C | G | W | R | S | S | D | T | HP | IN |
| 15.7% | 15.3% | 10.3% | 10.2% | 8.6% | 18.0% | 11.0% | 10.0% | 9.0% | 7.0% | |
| 112.2 | G | S | L | C | W | IT | SP | VN | FGL | HRA |
| 15.9% | 14.3% | 12.2% | 10.2% | 7.0% | 11.0% | 10.0% | 8.0% | 6.0% | 5.0% | |
| 112.1 | G | R | S | E | Y | N | H | R | YD | S |
| 15.6% | 12.8% | 9.2% | 7.7% | 6.6% | 15.0% | 12.0% | 10.0% | 9.0% | 8.0% | |
| 112 | Y | P | A | E | S | S | I | P | LTN | VR |
| 13.7% | 12.0% | 9.9% | 9.1% | 6.9% | 14.0% | 11.0% | 9.0% | 8.0% | 7.0% | |
| 113 | Y | G | P | A | C | S | CT | HF | L | D |
| 12.1% | 11.5% | 9.8% | 8.8% | 8.3% | 13.0% | 10.0% | 9.0% | 8.0% | 7.0% | |
| 114 | E | D | G | S | L | H | SN | YA | RL | IT |
| 25.1% | 17.5% | 11.5% | 11.2% | 5.2% | 11.0% | 10.0% | 8.0% | 7.0% | 6.0% | |
| 115 | Y | F | W | M | V | S | R | T | HN | GD |
| 50.4% | 17.6% | 6.6% | 4.9% | 3.6% | 12.0% | 10.0% | 9.0% | 8.0% | 7.0% | |
| 116 | N | D | G | R | A | T | S | V | RP | DN |
| 40.1% | 12.3% | 11.2% | 7.2% | 4.9% | 13.0% | 10.0% | 9.0% | 8.0% | 7.0% | |
| 117 | Y | F | S | H | V | N | D | SY | I | VH |
| 79.3% | 3.4% | 3.2% | 2.8% | 2.0% | 16.0% | 12.0% | 10.0% | 8.0% | 7.0% | |
注:V—缬氨酸(Val);F—苯丙氨酸(Phe);M—甲硫氨酸(Met);G—甘氨酸(Gly);S—丝氨酸(Ser);Y—酪氨酸(Tyr);H—组氨酸(His);E—谷氨酸(Glu/Gln);D—天冬氨酸(Asp);R—精氨酸(Arg);I—异亮氨酸(Ile);L—亮氨酸(Leu);C—半胱氨酸(Cys);A—丙氨酸(Ala);T—苏氨酸(Thr);W—色氨酸(Trp);P—脯氨酸(Pro);Q—谷氨酰胺(Gln);N—天冬酰胺(Asn);K—赖氨酸(Lys)

CDR3—抗原互补决定区3;V—缬氨酸(Val);F—苯丙氨酸(Phe);M—甲硫氨酸(Met);G—甘氨酸(Gly);S—丝氨酸(Ser);Y—酪氨酸(Tyr);H—组氨酸(His);E—谷氨酸(Glu/Gln);D—天冬氨酸(Asp);R—精氨酸(Arg);I—异亮氨酸(Ile)L—亮氨酸(Leu);C—半胱氨酸(Cys);A—丙氨酸(Ala);T—苏氨酸(Thr);W—色氨酸(Trp);P—脯氨酸(Pro);Q—谷氨酰胺(Gln);N—天冬酰胺(Asn);K—赖氨酸(Lys)
载体pMECS定量结果为183.6 ng/μl,用pstⅠ、notⅠ限制性内切酶分别对载体和目的基因进行双酶切,对双酶切成功的质粒进行纯化回收,纯化回收定量结果为136.8 ng/μl。将纯化的载体和基因按照质量比1∶3进行连接,连接产物定量结果为42.4 ng/μl。将连接产物通过电转的方式(共6次)转入到制备好的TG1感受态细胞中,最终获得7.36×108数量的人工突变的噬菌体抗体文库。(图3)
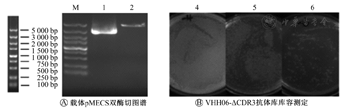

M—DNA maker;VHH06-ΔCDR3—可变区抗体06-抗原互补决定区3
随机挑选VHH06-ΔCDR3突变文库中的30个单克隆进行了菌落PCR,了解文库重组子阳性率。结果表明,30个克隆目的基因均在750 bp处正确插入,说明该抗体抗库的阳性率可近似达100%,可用于体外抗体的筛选工作。(图4)

M—DNA maker;CN—阴性对照
于VHH06-ΔCDR3合成抗体库中随机挑选30个克隆序列进行DNA测序。结果显示,有28条序列符合要求(表2),表明VHH06-ΔCDR3抗体库的正确率可达90%以上。将符合要求的28条序列通过DNAMAN软件进行比对,结果显示无重复序列,表明VHH06-ΔCDR3突变文库多样性良好,可采用多种抗原对其进行筛选。

VHH06-ΔCDR3库中符合要求的克隆序列
VHH06-ΔCDR3库中符合要求的克隆序列
| 序列 | CDR3氨基酸测序结果 | 序列 | CDR3氨基酸测序结果 |
|---|---|---|---|
| 1 | ALPPVAGCILLDYFTG | 15 | ATRYPHIACNRFPGCP |
| 2 | ATLSINNVNNLDRGSA | 16 | APGYNTSNFPRDARHP |
| 3 | ADLYHPCSCHYDSASY | 17 | AHPSHHVNVNHDNLYY |
| 4 | ANRNGLCNSHANHCLG | 18 | ANLIPPYTLFSSVTCA |
| 5 | AFGHSGSLPLGYHPPD | 19 | APLTCHHNGRIIHSPH |
| 6 | ATDASVPPFPRRFVGN | 20 | AYVFDHNYFYINSRDY |
| 7 | ADSPPDDNTRLILSRV | 21 | APHHLDTCDCISSDCA |
| 8 | AYLHGDVGVYGVLTCL | 22 | ADTSIDALHTGGPTRY |
| 9 | AGSNCTVYPCLTSTRH | 23 | ANCNIHITNYSYFIFD |
| 10 | AYDNTSFSPDTTSTPR | 24 | APDINHGSLPPLYRPT |
| 11 | AYNNYYDTITAFSNDA | 25 | ACNDLDSSPNSLSRNI |
| 12 | ATGARWPPLSPSAYLD | 26 | AYYRHNYDNRPIYPNP |
| 13 | AASYGTGSTNILSHPV | 27 | RTIHRATRPTSNTRP |
| 14 | ATAYSTVNTNNCIGHP | 28 | AISSPNRHLTICCDRN |
注:CDR3—抗原互补决定区;V—缬氨酸(Val);F—苯丙氨酸(Phe);M—甲硫氨酸(Met);G—甘氨酸(Gly);S—丝氨酸(Ser);Y—酪氨酸(Tyr);H—组氨酸(His);E—谷氨酸(Glu/Gln);D—天冬氨酸(Asp);R—精氨酸(Arg);I—异亮氨酸(Ile);L—亮氨酸(Leu);C—半胱氨酸(Cys);A—丙氨酸(Ala);T—苏氨酸(Thr);W—色氨酸(Trp);P—脯氨酸(Pro);Q—谷氨酰胺(Gln)N—天冬酰胺(Asn);K—赖氨酸(Lys)
采用FSHR作为抗原,辅助噬菌体M13K07,滴度为3.055×1013对突变文库进行了6轮亲和筛选。
筛选过程中,主要通过降低抗原包被浓度和增加洗涤次数2个方面的筛选压力迫使针对抗原FSHR的克隆得到富集。亲和筛选过程中每一轮均对所得噬菌体抗体库滴度进行测定,筛选结果如表3所示。
VHH06-ΔCDR3抗体库6轮筛选结果
VHH06-ΔCDR3抗体库6轮筛选结果
| 轮次 | 抗原质量浓度(μg/ml) | 洗涤次数(n) | 输入(cfu/ml) | 输出(cfu/ml) |
|---|---|---|---|---|
| 1 | 30 | 5 | 9.0×1012 | 3.9×107 |
| 2 | 20 | 10 | 4.6×1012 | 4.4×106 |
| 3 | 20 | 15 | 2.0×1012 | 5.0×106 |
| 4 | 10 | 15 | 1.2×1012 | 8.0×107 |
| 5 | 10 | 20 | 9.4×1012 | 2.0×108 |
| 6 | 5 | 20 | 1.5×1012 | 6.5×108 |
VHH06-ΔCDR3合成文库筛选完成之后,为了解合成文库的富集状况,进行多克隆噬菌体ELISA。抗原(FSHR)包被质量浓度为1 μg/ml,以BSA作为阴性对照。将先前筛选得到的噬菌体库均稀释至1×1012 cfu/ml并进行孵育。以Anti-M13KO7-HRP作为二抗(稀释度为1∶5 000),检测450 nm处的吸光度值。结果表明,噬菌体在第4轮筛选后逐步开始积累,且在第6轮筛选后积累率最高。(图5)
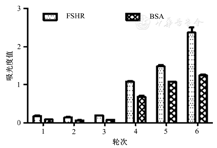

FSHR—卵泡刺激素受体;BSA—牛血清蛋白
随机挑选第6轮筛选中的100个克隆,进行单克隆噬菌体ELISA,抗原(FSHR)包被质量浓度为5 μg/ml,以BSA作为阴性对照,以Anti-M13KO7-HRP作为二抗(稀释比例为1∶5 000),孵育并检测450 nm处的吸光度值。结果显示,FSHR组的100个克隆和阴性组相比,差异无统计学意义(P<0.05)。
Knappik等于2000年成功构建了合成抗体库,近年来学者又成功构建全合成ScFv抗体库等一系列合成文库,并通过这些文库获得了亲和力较高的抗体。合成文库已成为现代生物学研究的重要手段[16,17]。抗体库的多样性是衡量一个文库质量的重要指标。而抗体的多样性主要由抗体结构的CDR区决定的,其中CDR3区起主要作用。因此,采用CDR3随机突变合成,可很大程度上提高抗体库的多样性。本研究是以纳米抗体VHH06为基础,对其CDR3区进行突变(NNY)16,成功构建VHH06-ΔCDR3随机突变文库。结果表明,该文库的多样性良好。本研究中采用的天然文库基因来自骆驼体内的B淋巴细胞中的RNA,通过2轮PCR获得VHH基因。在这个过程中,PCR引物设计对抗体库多样性方面起到至关重要的作用,引物设计不合适就有可能导致某些抗体家族基因丢失或者抗体库中的基因与原始基因间存在差别,进而影响最终抗体库的多样性。自然状态下,由于骆驼机体自身的免疫耐受和生存环境的影响,其抗体序列中一些位点上的氨基酸分布不是随机的,而是有一定的偏好性,如本研究中高通量测序结果显示在第105、106位出现Ala的概率很高,107位的Gly,115、117位的Tyr及116位的Asn的出现比率也相对较高(表1),这种偏好性会对文库的多样性产生一定影响。
本研究中使用的抗体库为合成抗体库,菌落PCR和测序结果显示,该文库的多样性和库容量都符合要求。在对随机突变文库的筛选过程中和之后的验证结果(多克隆Phage ELISA结果)证明,针对特定抗原FSHR的克隆有明显富集,但最终没有获得亲和力达到预期强度的纳米抗体。通过分析,认为其原因可能有2个方面。首先,随机突变文库的基因序列是人工合成的,虽然该文库的多样性很好,但其中包括了部分没有生物学活性的克隆,即无效克隆[17],因此该突变文库的多样性还需要进一步进行探究。为减少无效克隆出现,在抗体库的设计过程中尤其是关键位点的氨基酸设计一定要合理,可以采用生物信息学分析,预先了解该物种抗体序列中氨基酸的分布情况,模拟设计出更加接近天然抗体的序列。在该序列的基础上再进行突变,这样的策略不仅可减少无效克隆的出现,也是提高抗体亲和力的途径之一。其次,抗体库的库容越大,最终获得特异性抗体的亲和力越高。有文献报道,当抗体的库容达到107~108其筛选获得的抗体只能达到μmol/L的亲和性[18]。VHH06-ΔCDR3突变文库的库容量为108,但真实库容量小于理论值。如果想获得更高亲和力的纳米抗体,可能需要构建库容量更大的噬菌体抗体库。此外,影响库容的因素不是单方面的,在抗体库的构建过程中,载体和基因的连接效率和重组子的电转效率等都是重要影响因素。随着人类对抗体结构的深入研究并分析抗体结构的特点,合成抗体库将成为获得库容量大和多样性好的优质文库的有效途径。
所有作者均声明不存在利益冲突



























