
从蛋白质吸附量、吸附层厚度、蛋白质吸附后分子构象变化、蛋白质吸附后分子形貌及蛋白质分子吸附过程模拟5方面,综述了近年来生物材料表面蛋白质吸附领域所涉及的表征方法,包括生化分析法、表面等离子共振(SPR)、耗散型石英晶体微天平(QCM-D)、椭圆偏振光法(ELM)、光干涉反射法(RIFS)、傅里叶变换衰减全反射红外光谱法(ATR-FTIR)、圆二色谱法(CD)、原子力显微镜(AFM)和计算机分子模拟技术。重点介绍了上述表征方法的基本原理、优缺点及相关的研究工作,为蛋白质吸附、生物材料设计等研究中蛋白质实验表征方法的选择提供了全面、可靠的依据,为蛋白质相关领域的研究提供了新思路。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
当一块固体材料(导管、支架、人造髋关节或组织工程材料)植入人体后,植入材料首先与蛋白质溶液(血液、组织液)接触,溶液中的蛋白质会迅速吸附于材料界面,在几秒至几分钟内达到饱和,形成一层蛋白质吸附层,故宿主细胞并不直接与材料表面接触,而是与材料表面吸附的蛋白质发生吸附[1]。细胞膜表面特异性受体与吸附层蛋白质的生物活性位点结合[2],经一系列复杂的细胞信号转导,外部信号通过细胞膜进入细胞内部,刺激细胞产生应答,最终决定细胞在材料表面的细胞反应[3]。由此可见,吸附于材料表面的蛋白质的种类、数量、层厚、构象[4]、形貌[5]等因素将决定材料表面的细胞反应,而生物材料表面蛋白质吸附行为和机制的研究对生物材料的设计具有重要意义,因此蛋白质吸附一直是生物材料领域的研究重点。
多年来,生物材料表面蛋白质吸附的研究虽得出了大量结论,但由于该研究涉及蛋白质、吸附环境和吸附材料3方面因素[6],蛋白质种类繁多、结构多变,吸附环境复杂以及吸附材料多样导致许多研究的结论彼此不相关、不一致,甚至相反[7]。目前,对生物材料表面蛋白质吸附行为和机制尚未清楚,因此通过生物材料表面蛋白质吸附等研究,指导生物材料表面设计和修饰[8],进而调控材料表面的细胞行为仍是未来生物材料领域研究者的努力方向。
值得庆幸的是随着科学技术的发展,出现了一系列先进的检测技术。通过这些检测技术可获取蛋白质吸附量、吸附层厚度、蛋白质吸附后分子构象变化、蛋白质吸附后分子形貌和蛋白质吸附过程模拟等信息,不同角度和层面的检测信息有助于全面了解蛋白质吸附的过程和机制。本文综述了近年来生物材料表面蛋白质吸附领域所涉及的多种表征方法,包括生化分析法、表面等离子共振(surface plasmon resonance,SPR)、耗散型石英晶体微天平(dissipative quartz crystal microbalance,QCM-D)、椭圆偏振光法(ellipsometry,ELM)[9]、光干涉反射法(reflectometry interference spectroscopy,RIFS)[10]、傅里叶变换衰减全反射红外光谱法(attenuated total reflection Fourier transformed infrared spectroscopy,ATR-FTIR)[11]、圆二色谱法(circular dichroism,CD)[12]、原子力显微镜(atomic force microscopy,AFM)[13]和计算机分子模拟技术[14],重点介绍了上述表征方法的基本原理、优缺点及相关的研究工作,为蛋白质吸附、生物材料设计等研究中蛋白质实验表征方法的选择提供了全面和可靠的依据,为蛋白质相关领域的研究提供了新思路。
以二喹啉甲酸(bicinchoninic acid, BCA)法为例,染色法是利用蛋白质与染料结合形成复合物,定量测定微量蛋白质浓度的一种方法。在碱性条件下,二价铜离子被蛋白质还原成一价铜离子,一价铜离子和BCA分子螯合形成水溶性复合物,该复合物在562 nm处显示强烈的吸光性,吸光度和蛋白质浓度在较大范围内具有良好的线性关系,可根据吸光度值推算出蛋白质浓度,最小检测蛋白量可达0.5 μg。该法具有简单、快速、灵敏等特点,已成为最常用的蛋白质浓度定量检测手段之一。笔者曾使用BCA法系统研究了羟基磷灰石(hydroxyapatite, HA)颗粒表面的微纳结构[15]、Zeta电位[16]等因素对蛋白质吸附行为的影响,发现HA表面的微纳结构对蛋白质具有选择性吸附作用,带状HA(图1A)对酸性蛋白质如牛血清白蛋白(bovine serum albumin, BSA)、纤维粘连蛋白(fibronectin, FN)具有选择性吸附作用(图1B)[15]。该法定量精确,是研究生物矿化(骨骼、牙齿)中磷酸钙盐与蛋白质吸附行为的重要表征技术。


LYS—溶菌酶;FN—纤维粘连蛋白;BSA—牛血清白蛋白
同位素法是通过检测样品溶液的放射量,再根据已知浓度蛋白质溶液的放射量,计算出样品溶液蛋白质浓度的方法(图2)。同位素法可对被标记的蛋白质进行精确的定量检测,因而被广泛用于生物材料表面蛋白质吸附的研究。Yu等[17]利用125I同位素法研究了人血清白蛋白(human serum albumin, HSA)在N-异丙基丙烯酰胺接枝表面的吸附行为,发现与未改性硅表面相比,N-异丙基丙烯酰胺接枝表面的HSA吸附量下降97%,说明N-异丙基丙烯酰胺接枝表面具有较好的阻抗HSA非特异性吸附的性能。


同位素法精确度极高,可用于精确检测不同材料表面的蛋白质吸附行为[18,19],还可用于研究血液接触材料表面的弗罗曼效应(Vroman effect)[20,21]。需要指出的是该法不能提供蛋白构象方面的信息,同时对标记废物的处理程序复杂、费用较大。
SPR是一种光学现象,可用于精确定量生物分子的相对分子质量。如图3所示,蛋白质分子结合在金表面,导致折射率发生改变,折射率改变的程度与结合蛋白质分子的相对分子质量成正比;而折射率的改变又导致入射光临界角(即SPR共振角)发生变化,通过测定SPR共振角可得到吸附于金表面的蛋白质的相对分子质量[22]。利用SPR检测蛋白质在材料表面的吸附量和吸附强度,有利于表征材料的蛋白质吸附性能。Chang等[23]利用聚环氧乙烷-聚氧化丙烯-聚环氧乙烷三嵌段聚合物对甲基为末端的自组装单分子层材料表面进行涂层处理,通过改变聚环氧乙烷/聚氧化丙烯的质量比和共聚物的相对分子质量获得了一系列不同性能的表面;SPR表征结果显示,当共聚物相对分子质量为15 000、聚环氧乙烷/聚氧化丙烯的质量比为4∶1时,对应的材料表面具有最强的抗蛋白质吸附能力。Liu等[24]在金表面制备聚N-乙烯吡咯烷酮,利用SPR技术表征纤维蛋白原在其表面的吸附量,发现聚N-乙烯吡咯烷酮具有较强的抗蛋白质吸附性能。同样,Luk等[25]利用SPR技术表征甘露糖醇单分子层的蛋白质吸附及细胞黏附性能,发现甘露糖醇修饰的表面具有较强的抗蛋白质吸附和抗细胞黏附性能。
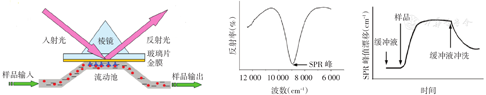

SPR—表面等离子共振
QCM-D可将蛋白质吸附质量和黏弹性信号转换为电路中的频率信号,是专门检测蛋白质等生物黏弹性物质的微天平(图4)。随着蛋白质分子在待测材料表面发生吸附,QCM-D将检测到质量的增加,精确度可达纳克级别,从而可实时检测蛋白质分子在待测材料表面的吸附质量。Lord等[26]利用QCM-D技术研究了聚甲基丙烯酸甲酯和几种以聚甲基丙烯酸-2-羟乙酯水凝胶为基底的材料对BSA的吸附行为,结果发现与以聚甲基丙烯酸-2-羟乙酯水凝胶为基底的亲水性材料相比,疏水性的聚甲基丙烯酸甲酯材料对BSA具有更强的吸附性能。Menz等[27]利用QCM-D技术研究了聚乙二醇改性表面和羟基改性表面的蛋白质吸附行为,发现聚乙二醇改性表面具有较强的抗蛋白质吸附性能。Li等[28]通过实时检测吸附质量的变化,得到了人血纤维蛋白原(fibrinogen,FIB)在金片表面随pH值变化的吸附曲线,发现FIB在酸性条件下的吸附速率明显快于碱性条件。
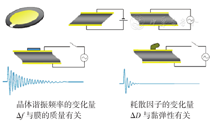

ELM是一种利用椭圆偏振光进行光学测量的常规技术,该法可用于检测并计算材料表面的蛋白质吸附量[29]。近年来,该法被广泛用于检测聚合物[30]、硅及HA[31]等生物材料表面蛋白质吸附层的厚度。另外,在很多蛋白质吸附研究中,ELM被用于表征单分子层、聚合物涂层的厚度。黄金[32]利用ELM测定了双亲性V型聚合物刷样品的厚度,精确度达±0.4 nm。ELM的缺点是对表面要求较高,需表面平坦或具有反射底层,且该法不能区分蛋白质的种类[33]。
RIFS是近年来发展起来的一种新型生物信息获取技术。该技术集生物医学、生物电子学和工程学等技术为一体,是测量蛋白质吸附层厚度的有力手段。吕晓迎等[34]利用RIFS技术研究了BSA、人血FIB和免疫球蛋白(IgG)在HA和聚氨酯两种材料表面的吸附行为,动态检测了蛋白质在上述两种材料表面的吸附层厚度。该法具有免标记、精确、灵敏、支持实时动态分析等优点,是研究蛋白质吸附的一种新检测技术。
ATR-FTIR是在傅里叶变换红外光谱(FTIR)技术基础上,结合衰减全反射(ATR)技术发展起来的一种表层物质成分和构象分析的新技术。一般认为,蛋白质的酰胺I带(1 700~1 600 cm-1)反映了α-螺旋、β-折叠、无规则卷曲、转角等二级结构的信息[35]。通过在锗晶体表面制备不同材料的薄膜,对比蛋白质在材料表面吸附前后酰胺I带峰的相对强度和位移,可推测蛋白质分子的构象变化,用以评价材料对蛋白质生物活性的影响,为生物材料的功能设计提供实验依据。叶青等[36]利用ATR-FTIR技术研究了BSA在HA表面的吸附,发现BSA在HA表面吸附后,BSA的二级结构由β-折叠转变为α-螺旋和小部分β-转角,研究认为这是导致BSA在HA表面具有较大吸附量的原因,这一特性有助于细胞对HA界面的识别,进而加速生物矿化过程。另外ATR-FTIR技术也被用于研究二氧化钛薄膜表面的蛋白质吸附行为,Bouhekka和Bürgi[37]利用该技术研究了BSA在二氧化钛薄膜表面吸附前后二级结构的变化,发现BSA吸附后分子二级结构发生了改变;同时发现BSA被可见光照射后,α-螺旋含量逐渐减少,无规则卷曲含量逐渐增大。
ATR-FTIR技术可针对性地分析与材料表面发生吸附的化学物质的成分和构象,并能排除未与界面发生吸附的物质的干扰[38]。该技术避免了常用透过光谱法对样品结构的粉碎性破坏,极大简化了一些特殊样品的测试,具有原位、实时、无放射性和无损等特点,适用于非均匀、凹凸表面,可获得微区表面化合物官能团空间分布的红外光谱图像,因此受到广泛关注和应用。
CD是一种特殊的吸收谱,其通过测量蛋白质等生物大分子的圆二色光谱,得到生物大分子的二级结构信息,因其操作简单、快捷等特点,广泛应用于蛋白质折叠、构象、酶动力学及蛋白质吸附研究中。
蛋白质吸附后发生的构象变化可体现在α-螺旋结构的改变,CD可检测蛋白质中α-螺旋结构的含量,故被广泛用于蛋白质吸附后的构象变化检测[12]。Tanaka等[39]在研究聚丙烯酸-2-甲氧基乙酯材料表面血液相容性时发现,该材料能有效抑制血小板的黏附,具有较好的血液相容性。进一步利用CD检测血浆蛋白在材料表面吸附后的构象变化,结果发现吸附于聚丙烯酸-2-甲氧基乙酯材料表面的血浆蛋白构象(α-螺旋)仅发生很小变化,而吸附于对照组材料(聚甲基丙烯酸-2-羟乙酯)表面的血浆蛋白构象发生了较大程度改变,由此认为聚丙烯酸-2-甲氧基乙酯材料具有良好血液相容性的主要原因是吸附于其表面的蛋白质几乎不发生构象变化,能较好地保持蛋白生物活性。Norde和Favier[40]利用CD研究了溶菌酶在二氧化硅颗粒表面吸附、脱附过程中α-螺旋的含量变化,结果发现在饱和吸附状态下溶菌酶分子能较好地维持其天然构象,而在吸附-脱附过程中溶菌酶发生了不可逆的构象变化。
AFM可在溶液环境下对自然活性状态的蛋白质进行高分辨率免疫标记探测,是ATR-FTIR、ELM、同位素法、SPR等传统研究技术的有力补充,已成为研究蛋白质吸附的重要手段。
早在1990年,Lin等[41]即开始用AFM研究蛋白质吸附过程,他们实时观测了鼠荧光素单克隆抗体在云母界面的动态吸附过程,结果发现该抗体能在云母表面发生吸附,孤立吸附在云母表面的抗体分子易发生脱附,而吸附在云母表面的相邻抗体分子团聚后能稳定在云母表面而不发生脱附,表明云母表面相邻蛋白质分子的侧面吸附有助于蛋白质分子吸附的稳定。AFM技术可直观了解蛋白质分子吸附后的形貌变化。Agnihotri和Siedlecki[5]用AFM分别研究了FIB在疏水性石墨表面和亲水性云母表面的吸附行为。如图5所示,AFM下清晰可见随着吸附的进行,FIB在疏水性石墨表面逐渐铺展,吸附作用较强;而在亲水性云母表面,FIB的形貌变化较小,吸附作用较弱。另外,利用AFM探测FIB中结构域D和E之间的高度差,结果显示FIB在疏水性石墨表面吸附后结构域D和E之间高度差减小,这为解释蛋白质在疏水性表面吸附后发生较大程度构象变化提供了直观证据。


需要指出的是,通过AFM能清楚地看到单个蛋白质分子在界面上吸附后的分子形态以及动态吸附过程,但由于AFM探针会不可避免地对蛋白质分子造成扰动[42],因此通过AFM观察到的形态不能代表蛋白质分子与界面相互作用的真实情况,这种弊端有待未来通过优化AFM的工作模式来避免。
目前,生物材料表面蛋白质吸附的研究大多是通过测定宏观的量,再经推理得到一些结论,而不能从分子、原子尺度揭示蛋白质吸附的具体过程,但许多涉及蛋白质吸附的命题,如生物矿化、硬组织形成机制等,均需从原子、分子水平理解蛋白质吸附的具体过程,进而从结构与功能角度进行材料设计。
计算机模拟技术可整合现有实验数据,在固体表面模拟出蛋白质吸附后的结构[43],从而提供原子、分子尺度的有关信息。目前,计算机模拟技术已被用来研究蛋白质在固体表面的吸附机制[44]。Dong等[45]利用分子动态平衡模拟、拉伸分子动力学模拟及量子化学计算等方法研究了骨形态发生蛋白-2(bone morphogenetic protein-2, BMP-2)在HA(001)晶面上的吸附行为,发现HA(001)晶面上的吸附基团主要为—OH、—NH2和—COOH,参与蛋白质吸附的基团可为其中的1种或多种;静电引力是—COOH与HA相互作用的主要作用力,氢键是—OH、—NH2与HA发生吸附的主要作用力;此外还发现Asp、Glu、Lys和Arg这4种氨基酸残基在各种蛋白质中普遍被吸附到HA表面。Liao等[46]模拟了FNⅢ型域模块10(FN-Ⅲ10)在HA(001)晶面的吸附过程,结果发现在吸附初始阶段,静电引力是主要的吸附作用力,Arg93残基首先吸附于Ca(Ⅰ)原子所在空位(图6A);吸附时间为6.11 ns时,吸附能为-90 kJ/mol,此时Arg93残基与HA表面的距离为0.2 nm(图6B);随着吸附的进行,在吸附时间为20~100 ns时,大量其他氨基酸残基逐渐在HA表面发生吸附,氨基酸残基与HA表面带电离子发生电性错配,导致静电斥力增大,静电引力作用减弱,范德华力逐渐增大并成为主要的吸附作用力,使FN-Ⅲ10中无规则卷曲部分的结构发生变化,导致FN-Ⅲ10的构象发生变化(图6C、图6D)。
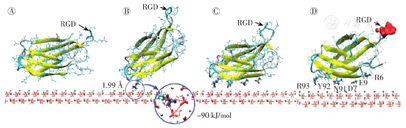

FN-Ⅲ10—纤维粘连蛋白III型域模块10;RGD—精氨酸-甘氨酸-天冬氨酸;1 Å=10-10 m
在蛋白质调控HA晶体成核、生长研究方面,Yang等[47]采用一段由10个氨基酸组成的肽链作为骨涎蛋白成核区域肽链的模型(图7),利用分子动态模拟技术模拟了模型肽链对溶液中Ca2+、PO43-空间分布的影响,以及不同构象的模型肽链对溶液中HA成核的影响。研究结果显示,各种构象的模型肽链均能起到成核作用,但不能形成具有稳定形状的HA微晶,研究认为骨涎蛋白成核模型能成核形成无定形态的磷酸钙团簇,后者最终转化为HA。
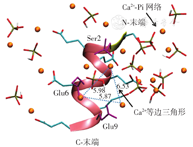

此外,计算机模拟技术还可用于研究蛋白质吸附后的构象变化。Zhou等[48]采用分子动力学模拟配合驾驭式分子动力学模拟的策略研究了骨形态发生蛋白-7(BMP-7)在HA表面吸附后的构象变化,他们设计了HA的2种自然晶面和4种极限晶面,发现BMP-7在2种自然晶面吸附后的构象变化不大,而在4种表面离子密度较大的极限晶面上,BMP-7的构象发生了不同程度的变化。
本文从蛋白质吸附量、吸附层厚度、蛋白质吸附后分子构象变化、蛋白质吸附后分子形貌及蛋白质分子吸附过程模拟5方面,对近年来生物材料表面蛋白质吸附领域所涉及的表征方法进行了归纳和总结,主要介绍了各种表征方法的基本原理、优缺点及相关的研究工作。本文为蛋白质吸附、生物材料设计等研究中蛋白质实验表征方法的选择提供了全面和可靠的依据,为蛋白质相关领域的研究提供了新思路。
所有作者均声明不存在利益冲突



























