
探究驱动蛋白家族成员C1(KIFC1)在三阴性乳腺癌(TNBC)中的功能和作用。
通过生物信息学的方法分析KIFC1在TNBC组织及癌旁正常组织中的mRNA水平,并分析其表达与患者的生存率间的关系。回顾性分析96例TNBC患者的临床病理特征资料。用免疫组织化学的方法检测肿瘤组织和癌旁正常组织中KIFC1的表达,分析TNBC患者的KIFC1表达及与临床病理特征的关系。
生物信息学分析结果显示,TNBC组织中KIFC1高表达且与患者的无病生存期相关(P<0.05)。在TNBC组织中,KIF23阳性高表达率为58.3%,而在癌旁组织中主要是低表达或无表达。KIF23的高表达与TNBC患者的肿瘤分级相关(P<0.05)。体外细胞实验结果表明,敲低KIFC1可显著降低TNBC细胞的集落形成能力和增殖能力。小鼠体内实验结果表明,敲低KIFC1可显著降低肿瘤体积。
KIFC1可作为TNBC的预后因子,低表达KIFC1可在体内外抑制TNBC细胞的增殖。KIFC1有望作为TNBC的预后标志物和治疗靶点。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
乳腺癌是女性最常见的恶性肿瘤,是女性患者癌症相关死亡的第二大原因[1,2,3]。在乳腺肿瘤的亚型中,有15%~20%为伴有乳腺癌易感基因(breast cancer susceptibility genes,BRCA)种系突变的三阴性乳腺癌(triple-negative breast cancer,TNBC)[4,5,6]。TNBC作为很少表达雌激素受体、孕激素受体和人表皮生长因子受体2(human epidermal growth factor receptor 2,HER2)的肿瘤,其发病率在世界范围内快速增长[2,3]。然而,因为缺乏内分泌治疗和靶向治疗方法,TNBC在所有类型的乳腺癌中预后最差[7,8,9]。因此,确定治疗TNBC的分子靶点具有重要意义。
驱动蛋白家族成员C1(kinesin family member C1,KIFC1)是负端定向运动蛋白,与有丝分裂过程中的纺锤体形成、中心体聚集和微管运输有关[10,11,12,13]。KIFC1属于驱动蛋白14家族的C型驱动蛋白。此前的研究结果表明,KIFC1参与卵母细胞发育[14]、细胞器转运[10]、双链DNA转运[15]、精子发生[16]等生物学功能。研究结果还表明,KIFC1参与多种肿瘤,包括卵巢癌[17]、前列腺癌[18]、非小细胞肺癌[19]、食道鳞状细胞癌[20]。此外,KIFC1可作为非裔美国女性TNBC的预后标志物[21]。
在本研究中,研究TNBC患者中KIFC1蛋白的表达水平,论证KIFC1蛋白的过表达与TNBC不良临床结局的关系并进一步探讨其机制。
选择2010年11月至2020年11月天津医科大学第二医院收治的TNBC乳腺癌患者96例,所有患者均为女性,年龄46.2岁±5.93岁。全部病例均经过临床病理证实,并具有完整的病例资料。收集患者的肿瘤组织标本和相应的癌旁正常组织样本。本研究中涉及的所有患者的资料及标本采集工作均得到了患者的知情同意。
BALB/c裸鼠(18~22 g)(北京维达河实验动物科技有限公司),MCF-7和MDA-MB-231人源TNBC细胞株、青霉素、链霉素(美国模式培养物研究所),Eagle最低基础培养基(Eagle's minimal essential medium,EMEM)、Dulbecco改良Eagle培养基(Dulbecco's modified Eagle medium,DMEM)、胎牛血清(fetal bovine serum,FBS)(美国GIBCO公司),KIFC1重组抗体(ab172620)、抗β-肌动蛋白抗体(ab8227)、Ki67重组抗体(ab16667)、增殖细胞核抗原(proliferating cell nuclear antigen,PCNA)抗体(ab18197)(英国Abcam公司),RNA iMAX试剂(美国Thermo Fisher Scientific公司),SuperMix逆转录聚合酶链式反应(reverse transcription polymerase chain reaction,RT-PCR)试剂盒(北京全式金生物技术股份有限公司),全蛋白检测试剂盒(江苏凯基生物技术股份有限公司),聚偏氟乙烯[poly(vinylidene fluoride),PVDF]膜(美国Millipore公司),细胞计数试剂盒-8(cell counting kit8,CCK-8)(碧云天生物技术有限公司)。
CO2恒温细胞培养箱(新加坡ESCO公司)。
利用癌症基因组图集(the cancer genome atlas,TCGA)数据库[22]进行生物信息学分析,分析KIFC1的mRNA在TNBC组织和正常组织中的表达情况,并探讨其预后评估价值。用Kaplan-Meier曲线分析KIFC1高表达者和低表达者之间的生存差异。TCGA的网址为http://fireb rowse.org/。
首先对TNBC组织进行脱蜡处理;然后用梯度乙醇溶液(100%,100%,95%,85%,75%)水合;水合完毕后,装入压力为103 kPa的高压锅中,使用柠檬酸缓冲液孵育30 min提取抗原;之后用1∶100稀释的KIFC1重组抗体进行免疫染色;最后对抗体进行酶促检测。免疫组化结果由两位独立的病理专家进行审查和评分。
将MCF-7细胞置于DMEM培养基中,MDA-MB-231细胞置于DMEM培养基中,然后分别加入体积分数为10%的FBS、100 U/L的青霉素和0.1 mg/ml的链霉素。处理后的细胞均置于5%的CO2细胞培养箱中,37 ℃保存。
为了探讨KIFC1对TNBC细胞的影响,本研究中利用shRNA载体建立敲低KIFC1的MCF-7和MDA-MB-231细胞株。购买商品化的腺病毒(adenovirusassociatedvirus,AAV)载体进行shRNA克隆,目标序列如下。sh1:5′-AAATTACCACATCCCACCCAAGA-3′;sh2: 5′-AAACGTTGGACCAAGAGAACCAG-3′;sh3: 5′-AAGTGGACAGGATGAAGTGTTTG-3′;sh4: 5′-AACAGCAAACTGACCTACCTGCT-3′。采用RNA iMAX试剂转染的MCF-7和MDA-MB-231细胞为shRNA组;以错义序列转染的MCF-7和MDA-MB-231细胞为对照组。
使用RT-PCR试剂盒进行cDNA分析。首先,将TRIZOL试剂加入培养的细胞中,提取总RNA;然后,将分离出的RNA逆转录成cDNA;最后,使用RT-PCR方法对cDNA进行分析。
以β-肌动蛋白(β-actin)为内参,引物如下。KIFC1的正向引物为5'-TGAGCAACAAGGAGTCCCAC-3',反向引物为5'-TCACTTCCTGTTGGCCTGAG-3';β-actin的正向引物为5'-TGACGTGGACATCCGCAAAG-3',反向引物为5'-CTGGAAGGTGGACAGCGAGG-3'。以β-actin作为标准化对照,采用比较CT(cycle-threshold)值法对样品扩增进行计算,再采用2-ΔΔCT法计算KIFC1的相对表达量。
使用蛋白检测试剂盒测定蛋白浓度。蛋白样品通过十二烷基硫酸钠聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamide gel electrophoresis,SDS-PAGE)法进行评估。用含10%的聚丙烯酰胺的SDS-PAGE凝胶进行电泳,使蛋白质样本分离;将凝胶上的蛋白转移到PDVF膜上;用含5%脱脂乳的TBST缓冲液进行阻断,用抗体在4 ℃下孵育过夜;将样品与二抗在室温下孵育1 h;用化学发光(enhanced chemiluminescence,ECL)法对条带进行可视化处理。使用β-actin作为内参。用PCNA和Ki67来反映TNBC细胞的增殖活性。
将每孔500个细胞接种于6孔板中培养2周,用甲醇固定菌落,用0.1%的结晶紫染色,方便获取集落大小和数量。
使用CCK-8试剂盒测定细胞存活率。在96孔板上每孔接种2 000个细胞,并于第3天记录细胞存活率。
裸鼠的养育和操作经过了天津医科大学第二医院动物伦理委员会的批准。将4周龄的雌性无胸腺BALB/c小鼠分为两组,分别在小鼠右侧皮下注射上述细胞培养中成功建立的两种稳定MDA-MB-231细胞系(KIFC1抑制组和对照组)。注射2周后,每7天测量肿瘤尺寸;注射7周后处死小鼠,分离肿瘤组织并拍照。
采用SPSS22.0软件进行统计学分析。复合正态分布的样本以均数±标准差(Mean±SD)表示。两组间比较采用独立样本t检验,临床病理特征与KIFC1表达的相关性采用χ2分析。以P<0.05为差异有统计学意义。
从TCGA数据库中获取的相关数据集中包括癌旁正常组织291例,TNBC组织1 085例。结果表明,TNBC组织中KIFC1 mRNA水平明显低于正常组织(P<0.001),如图1所示。此外,KIFC1 mRNA水平与TNBC患者的无病生存率相关(P=0.043),如图2所示。上述结果提示,KIFC1在人TNBC组织中表达增强,并与TNBC患者的不良预后相关。
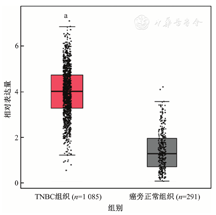

TNBC—三阴性乳腺癌;与癌旁正常组织比较,aP<0.001
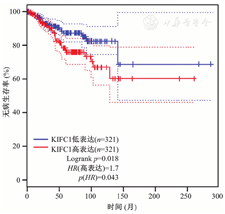

HR—风险比;Logrank—时序检验
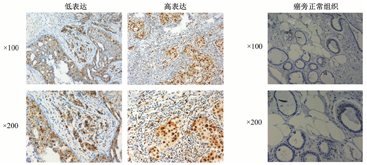
免疫组化结果表明,KIFC1在TNBC组织中的高表达率为58.3%(56例/96例),低表达率为41.7%(40例/96例),而KIFC1在癌旁正常组织中主要是低表达或无表达(图3)。进一步分析KIFC1的表达与TNBC的临床病理特征的关系。结果显示,KIFC1在TNBC组织中的表达与肿瘤分级相关(P=0.016),且高表达KIFC1者有发生远处转移的趋势。但是KIFC1高表达和低表达者在年龄、肿瘤大小、淋巴结转移等方面的差异无统计学意义(均P>0.05),见表1。
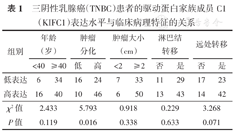
三阴性乳腺癌(TNBC)患者的驱动蛋白家族成员C1(KIFC1)表达水平与临床病理特征的关系
三阴性乳腺癌(TNBC)患者的驱动蛋白家族成员C1(KIFC1)表达水平与临床病理特征的关系
| 组别 | 年龄(岁) | 肿瘤分化 | 肿瘤大小(cm) | 淋巴结转移 | 远处转移 | |||||
|---|---|---|---|---|---|---|---|---|---|---|
| <40 | ≥40 | 低 | 高 | <2 | ≥2 | 否 | 是 | 否 | 是 | |
| 低表达 | 6 | 34 | 16 | 24 | 7 | 33 | 11 | 29 | 17 | 23 |
| 高表达 | 16 | 40 | 10 | 46 | 6 | 50 | 13 | 43 | 14 | 42 |
| χ2值 | 2.433 | 5.793 | 0.918 | 0.229 | 3.268 | |||||
| P值 | 0.119 | 0.016 | 0.338 | 0.633 | 0.071 | |||||

上述结果表明,KIFC1在TNBC组织中的表达水平很高,并且与临床病理特征相关。
用RT-PCR和Western blot方法验证敲低KIFC1的MCF-7和MDA-MB-231细胞,结果如图4所示。结果表明,shRNA1(sh1)的敲低效果最明显。
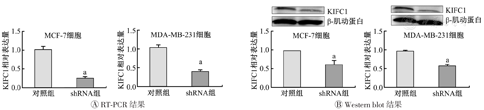

RT-PCR—逆转录定量聚合酶链反应;KIFC1—驱动蛋白家族成员C1;对照组为未敲低KIFC1的MCF-7和MDA-MB-231;细胞;shRNA组为敲低KIFC1的MCF-7和MDA-MB-231;与对照组比较,aP<0.05
平板菌落实验结果表明,当KIFC1被敲低时,MCF-7和MDA-MB-231细胞的克隆形成能力降低;CCK-8细胞计数实验结果显示,当KIFC1基因被敲低时,MCF-7和MDA-MB-231细胞的活力和增殖能力均下降;Western blot结果表明,敲低KIFC1基因的MCF-7和MDA-MB-231细胞的Ki67和PCNA表达量均显著降低,说明敲低KIFC1后,细胞的增殖受到抑制。(图5)
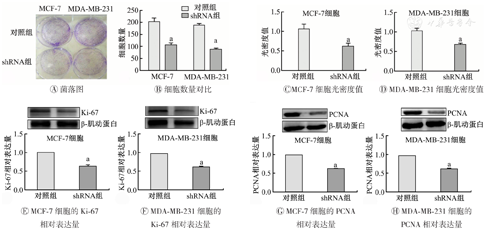

KIFC1—驱动蛋白家族成员C1;PCNA—增殖细胞核抗原;对照组为未敲低KIFC1的MCF-7和MDA-MB-231;shRNA组为敲低KIFC1的MCF-7和MDA-MB-231细胞;与对照组比较,aP<0.05
上述结果表明KIFC1在体外能显著促进TNBC细胞的增殖。
结果显示,敲低KIFC1的小鼠的肿瘤体积明显小于对照组(图6)。Western blot结果显示,敲低KIFC1的小鼠的KIFC1表达水平明显低于对照组(图7A);而免疫组化结果与Western blot结果一致(图7B)。上述结果表明,KIFC1在小鼠体内能促进TNBC细胞的增殖。
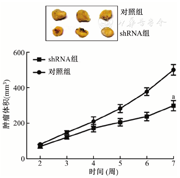

对照组为未敲低KIFC1的小鼠;shRNA组为敲低KIFC1的小鼠;与对照组比较,aP<0.05
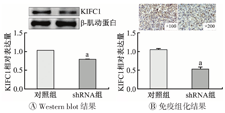

与对照组比较,aP<0.05
TNBC是所有乳腺癌中预后最差的恶性肿瘤亚型[9,23]。然而,TNBC患者缺乏内分泌治疗和靶向治疗。因此,需要新的方法和策略来治疗TNBC。在本研究中发现KIFC1在大多数TNBC患者中表达,其表达水平与TNBC患者的无病生存期相关。与本研究的结果类似,已有报道KIFC1蛋白表达升高与卵巢癌[17]、前列腺癌[18]、非小细胞肺癌等预后较差相关[19,20,24,25]。因此,KIFC1在未来有可能将成为TNBC治疗的新靶点。
KIFC1是负端定向运动蛋白,与有丝分裂过程中的纺锤体形成、中心体聚集和微管运输有关[10,13]。KIFC1通过输入RanGTP和α/β酶,并与未定义的核孔蛋白配合物相互作用,将相关蛋白运到细胞核中[16,26,27]。在其他肿瘤中,KIFC1有促进肿瘤增殖的作用[19,28,29]。本文中,探讨了KIFC1对TNBC的增殖作用,通过建立两个敲低KIFC1的TNBC细胞系,体外研究了KIFC1对TNBC细胞增殖的促进作用。此外,基于KIFC1敲低小鼠模型,用体内实验进一步验证了KIFC1对TNBC细胞的增殖作用。
Xiao等[25]报道KIFC1在膀胱癌中也可促进上皮间质转化(epithelial mesenchymal transition,EMT)的发生。Maurizio等[30]发现KIFC1在高迁移率族蛋白A1(high mobility group protein A1,HMGA1)沉默的乳腺癌细胞中表达降低。HMGA1可诱导信号传导及转录激活蛋白(signal transducer and activator of transcription,STAT3)的表达,STAT3也上调HMGA1,导致两种基因表达增强[31]。Han等[32]关注KIFC1的上游,其报道称KIFC1受miR-532-3p调控,然后通过gankyrin/AKT信号通路促进EMT和肝细胞癌转移。
本研究存在以下几点缺陷:一是预后结果主要来自公共数据库;二是组织标本数量较少,且没有进行多中心研究;三是针对机制的研究不足。因此,应在未来对KIFC1进行更深入的研究,以便进一步揭示KIFC1对TNBC的作用机制。
综上所述,KIFC1是TNBC的预后因素,KIFC1在体内和体外均能促进TNBC细胞的增殖。本研究结果为KIFC1作为TNBC潜在的预后标志物和治疗靶点的应用提供了理论依据。
所有作者均声明不存在利益冲突































