
直肠癌是我国常见的消化道恶性肿瘤之一,准确、合理地评估直肠癌术前分期可以显著提升治疗效果,改善患者预后。磁共振成像是直肠癌局部分期的首选技术,在直肠原发肿瘤(T)和肠周淋巴结(N)的诊断上具有显著优势。围绕多模态磁共振图像,综述了传统影像组学和深度学习方法在直肠癌术前TN分期预测方面的研究思路和进展,以期为实现全自动的直肠癌TN分期算法提供新的思路。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
直肠癌是指位于直肠和乙状结肠交界处至肛门齿状线之间的癌,是消化道最常见的恶性肿瘤之一,约占结直肠癌总量的1/3。据统计,2020年全球结直肠癌发病率和死亡率在所有癌症中分别位居第3、2位[1]。其中,早期局部结直肠癌患者的5年生存率高达90%,而晚期患者生存率仅为14%,但多数患者在确诊时已属于中晚期[2]。目前,直肠癌的诊治多采用美国癌症协会和国际抗癌联盟提出的结直肠癌肿瘤结节转移(tumor, node, metastasis,TNM)分期系统,以综合评估原发肿瘤、区域淋巴结受累和远处转移情况。根据患者的分期结果,早期肿瘤往往直接采用外科手术切除,而晚期则需要选择性地采用如术前新辅助治疗、区域淋巴结清扫和术后辅助放化疗等手段。因此,准确、合理的术前分期对最佳治疗方案的制定尤其重要,有助于降低术后复发率,改善患者预后,提高患者生存质量[3]。
近年来,随着人工智能的迅速崛起,基于磁共振图像构建直肠癌计算机辅助诊断模型的研究与日俱增,在提高原始肿瘤分期和新辅助治疗后再分期的准确性、解决放疗靶区肿瘤的分割定位和实现放化疗疗效预测等方面取得了诸多进展[4,5]。本文围绕多模态磁共振图像,论述传统影像组学和深度学习方法在直肠癌术前TN分期预测方面的研究思路和进展。现有的直肠癌TN分期算法大多基于肿瘤分割掩膜构建传统影像组学分类模型,而直接使用深度学习技术实现的较少。同时,考虑到直肠癌T分期与肿瘤浸润深度,N分期与转移淋巴结数量息息相关。因此,本文对基于深度学习方法辅助直肠癌TN分期的相关研究进行了拓展介绍,包括辅助原发肿瘤分割和辅助淋巴结诊断2个方面。最后,讨论了目前研究中的不足,并对直肠癌分期的未来发展方向进行展望,以期为构建高精度、全自动的直肠癌TN分期模型提供参考。
直肠癌诊断常规行直肠指检,配合病理组织学检查、直肠内镜和影像学检查,以综合评估癌症情况,包括肿瘤大小、侵犯深度和是否存在可疑淋巴结转移等。磁共振成像(magnetic resonance imaging,MRI)具有多模态、多方位和多角度的特点,是直肠癌局部分期的首选影像学检查手段,多用于明确肿瘤的位置、形态、TN分期、直肠系膜筋膜状态和有无肠壁外血管侵犯等,在直肠癌治疗前后的评估中发挥着关键作用[6]。其中,T2加权成像(T2 weighted image,T2WI),尤其是非抑脂、小FOV和轴位高分辨的T2WI(层厚<3 mm),可以获得高质量的软组织对比度图像,对于主要解剖结构及其与肿瘤关系的表征至关重要,是诊断和研究中使用的主要序列[3,6,7]。另外,扩散加权成像(diffusion weighted imaging,DWI),尤其是高b值DWI(b≥800 sec/mm2),可以敏感探测活体组织及肿瘤内水分子的布朗运动,显示病变位置的活跃程度,有助于辅助评估患者新辅助治疗的效果[7,8]。再者,基于DWI计算的表观扩散系数(apparent diffusion coefficient,ADC)可以量化病变组织的微观病理变化,在直肠癌评估中具备一定潜力,但仍需大范围的标准化研究确认[8,9]。此外,肝脏增强MRI虽然可以进一步评估直肠癌肝转移,但是CT和正电子发射计算机断层显像(positron emission tomography,PET)-CT更适合于检测远处转移。因此,本文主要讨论常规直肠MRI进行术前TN分期的研究。
传统影像组学技术依靠从病灶中提取海量特征来量化肿瘤表型,有效解决了肿瘤异质性难以定量评估的问题,在肿瘤分级、癌症分期和预后评估等方面应用广泛[10]。这类方法通常包括图像获取与预处理、目标区域勾画、图像特征提取、特征选择、构建分类模型和模型评估这几个步骤,整体流程如图1所示。其中,特征提取是实现病灶高通量定量分析的核心步骤,针对二维感兴趣区域(region of interest,ROI)或三维感兴趣体积(volume of interest,VOI),传统影像组学技术可以在全局(整个肿瘤)或局部区域(肿瘤内、平面内或多平面)的尺度上提取大量特征。这些特征描述了影像的灰度值分布和像素空间排列关系,可以充分表征肿瘤异质性,以便后续算法进一步分析。
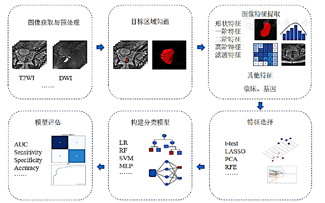

T2WI—T2加权成像;DWI—扩散加权成像;AUC—曲线下面积;Sensitivity—敏感性;Specificity—特异性;Accuracy—准确性;LR—Logistic回归;RF—电磁频率;SVM—支持向量机;MLP—多层感知机;LASSO—最小绝对收缩选择算子;PCA—主成分分析;RFE—递归特征减少法
直肠癌T分期反映了肿瘤原发灶的情况,根据肿瘤浸润程度和邻近组织受累范围的不同,可划分为T1~T4期[3]。根据MRI诊断T分期时,肿瘤周围的炎性反应、渗出性改变、微血管侵犯固有肌层和促结缔组织增生反应等现象会造成肠壁及其周围结构产生类似肿瘤浸润的征象,导致过分期和分期不足的问题,而T2、T3期直肠癌的准确区分更是其中的一大难点[11,12,13]。因此,现有的T分期研究大多采用病理分期作为金标准,使用术前多模态MRI针对性地开展pT1-2和pT3-4期直肠癌的二分类研究。
Sun等[14]基于全体积勾画的肿瘤轴位T2WI,从中提取256个影像组学特征,配合最小绝对收缩选择算子(least absolute shrinkage and selection operator,LASSO)算法构建列线图模型,预测T分期的曲线下面积(area under curve,AUC)达0.852,敏感性为79.0%,特异性为82.0%,初步表明影像组学特征和T分期的相关性。除了直肠影像,血清肿瘤标志物也是直肠癌诊断的参考指标,已有研究表明,癌胚抗原(carcinoembryonic antigen,CEA)和糖类抗原19-9(carbohydrate antigen 19-9,CA19-9)阳性率与结直肠癌患者肿瘤分期呈正相关[15]。为了引入临床特征联合预测分期,Lin等[12]将LASSO筛选出的9个最优预测特征通过线性加权融合为影像组学评分,再结合CEA,构建基于多元Logistic回归(Logistic regression,LR)分析的列线图模型,相较于仅使用影像特征,AUC提升了0.039。斜轴位T2WI是判断浸润深度的主要序列,但是受限于Z轴分辨率,往往会忽略层间的信息。对此,Lu等[13]认为轴位T2WI结合矢状位脂肪抑制T2WI能更为全面地反映肿瘤情况,提高T分期的预测性能,为联合多视角图像建模的有效性提供了依据。
通常原发肿瘤在T2WI上呈中等信号,表现为局灶性肿块或异常壁增厚区,有时难以准确辨别。由于肿瘤组织的细胞体积、密度和核质比均高于正常组织,导致水分子的扩散受限且增多,因此肿瘤在高b值DWI上呈高信号,在ADC图上呈低信号,二者可以为肿瘤诊断提供参考。最初,Liu等[16]基于68例直肠癌患者的ADC图,从中提取ADC值特征、直方图特征和灰度共生矩阵特征,探索基于ADC图的纹理分析预测直肠癌局部浸润深度(pT1-2/pT3-4)和淋巴结状态(pN0/pN1-2)的可能性。与其他研究相比,这项初步研究数据集最小,且仅提取8个二维特征,因而结果最差。Yin等[17]采用类似的方法扩充了数据量和提取特征数量,并结合DWI和ADC图共同预测局部浸润和淋巴结受累,所构建的模型将预测局部浸润的AUC提升至0.793,预测淋巴结侵犯的AUC提升至0.802。虽然DWI和ADC图可以提供额外的肿瘤信息,但受限于图像分辨率,难以准确反映肿瘤和肠壁各层结构的位置关系,单独使用可能难以获得最佳效果。因此,You等[9]结合高分辨率T2WI和ADC图提取全肿瘤纹理特征,并使用LASSO和支持向量机(support vector machine,SVM)算法构建术前T分期预测模型,AUC高达0.910,相较于单独使用T2WI和单独使用ADC分别获得了0.065和0.029的提升,进一步表明ADC图在预测直肠癌T分期上的潜力。此外,Sun等[18]针对T1-2和T3a分期,引入fMRI参数结合T2WI影像组学评分共同构建分类模型,结果优于上述所有基于二维ROI开展的研究,表明ADC和动态对比增强磁共振(dynamic contrast enhancement MRI,DCE-MRI)参数有助于改进分期预测性能。针对样本量少的普遍问题,Hou等[19]使用至今最大规模的数据集,同时,通过迁移学习引入超分辨率模型进行预处理,解决了直肠磁共振图像的Z轴和XY轴分辨率差异巨大的问题,对比直接使用原图AUC提升了0.059。
关于直肠癌T分期的研究见表1,其中,多数研究的数据量集中于200例以内,以T2WI为主要研究序列,且三维VOI的效果普遍优于二维ROI。高T期直肠肿瘤会因癌变加剧导致内部纹理复杂化,异质性增大,这与各项研究的特征分析结果相符。其次,高T期直肠肿瘤侵袭性强,浸润深度大,往往在形态上更为不规则,因而,形状特征也有助于提升T分期效果。
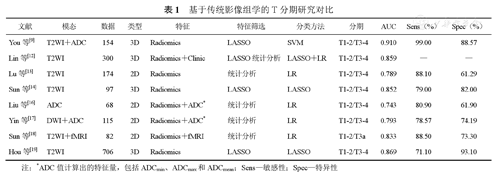
基于传统影像组学的T分期研究对比
基于传统影像组学的T分期研究对比
| 文献 | 模态 | 数据 | 类型 | 特征 | 特征筛选 | 分类方法 | 分期 | AUC | Sens(%) | Spec(%) |
|---|---|---|---|---|---|---|---|---|---|---|
| You等[9] | T2WI+ADC | 154 | 3D | Radiomics | LASSO | SVM | T1-2/T3-4 | 0.910 | 99.00 | 88.57 |
| Lin等[12] | T2WI | 300 | 3D | Radiomics+Clinic | LASSO统计分析 | LASSO+LR | T1-2/T3-4 | 0.859 | — | — |
| Lu等[13] | T2WI | 174 | 2D | Radiomics | 统计分析 | LR | T1-2/T3-4 | 0.789 | 88.10 | 61.29 |
| Sun等[14] | T2WI | 97 | 3D | Radiomics | LASSO | LASSO | T1-2/T3-4 | 0.852 | 79.00 | 82.00 |
| Liu等[16] | ADC | 68 | 2D | Radiomics+ADC* | 统计分析 | LR | T1-2/T3-4 | 0.743 | 80.90 | 61.90 |
| Yin等[17] | DWI+ADC | 115 | 2D | Radiomics+ADC* | 统计分析 | LR | T1-2/T3-4 | 0.793 | 78.57 | 74.19 |
| Sun等[18] | T2WI+fMRI | 82 | 2D | Radiomics+fMRI | 统计分析 | LR | T1-2/T3a | 0.833 | 88.50 | 73.30 |
| Hou等[19] | T2WI | 706 | 3D | Radiomics | LASSO | LASSO | T1-2/T3-4 | 0.869 | 71.10 | 93.10 |
注:*ADC值计算出的特征量,包括ADCmin、ADCmax和ADCmean;Sens—敏感性;Spec—特异性
直肠癌N分期反映肿瘤转移引起区域淋巴结受累的情况,根据转移淋巴结的数量,可分为N1和N2期[3]。淋巴结转移是直肠癌最主要、最常见的转移途径,对治疗方案的选择、局部复发和预后评估等具有重要意义。临床上,使用MRI评估淋巴结转移时多基于淋巴结大小(面积/最小短径)结合形状不规则(毛刺状或小叶状)、边界不清晰和内部信号强度不均匀等标准综合判断[20],但形态特征受评估者主观影响较大,重复性较差[21]。虽然直肠癌N分期是根据转移淋巴结数量直观划分,但是实际中往往难以获取与淋巴结影像诊断结果逐结节对应的病理验证结果。因此,目前的N分期研究大多基于原发肿瘤特征预测整体是否发生淋巴结转移(pN0/pN1-2),直接针对淋巴结的研究很少。
Yang等[22]基于T2WI结合单因素和多因素分析方法初步探索全肿瘤直方图特征与区域淋巴结转移的关系,结果表明,低偏度是直肠癌区域淋巴结转移的独立危险因素,预测AUC可达0.750。但直方图特征只能反映像素整体分布情况,且该研究仅纳入88例患者,数据量偏小。Song等[23]进一步纳入纹理特征分析像素空间位置关系,同时,结合矢状位脂肪抑制和斜轴位T2WI提取二维ROI特征共同构建多视角图像N分期预测模型,将AUC提高至0.772。临床评估时采用的淋巴结形态特征由图像和ROI共同分析确定,相比单纯使用ROI计算出的形状特征,与转移状态的相关性更大,可解释性更强,因此常常用于构建联合模型提升预测效果。Li等[24]使用T2WI和ADC模态分别提取1 376个三维影像组学特征,经LASSO筛选后使用SVM计算影像组学评分,最后加入年龄和淋巴结尺寸并通过多变量Logistic回归分析构建列线图,AUC达0.884。此外,Liu等[25]使用多区域影像组学特征结合淋巴结最大短径、原发肿瘤位置预测淋巴结转移,证明T2WI全肿瘤、DWI全肿瘤和T2WI瘤周直肠系膜3种VOI特征联合临床特征共同建模可以有效提升预测性能。直肠系膜VOI勾画相对容易,且包含肿瘤、淋巴结状态信息,但也存在肠周正常组织的众多混杂信息,该研究未充分探索其有效性,有待进一步单独验证。上述研究多基于原发肿瘤预测淋巴结转移,而Zhu等[26]基于新辅助放化疗前后MRI中多个淋巴结的集合特征预测淋巴结分期(ypN0/ypN1-2),取得了优于肿瘤模型的效果,AUC达0.812。集合特征可令图像中全部淋巴结对应一个分期标签进行分析,避免获取单结节病理分期结果,降低了金标准的数据要求,想法更为新颖。此外,Li等[27]直接使用短径≥3 mm淋巴结的影像组学特征和临床特征联合建模,AUC高达0.94,有助于分析单个淋巴结的转移状态,但是没有直接实现淋巴结分期预测。
直肠癌N分期研究与T分期具有相似的特点见表2。其中,Yang等[22]采用3D VOI的效果偏差可能是因为只分析了直方图偏度。淋巴结转移概率会随T分期升高而逐步增大,因而可以借助原发肿瘤特征预测N分期。结果表明,淋巴结转移与原发肿瘤内部图像特征相关性更强,而与形状特征关联不大,而且引入淋巴结形态特征联合建模能带来性能提升。另有研究表明,由T2 mapping、体素内不相干运动扩散加权成像(intravoxel incoherent motion diffusion weighted imaging,IVIM-DWI)等计算出的定量磁共振参数也与转移淋巴结有较强的相关性[28,29],后续研究可以考虑使用这些特征联合建模。
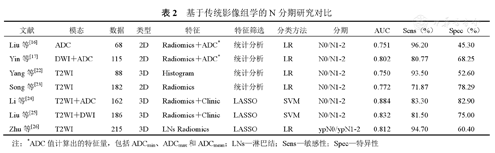
基于传统影像组学的N分期研究对比
基于传统影像组学的N分期研究对比
| 文献 | 模态 | 数据 | 类型 | 特征 | 特征筛选 | 分类方法 | 分期 | AUC | Sens(%) | Spec(%) |
|---|---|---|---|---|---|---|---|---|---|---|
| Liu等[16] | ADC | 68 | 2D | Radiomics+ADC* | 统计分析 | LR | N0/N1-2 | 0.751 | 96.20 | 45.30 |
| Yin等[17] | DWI+ADC | 115 | 2D | Radiomics+ADC* | 统计分析 | LR | N0/N1-2 | 0.802 | 80.77 | 68.25 |
| Yang等[22] | T2WI | 88 | 3D | Histogram | 统计分析 | LR | N0/N1-2 | 0.750 | 93.50 | 52.60 |
| Song等[23] | T2WI | 182 | 2D | Radiomics | 统计分析 | LR | N0/N1-2 | 0.772 | 71.87 | 78.29 |
| Li等[24] | T2WI+ADC | 162 | 3D | Radiomics+Clinic | LASSO | SVM | N0/N1-2 | 0.884 | 83.30 | 82.90 |
| Liu等[25] | T2WI+DWI | 186 | 3D | Radiomics+Clinic | LASSO | SVM | N0/N1-2 | 0.832 | 81.50 | 75.00 |
| Zhu等[26] | T2WI | 215 | 3D | LNs Radiomics | LASSO | LR | ypN0/ypN1-2 | 0.812 | 94.70 | 60.40 |
注:*ADC值计算出的特征量,包括ADCmin、ADCmax和ADCmean;LNs—淋巴结;Sens—敏感性;Spec—特异性
基于传统影像组学的研究普遍存在异质性,各环节的差异都会影响特征的重复性,但这恰恰是进一步临床应用所必须解决的。已有研究表明,在图像获取时,不同中心的硬件和采集参数是图像差异的主要来源,且ADC的变化大于T2WI;在肿瘤勾画时,不同经验专家的勾画结果所提取特征的重复性均较好,但勾画最小最清晰的实体边界所提取的特征鲁棒性更好[11,30,31,32]。就特征而言,一阶特征、形状特征、灰度共生矩阵(gray level co-occurence matrix,GLCM)特征的重复性更好,而灰度游程矩阵(gray level run length matrix,GLRLM)、灰度大小区域矩阵(gray level size zone matrix,GLSZM)和邻域灰度差矩阵(neigbouring gray tone difference matrix,NGTDM)特征易受图像质量、提取软件的影响,重复性较差,上述TN分期模型所选取的特征类别分布也符合该观点。多中心数据的差异依赖相关机构制定统一的标准化成像协议,而为了实现可重复的特征提取,图像生物标志物标准化倡议(image biomarker standardization initiative,IBSI)已给出了标准的特征定义和计算方式。现有的研究大多基于Python和Matlab展开,二者分别提供PyRadiomics和Radiomics包用于特征提取,且均遵循IBSI规范,在保持用户自定义计算设置一致的情况下,可以有效提高跨平台影像组学特征的可靠性[33]。
综上所述,基于多模态MRI的原发肿瘤特征可以有效预测直肠癌TN分期,但是这类方法需要手动勾画肿瘤区域,人工成本太高,难以实现全自动化的诊断。相比之下,深度学习可以通过卷积、池化和全连接等操作从图像中自动提取和选择特征,构建端到端的计算机辅助诊断算法,减轻医生工作负担。
深度学习技术可以从大量数据中自动学习抽象特征,实现复杂的医学图像分析,在结节检测、肿瘤分割、病灶分类和图像配准等诸多任务中应用广泛[34]。目前,基于深度学习直接实现直肠癌TN分期的研究数量有限,但却提供了有价值的探索方向。Wu等[35]将Faster R-CNN应用于直肠癌T分期自动诊断平台,为目标检测算法在直肠癌分期中的应用提供了可能性。Kim等[36]受临床诊断流程启发,通过2个U-Net模型分别分割肿瘤和直肠,再将原图和2张分割结果图组合输入Inception V3模型来区分T2/T3期直肠癌。该方法创新性地将分割模型的推理结果用于分类,充分利用了分割图中包含的肿瘤位置和形状信息,超越了仅使用原图的模型效果,AUC高达0.96。上述研究表明,分割、检测任务可以作为前处理步骤辅助实现直肠癌TN分期。因此,分别对深度学习方法在辅助T分期肿瘤分割和辅助N分期淋巴结诊断方面的研究进行介绍。
近年来,直肠癌原发肿瘤分割算法主要围绕T2WI展开,分为使用具有诊断价值的2D切片图像和全肿瘤3D图像2类研究,且大多采用Dice相似系数(Dice similariy coefficient,DSC)评估网络分割结果与金标准的重叠程度。Trebeschi等[37]通过配准和通道拼接融合T2WI和DWI,构建基于二维patch的体素级CNN分类器,DSC达0.68/0.70。虽然该模型可以通过类似滑动窗口的方法实现三维分割,但是patch间的重叠冗余会使分割效率低下。相比之下,全卷积网络(fully convolutional networks,FCN)采用全卷积架构和跳跃连接,将语义信息与表观信息相结合,实现端到端的语义分割,大幅提升了分割效率。Soomro等[38]通过先使用三维FCN进行粗分割,再结合水平集算法细化轮廓和边缘,DSC高达0.937 8。U-Net是FCN的改进结构,由捕获上下文的收缩路径和提供精确定位的对称扩展路径组成,同层级特征图间的跳跃连接将低层次的细节信息引入高层次特征,进一步提升了分割精度。Wang等[39]和Zhu等[40]分别使用基础U-Net分割T2WI和DWI中的直肠肿瘤,DSC为0.74和0.675,后者效果更差,可能是由于3D网络需要更多的数据训练。
由于直肠肿瘤的位置多变,大小不一,简单的网络难以精确分割,因此诸多研究采用多尺度信息融合方案提升效果,而如何设计合理的提取和融合结构是其中的一大重点。常见的网络大都使用卷积层提取图像特征,配合池化操作压缩特征,减轻网络计算量,从而产生了多尺度特征图。由此引出的一个思想是从不同层级的输出提取多尺度信息再经通道拼接融合,Jian等[41]和Wang等[42]分别基于VGG-16和ResNet50结合side-output结构从Block间提取多尺度特征信息融合建模,DSC分别为0.835 6和0.823 9。Men等[43]采用逐级串联的方法结合ResNet-101主干、级联空洞卷积和空间金字塔池化构建网络,使用高层特征图提取多尺度信息,优化了计算速度。此外,Zheng等[44]通过设计双通道分离卷积分支并加入残差连接,改进了U-Net内部的连续卷积层模块,在同一编码层中实现了多尺度信息融合,避免了不同编解码层之间特征融合造成的信息丢失,使网络可以学习更多的肿瘤边界信息。除了模型主体、输出和内部模块间的多尺度方案外,Soomro等[45]在图像输入端通过池化建立粗和细尺度2条特征提取路径,并结合细层和粗层之间的多尺度密集互连,充分融合双路径的上下文信息,再配合水平集后处理算法,DSC可达0.858 5。
相较于单任务,多任务学习能获得更为丰富的关联信息,可能带来效果上的提升。Huang等[46]使用混合损失函数和多分辨率模型集成策略构建了结合轮廓和区域分割的多任务学习框架HL-FCN,可以在保持边界细节的同时抑制假阳,DSC达0.721。在此基础上,Huang等[47]进一步提出联合ROI定位和分割的三维RU-Net框架,该框架采用共享的全局图像编码器实现基于全局信息的ROI定位,并基于金字塔形区域内特征使用局部区域解码器进行ROI分割,将DSC进一步提升至0.755。Li等[48]引入注意力机制改进U-Net基础网络并融合多尺度信息同时分割肿瘤和预测轮廓,实现了直肠癌的有效分割。
基于深度学习的T分期肿瘤分割研究对比如表3所示,其中多数研究的数据量在200例以下,结果存在过拟合的可能。虽然U-Net在医学图像分割上的有效性已经得到了广泛研究,但基础U-Net结构在直肠癌分割上的表现不佳,引入注意力机制和多尺度结构,可以使模型聚焦关键位置,排除周围相似组织的干扰,提升分割效果。
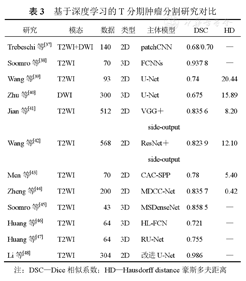
基于深度学习的T分期肿瘤分割研究对比
基于深度学习的T分期肿瘤分割研究对比
| 研究 | 模态 | 数据 | 类型 | 主体模型 | DSC | HD |
|---|---|---|---|---|---|---|
| Trebeschi等[37] | T2WI+DWI | 140 | 2D | patchCNN | 0.68/0.70 | — |
| Soomro等[38] | T2WI | 70 | 3D | FCNNs | 0.937 8 | — |
| Wang等[39] | T2WI | 93 | 2D | U-Net | 0.74 | 20.44 |
| Zhu等[40] | DWI | 300 | 3D | U-Net | 0.675 | 15.89 |
| Jian等[41] | T2WI | 512 | 2D | VGG+side-output | 0.835 6 | 8.20 |
| Wang等[42] | T2WI | 568 | 2D | ResNet+side-output | 0.823 9 | 12.10 |
| Men等[43] | T2WI | 70 | 2D | CAC-SPP | 0.78 | 5.40 |
| Zheng等[44] | T2WI | 200 | 2D | MDCC-Net | 0.835 7 | 0.42 |
| Soomro等[45] | T2WI | 43 | 3D | MSDenseNet | 0.858 5 | — |
| Huang等[46] | T2WI | 64 | 3D | HL-FCN | 0.721 | — |
| Huang等[47] | T2WI | 64 | 3D | RU-Net | 0.755 | — |
| Li等[48] | T2WI | 304 | 2D | 改进U-Net | 0.986 | — |
注:DSC—Dice相似系数;HD—Hausdorff distance豪斯多夫距离
放射科医生在评估直肠癌N分期时,会对整个MRI图像中存在的淋巴结进行检测、分类和选择,以明确转移淋巴结的数量和位置,进而确定最终分期。与之类似,现有的深度学习技术在N分期中的应用大多关注淋巴结辅助检测和分类,虽然不能直接帮助医生实现N分期,但能减少淋巴结漏诊和误诊概率,降低分期难度。
Faster R-CNN将特征检测、候选区域生成、区域图像分类和位置细化4个基本的目标检测和识别过程集成到一个统一的深度卷积网络中,与转移淋巴结诊断流程类似。Lu等[20]使用盆腔MRI平扫图像中的抑脂T2WI和DWI构建数据库,借助迁移学习的方法微调预训练的Faster R-CNN模型,检测短径≥3 mm的转移淋巴结,AUC高达0.912。Mask R-CNN是在Faster R-CNN的基础上添加分割分支并修改损失函数和对齐方法改进而来,可以在检测的同时提供分割结果。Zhao等[49]将T2WI和DWI经配准、通道叠加后融合成三通道图像,再使用Mask-RCNN网络对图像中短径≥3 mm的淋巴结同时进行检测和分割。但由于淋巴结标注使用3位医生的综合建议产生,良恶性缺乏病理金标准,故该研究没有进一步区分淋巴结的良恶性。Li等[50]将放射科医生标注的直肠系膜和直肠上动脉区域的淋巴结逐一匹配病理良恶性结果,然后,采用迁移学习的Inception-V3模型构建分类网络,进一步实现了淋巴结状态的鉴别。在此基础上,Li等[51]使用11个深度预训练模型开展结直肠癌淋巴结分类的研究,通过大量实验明确了最小数据集大小和最小训练次数,为后续研究建立了深度迁移学习的基准。
综上所述,深度学习技术可以在辅助原发肿瘤分割和辅助淋巴结诊断方面提供有力的支持,有助于简化分期流程,提高分期诊断效率,但现有研究的数据集规模仍达不到大数据集的要求,并且欠缺多设备、多中心方面的丰富度,难以确定算法在实际应用中的泛化性能。
本文回顾并总结了近年来基于多模态磁共振图像的直肠癌TN分期方法,其中,T2WI是TN分期的主要依据,DWI和ADC可以提供额外的信息辅助分期。现有的直肠癌术前TN分期算法主要通过传统影像组学技术提取肿瘤区域特征构建分类模型进行预测,而深度学习技术则集中于原发肿瘤分割、淋巴结诊断等辅助分期任务。未来直肠癌分期的研究方向主要有以下方面:(1)收集多中心、多设备、多模态的直肠癌MRI数据,构建标准的大规模基础数据集,为后续分期算法研究提供统一的评价基准;(2)探索合理有效的多模态信息融合策略,充分利用图像中的肿瘤诊断互补信息提升网络鉴别能力;(3)统一分割/检测和分类任务,构建多尺度和多任务联合分期模型,输出多样化的诊断结果;(4)融合传统影像组学技术和深度学习技术搭建模型,实现直肠癌全自动分期诊断。
所有作者均声明不存在利益冲突



























