
版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
二代测序(NGS)又称大规模平行测序,能够同时对上百万甚至数十亿个DNA分子进行测序,实现了大规模、高通量测序的目标,是继Sanger测序之后的革命性进步。目前在临床肿瘤实践中,NGS主要应用于驱动基因测序,是肿瘤精准诊疗的重要环节。中国临床肿瘤学会于2015年9月17日在厦门市召集了我国具有代表性的肿瘤学专家、病理及分子生物学专家及NGS技术专家,成立了中国肿瘤驱动基因分析联盟(CAGC)并启动了精准肿瘤学研究项目(CAGC-POI),确立了整体项目的组织框架和内容框架。CAGC-POI计划第一阶段拟在初步建立NGS技术临床应用共识的基础上,在肺癌、乳腺癌、肝癌、胃癌、结直肠癌、造血系统肿瘤等恶性疾病开展以NGS技术为主的驱动基因谱分析。在上述共识建立、主要6个瘤种的变异谱分析验证、优化流程的基础上,将在携带有临床可靶向作用驱动基因的患者中,开展相应的靶向精准诊治的实践与试验研究。最终提出适合我国临床肿瘤学实践的NGS技术标准和规范,提高为肿瘤患者服务的质量和能力。
NGS有明显的通量优势及发现未知基因变异的优势,但是在使用中也存在包括检测技术、数据管理、分析与报告、解读与临床咨询等方面的各种挑战。在我国基因检测已经成为临床精准诊治的前提,但缺乏统一的关于NGS的应用共识。在未来,肿瘤临床实践中甚至可能需针对外显子组、转录组、全基因组、表观遗传组的信息分析和应用。可以预见的是,NGS高通量技术将会在检测技术、分析内容、解读等方面一直产生变化,也会带来在实践应用中的各种挑战。
2017年,在美国来自商业公司与学术机构的医学NGS服务得到美国食品药品监督管理局(FDA)的认可,提示NGS已正式进入临床实践应用,同时对NGS各种医学服务的审核进度在加快[1]。
在我国现阶段,肿瘤患者数目庞大,驱动基因诊断和检测应用需求巨大。但在实际应用中会因为检测技术的准入门槛低、缺乏临床公认的NGS指导规范,肿瘤诊治中可能存在检测质量低下、结果准确性差,检测结果解读不专业,甚至盲目追求基因检测结果的现象。因此,我国肿瘤学界亟需对当前NGS在临床合理规范的使用进行充分讨论,形成共识予以发布,以指导我国临床肿瘤诊疗中的规范应用。
本技术共识阐述了NGS技术质量需求、临床肿瘤相关NGS检测内容、样本处理、测序流程、数据管理、信息学分析、结果报告解释和咨询等多个方面的内容。本共识也对患者知情同意、测试项目的质控、研究与诊断的应用差别等内容进行了说明。文中的"声明"是专家组讨论的各个要点,也即专家组形成的共识或推荐。
NGS技术正处于不断的发展和完善过程中,需对NGS技术现状有正确的认识。在过去十年肿瘤学界的研究和应用显示:在恰当的技术条件下,NGS确实能够高通量地检测分析肿瘤中的驱动基因变异或治疗性靶点,给部分患者带来治疗和生存的获益。
在临床实践中各实验室可能有不同的NGS技术平台。无论使用何种平台,各实验室均应按照本共识中的推荐(声明)开展NGS项目测试,对平台及相关分析工具进行验证后才应用于临床。
针对肿瘤的多基因检测,需设计特定的NGS测试项目。这些项目的检测结果应能够明确回答临床诊疗所需的基因状态。需明确指出的是,应避免分析技术能力尚未得到充分验证的NGS项目在临床应用。
声明1:临床肿瘤诊断应用的NGS项目应符合相应的诊断技术标准
未进行充分分析和临床验证的NGS技术不应在临床实践的驱动基因诊断中应用。相关实验室应明确能够提供的检测项目的内容范围和相关技术参数。
NGS用于临床肿瘤分子诊断能够带来明确的益处。NGS技术优势:一次性、特定时间(约10个工作日)产生覆盖基因组特定区域(从数个基因到数百个基因以至外显子组、转录组或全基因组)的通量数据,同步获得多基因、多位点、多种变异形式的分子检测结果。尽管目前NGS的多基因多重分析总成本较高,但相对于传统检测方法而言,单个基因单个位点的检测费用已大大下降。
影响NGS结果的限制性因素包括:平台类别、靶区域富集方法、文库构建及扩增效率、测序数据量、生物信息学分析流程等。在临床应用之前,对于特定突变位点的NGS检测结果在低质量数据条件下应采用其他方法进行结果验证。检测实验室的专家、肿瘤患者的主诊医师与患者应就开展肿瘤NGS分析的利弊进行充分沟通和知情同意,同时让临床医师了解特定NGS测试项目的医学意义。
声明2:NGS检测项目的目的和临床益处应明确
如果是用于临床靶点抑制剂(靶向药物)的使用决策参考,检测结果应能够明确一些在临床可采取特定药物靶向治疗的靶点变异信息。NGS测试项目如果是用于分子分型、疗效预测或预后判断则需在发现集的基础上建立分析模型,再通过技术和验证集的验证方可进一步转化应用于临床。
在临床实践中,不同的瘤种既可存在瘤种特异性的驱动基因变异,也可能存在共同的基因变异。一个瘤种存在众多的分子变异亚型,从而提出"同病异治"或"分型而治"的策略;不同瘤种之间由于存在共同的驱动基因分型,则可以采取"异病同治"的策略。由此可见,临床肿瘤的基于基因或基因组学的分子分型将逐步超越传统的病理组织形态学分型,成为临床诊治肿瘤的关键环节。
肿瘤的多基因分型对药物研发策略也产生了重大影响。在药物临床试验设计方面,近年来出现了两种基于分子靶点的试验研究设计类型。一种是"雨伞"设计类型,即针对单个瘤种的基于多个驱动基因分型及其靶向治疗,另一种则是"篮子"设计类型,是针对多个瘤种的驱动基因的同型同治。
无论是临床实践还是临床试验,用于基因分型的NGS检测项目内容均需精细的设计和技术可靠性的验证。NGS项目的检测内容应明确检测哪些基因、相应基因的受检区域、靶点的变异信息等。
声明3:如果NGS用于临床分子诊断,应明确至少哪些基因需纳入检测名单中
这个必须的"核心基因清单"需特定肿瘤亚专科相关的临床和实验室多学科专家共同讨论后制定。
核心基因清单论证及制定过程中应以患者的利益为中心,充分整合临床肿瘤学家对于精准诊治的观点,并充分考虑在我国推广应用的可操作性。以肺癌为例:目前美国国家综合癌症网络(NCCN)及国内肺癌临床指南、共识[9]或卫计委诊疗规范都明确指出表皮生长因子受体(EGFR)基因突变、鼠类肉瘤病毒癌基因(KRAS)突变、间变性淋巴瘤激酶(ALK)基因重排(融合)、c-ros肉瘤致癌因子-受体酪氨酸激酶(ROS1)基因重排(融合)、间质上皮转移因子(MET) 基因14号外显子可变剪切变异、MET拷贝数扩增、转染重排(RET)基因重排(融合)、人类表皮生长因子受体2(HER2)基因突变或扩增、鼠类肉瘤滤过性毒菌致癌同源体B1(BRAF)基因突变等驱动基因变异是临床实践中的重要必须基因。
从CAGC-POI项目的恶性肿瘤的整体角度考虑,检测内容(基因组合及其基因组区域)应既包括各瘤种特异性的组合,也应备有一个大而全的基因组合。因实体瘤和血液肿瘤的变异谱方面的差异较大,因此,在大而全的组合中对实体瘤和血液肿瘤应分别设计。
可靶向作用的原理及定义:驱动基因的定义是参与肿瘤发生发展、转移、耐药等疾病进程中关键步骤的基因。可靶向作用则是指针对该驱动基因的变异靶点或其上下游关键分子,能够采用靶向药物等手段进行治疗,从而达到抑制肿瘤进展或消退肿瘤的治疗效果。可靶向作用基因也可指该基因的变异可以用来作为明确的分子亚型诊断或预后判断。
声明4:可靶向作用基因的变异可能会影响临床决策,医师需根据基因分型的检测结果来指导临床诊疗方案
可靶向作用基因的变异可分为两大类,第一类包括国际、国内指南[中国临床肿瘤协会(CSCO)、美国临床肿瘤学会(ASCO)、NCCN、欧洲医学肿瘤学会(ESMO)等]中明确指定的、药物标签中说明的、与FDA及我国国家食品药品监督管理总局(CFDA)批准的适应证相关的临床分型基因变异,或是具有明确的分子诊断或预后判断价值的基因变异。第二类包括正在开展的任何期别(Ⅰ~Ⅲ期)临床试验中的药物相关靶点、在已经或即将开展的临床试验中作为入组条件的变异靶点、在国内外指南中其他肿瘤或疾病适应证的药物靶点、用于辅助诊断或预后判断的靶点等。
在NGS检测中,对于临床意义非常明确的体细胞突变的少数基因集分析,可以直接检测肿瘤标本而无须设置白细胞等对照样本。但对于需包含上述第二类可靶向作用基因的更大的基因集分析,尤其是数百基因乃至全外显子组测序等,建议在肿瘤样本进行NGS检测时设置患者自身白细胞样本对照分析。
声明5:应根据实际情况需对检测内容进行更新
随着肿瘤发生发展机制研究的不断深入,新的靶点及药物会得到不断的发现和研发。因此,有必要定期或不定期依据重要的临床研究进展对检测内容进行讨论并发布更新。
声明6:应对每个基因变异位点进行明确定义注释
检测基因数量越多,诊断信息会越多。但由于肿瘤信号通路的冗余性和复杂性,在基因清单中,除了核心基因之外,应对其他增加的每个基因进行明确定义注释,说明其基因型与潜在可靶向作用位点或通路的关系。在实际的NGS测试项目应用中,检测基因数量的增加不应以核心基因的检测质量受损为代价。
技术参数验证对保证检测结果的准确性很重要。所有NGS计量参数都应明确说明。在检测中应记录批次或分析样本特异性的特征。检测实验室应利用结构化的数据库来保证平台、各分析项目、样本处理等方面的质量分析。在检测分析流程中,每个样本都应做好唯一性标识。在平台稳定性验证中,应使得所有仪器和试剂都符合标准要求,提供特定驱动基因的准确性和精确性。
技术验证过程中应记录在案的相关内容或参数包括以下几方面:(1)样本相关的患者信息保护原则、唯一性标识、接收、存储、处理各步骤内容及时间;(2)实验操作过程中DNA/RNA处理每一步骤所用试剂名称、生产厂家、批号,记录每一步操作后的质量指标(如RNA的完整性数值等);(3)数据分析及数据库管理相关的软件(包括版本号,分析时所用各项参数)、数据的相关参数(数据量大小,查询缓存等)、数据库的安全性和备份情况等。
声明7:NGS项目的检测流程和生物信息学分析需针对特定平台和分析项目进行充分优化和标准化
流程验证中应提供分析敏感性和特异性的参数。因为分析方法的差异,针对单碱基位点变异(SNV)、插入或缺失(INDEL)、重排(融合)、拷贝数变异(CNV)等不同的变异应分别测试技术的敏感度和特异度。受样本取材或探针设计等因素影响或限制,NGS在某些变异类型如拷贝数扩增的检测方面有时存在一定的局限性。对于NGS方法准确性可能有影响的情况,应考虑采用可靠的单基因方法对检出变异进行验证。
NGS检测需文库构建,该步骤原始材料为DNA或来自RNA的cDNA。适合临床NGS分析的样本类型优先选择新鲜组织标本,也可选用甲醛固定-石蜡包埋(FFPE)样本、肿瘤细胞学标本及血浆(游离DNA/RNA)等。
NGS可用于疾病初诊时的分子靶点分析,也可用于治疗中疾病进展状态的监测及耐药分子机制的诊断。因而在疾病的全程管理中可合理地考虑在诊疗的关键时间节点收集临床标本进行NGS分析。决定是否采用NGS在多个时间节点的检测分析应做好多学科讨论和充分的知情同意。
各类样本采集:(1)对于手术和活检的新鲜组织:理想的保存方法是迅速置于液氮中,可保存于液氮罐或-80 ℃冰箱,该过程应在手术离体后30 min内完成,以防止RNA等核酸降解;或采用保存在样本保护剂中,尽早转移到-80 ℃冰箱保存。可采用冷冻切片染色评估样本中的肿瘤细胞含量。(2)FFPE:按病理操作规范进行取材。手术或活检离体的组织应在30 min内浸入100 ml的4%甲醛溶液中进行固定,避免使用酸性及含有重金属离子的固定液。大标本应切开后充分固定至6~48 h,不超过72 h。小活检标本可固定6~12 h。开展NGS检测前应进行HE染色评估肿瘤细胞的含量。(3)细胞学样本:脱落细胞学标本及细针穿刺细胞学标本用于基因检测时,必须进行病理质控,确定标本中的肿瘤细胞数量及与正常细胞的比例,符合质量要求或通过肿瘤细胞富集处理后符合要求的标本可直接抽提核酸,也可以制备成FFPE细胞学蜡块进行后续分析。体腔积液标本可提取无细胞上清标本中的循环肿瘤DNA(ctDNA)进行检测。(4)血浆或血液样本:ctDNA是存在于血浆中的游离DNA,肿瘤来源的DNA占血浆游离DNA的比例在不同肿瘤及病例中相差悬殊。采集外周血提取血浆游离DNA进行检测,取样时应使用一次性的含乙二胺四乙酸(EDTA)的抗凝真空采血管,采集8~10 ml全血,冷藏运输,2 h内分离血浆,提取游离DNA,保存于-80 ℃冰箱中,并避免反复冻融。如外周血需长时间运输,建议用商品化的游离DNA样本专用保存管,在常温条件下,ctDNA在全血中可稳定保存3~7 d。由于血细胞基因组DNA的潜在大量释放会极大地稀释血浆游离肿瘤DNA的浓度,经肉眼观察确认为溶血的样本不适用于对游离肿瘤DNA的NGS检测。当怀疑血浆游离DNA受到血细胞基因组DNA污染时,可考虑采用核酸片段大小分布分析来判断污染是否存在,而从判断样本是否适用于NGS检测。
采样质量的评价对后续结果的解读有重要意义,临床样本的采样过程、分析前的运输和处理流程以及病理的评估结果等均应做相应的记录。质量评价的内容应包括肿瘤细胞含量比例、肿瘤细胞的数量,是否按照要求进行处理与运输等。评价方法包括肉眼观察、显微镜下观察和血浆样本核酸浓度分析等。对于肿瘤细胞分布集中的区域应进行标注,必要时进行显微切割后再提取核酸。
样本采集和处理中的防污染:样本采集最好采用一次性材料;制备不同患者病理切片样本时,需更换新刀片,并清除操作器皿或切片机上先前样本的残留。
样本运送和保存:实验室应建立详细的样本运送标准操作规程(SOP),对临床医师提供样本采集手册,要求物流人员填写相关运送记录表,确保运送过程中各类样本的安全性和过程的可控性。石蜡材料可常温运输,血浆样本干冰条件下运输,核酸样本需在4 ℃或冷冻条件下运输。
声明8:为提高NGS检测肿瘤驱动基因的准确性,应提供高质量的临床生物材料样本
不同类型生物材料的收集及处理均应有明确的SOP指导。
声明9:样本登记过程中每个标本应在NGS分析中有唯一性标识
包括来自同一患者的不同组织病灶、不同组织切片、不同核酸样本等均有唯一性的标识。
声明10:应有充分的样本评估和起始核酸用量
除了血浆或血液来源的DNA/RNA,凡是涉及形态学的标本,应在显微镜下评估肿瘤细胞的含量,充分记录各类形态学恶性细胞的含量比例。评估记录还应包括标本收集和处理方法。肿瘤细胞比例低的标本必要时应采用包括显微微切割在内的有效富集肿瘤细胞的方法,提高肿瘤细胞比例。一般情况下,适合于NGS检测的组织标本中肿瘤细胞含量建议应达到20%以上。如果肿瘤细胞占比较低,患者知情后依然愿意进行NGS检测,应尽量采用显微切割技术富集肿瘤细胞,或增加NGS测序数据量以提高测序覆盖深度,并在报告中提示样本局限性。组织样本抽提获得的DNA量应达50 ng以上用于NGS检测。血液样本建议采血至少8 ml,分离血浆游离DNA用于NGS检测。
声明11:应对抽提后和文库构建后的核酸样本进行质控
组织DNA与血浆游离DNA等核酸样本均应进行检测前的质量控制分析,包括核酸的浓度和纯度分析。组织DNA样本应进行核酸完整性分析,以判断DNA质量。血浆游离DNA样本应进行片段长度分布分析,以判断是否存在血细胞基因组DNA的污染。在文库构建后应对核酸样本进行浓度测定与文库片段分布等质量控制。
声明12:应预留生物材料用于后续验证
为了便于后续采用其他技术方法对检测结果进行验证,在样本留取时,应适当考虑增加生物材料的留取量。在石蜡标本的切片过程中可考虑适当同步预留几张切片用于后续免疫组化、荧光原位杂交、聚合酶链锁反应(PCR)测序等方法验证。
在临床肿瘤诊断和监测中的NGS应用应充分关注试剂管理、样本的质控/质量保证、检测环境、人员资质和熟练程度、NGS技术参数标准(包括文库构建、测序流程、质控等)等环节,以SOP加以约束,以保证特定肿瘤NGS测试能够稳定准确地发挥作用。
测序技术的各个环节应做好实验室规范记录,最好有结构化的数据库进行管理,包括平台环境参数、试剂使用和性能、样本追踪、质控数据等管理。通过日常和定期分析质控保证检测的准确性。
为满足临床需求,实验室需对各个质量控制步骤出现异常、失败的样本规定合理的备选方案。比如采用其他分子诊断平台检测一部分重要位点,或对阳性位点采用其他平台确认性检测,或者重复高通量测序流程等,有条件的检测单位也可以重新选取标本。
声明13:NGS检测需符合严格的检测环境要求
各类样本的储存、核酸抽提处理及NGS分析应在通过审核验收的临床基因扩增检验实验室完成。
声明14:应对NGS试剂进行严格的管理和质控
NGS试剂应独立存放,防止交叉污染。对于实验室自配试剂或探针及采购试剂,均需做好每批试剂的性能评价,评价合格后才可用于临床样本分析。
声明15:每个特定NGS测试项目均需经过严格技术验证合格后才可用于临床肿瘤检测
诊断测试项目必须在准确性、分析敏感度、特异度、精确性等方面进行充分评价。如果检测项目中的试剂或步骤有任何主要变更,则质量参数需重新评价。在针对检测项目更新或升级的重新评价中,应明确测试所需的样本类型及例数。强烈建议各实验室参加NGS相关的室间质量评估项目来评测检测能力和质量。
声明16:应制定有明确的检测流程
这个检测流程包括样本质控/质量保证、样本预处理、接头连接、预扩增、基因组合靶区的捕获、靶区纯化、扩增文库构建、质控定量后上NGS测序仪检测等步骤。每个步骤的操作和质量控制都应建立SOP进行指导。如果使用扩增子建库的方式,同样需建立相应的SOP进行质控管理。
声明17:NGS技术检测项目应达到标准化
遵照相关技术规范,使得上述样本预处理至文库构建、测序、质控的技术标准化,也包括原始数据产生和管理、信息学分析过程的标准化。由此CAGC将在下一阶段可行性验证基础上形成一套NGS技术SOP。
声明18:人员配备和熟练程度应满足实践需求
应配有专门的质控人员。对于参与NGS实验流程操作的技术人员进行充分的培训,达到一定的熟练程度。每个平台应有技术员专职从事实验操作。
声明19:需使用核心基因清单与相应的检测标准品用于各个实验室间的质控比较
根据检测驱动基因的突变结果描述、目的区域的覆盖度等建立评分体系。这个评分体系将用于对实验室间NGS检测能力的分析与认证。
由于NGS数据庞大,数据管理、储存和分析需强大的计算平台支持。数据的质量检查、信息学分析、输出格式、储存等方面都应有SOP支持。最好有结构化的数据库来管理原始数据、质控数据以及分析结果数据等。
声明20:应对所采用的数据分析工具(软件)进行能力验证
需采用适量例数的原始数据针对NGS数据分析工具进行验证分析。通过采用携带不同种类与丰度变异的数据来验证整个分析流程的准确性与稳定性。特别是在增加检测基因的内容时需验证分析工具的可靠性。分析工具的能力验证应有详细的内部软件验证记录。CSCO、CAGC也将组织开展软件分析能力验证。
声明21:检测实验室应规范数据产生和管理储存流程
检测实验室必须采用结构化的数据库来注释单碱基位点变异、插入或缺失、重排(融合)、拷贝数变异等各类变异信息。
声明22:数据储存应采用通用的FASTQ、BAM、VCF格式,便于数据交换及实验室间评价
同时保存完整的日志文件,便于区别流程版本信息、追溯异常结果来源及分析原始数据向诊断报告生成的可重复性。诊断实验室应长期保存相关的数据集(至少15年)。
声明23:NGS原始数据的质量检查,应有严格的操作规程指导
所有参数需与检测性能评价过程中的参数进行比较,设置并执行接受与拒绝的标准(表1)。

FASTQ和BAM质量的计量参数
FASTQ和BAM质量的计量参数
| 参数 | 描述 | |
|---|---|---|
| FASTQ的计量参数 | ||
| ·每循环的中位碱基质量 | ·在序列片段末端碱基测序质量会急剧降低。Phred序列质量评分不能<20 | |
| ·重复片段占比 | ·重复测序片段占比是反映文库构建的复杂度的指标 | |
| ·去除接头序列碱基的占比(如果适用) | ·在去除接头序列时,去除的碱基所占比例是反映序列质量的指标 | |
| ·比对成功的片段占比 | ·能够比对到参考基因组序列上面的测序片段的比例 | |
| ·目标区域片段占比 | ·能够比对到富集目标基因组区域的片段在所有测序片段中所占的比例 | |
| ·目标区域的平均测序深度 | ·符合临床需求的目标检测区域中所有位点的平均测序深度 | |
| ·目标区域测序深度的分布 | ·在符合临床需求的目标区域所覆盖的所有位点中,绘制测序深度分布图或不同测序深度覆盖位点的占比表格 | |
| BAM计量参数 | ||
| ·总变异数量 | ·符合临床需求的目标区域中检出的总变异数目应当与同类患者群体且采用相同捕获区域以及处理方法相同的样本结果相类似 | |
| ·已知的多态性比例 | ·每个样本中大部分(> 90%)被检测到的变异都应是已知的基因多态性 | |
| ·插入或缺失变异比例 | ·插入或缺失变异占总变异数的比例 | |
| ·纯合型变异的比例 | ·纯合型变异占总变异数的比例 | |
| ·无义突变比例 | ·无义突变占总变异数的比例 | |
| ·转换/颠换比值 | ·转换/颠换突变之间的比值 | |
注:FASTQ:一种存储了生物序列(通常是核酸序列)以及相应质量评价的文本格式;BAM:序列比对之后常用的输出格式
声明24:不同瘤种的NGS数据分析应遵循相同的基本流程
分析流程包括初步分析、接头序列去除、引物序列去除、低质量序列去除、参照基因组序列比对、去重、插入或缺失重复比对、碱基质量得分校正、突变识别、注释、过滤后输出等步骤(表2)。CAGC-POI项目需在各个瘤种中的每一步都应做到同质性。
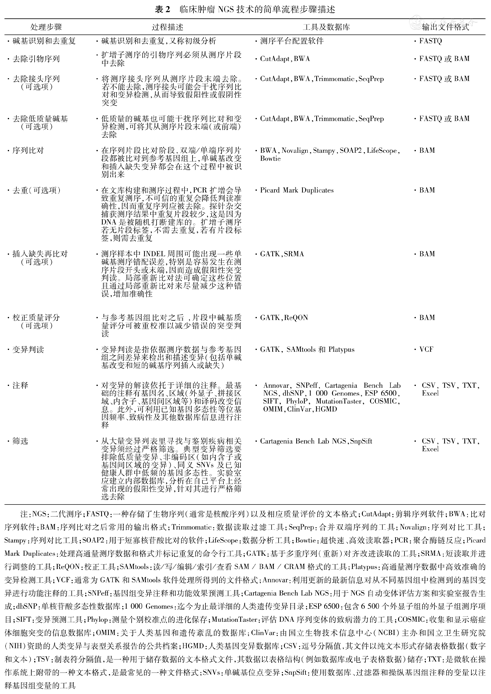
临床肿瘤NGS技术的简单流程步骤描述
临床肿瘤NGS技术的简单流程步骤描述
| 处理步骤 | 过程描述 | 工具及数据库 | 输出文件格式 |
|---|---|---|---|
| ·碱基识别和去重复 | ·碱基识别和去重复,又称初级分析 | ·测序平台配置软件 | ·FASTQ |
| ·去除引物序列 | ·扩增子测序的引物序列必须从测序片段中去除 | ·CutAdapt,BWA | ·FASTQ或BAM |
| ·去除接头序列(可选项) | ·将测序接头序列从测序片段末端去除。若不能去除,测序接头可能会干扰序列比对和变异检测,从而导致假阳性或假阴性突变 | ·CutAdapt,BWA,Trimmomatic,SeqPrep | ·FASTQ或BAM |
| ·去除低质量碱基(可选项) | ·低质量的碱基也可能干扰序列比对和变异检测,可将其从测序片段末端(或前端)去除 | ·CutAdapt,BWA,Trimmomatic,SeqPrep | ·FASTQ或BAM |
| ·序列比对 | ·在序列片段比对阶段,双端/单端序列片段都被比对到参考基因组上,单碱基改变和插入缺失变异都会在这个过程中被识别出来 | ·BWA,Novalign,Stampy,SOAP2,LifeScope,Bowtie | ·BAM |
| ·去重(可选项) | ·在文库构建和测序过程中,PCR扩增会导致重复测序,不可信的重复会降低判读准确性,因而重复序列应被去除。探针杂交捕获测序结果中重复片段较少,这是因为DNA是被随机打断建库的。扩增子测序若无片段标签,不需去重复,若有片段标签,则需去重复 | ·Picard Mark Duplicates | ·BAM |
| ·插入缺失再比对 (可选项) | ·测序样本中INDEL周围可能出现一些单碱基测序错配误差,特别是容易发生在测序片段开头或末端,因而造成假阳性突变判读。局部重新比对法可确定这些位置且通过局部重新比对来尽量减少这种错误,增加准确性 | ·GATK,SRMA | ·BAM |
| ·校正质量评分(可选项) | ·与参考基因组比对之后 ,片段中碱基质量评分可被重校准以减少错误的突变判读 | ·GATK,ReQON | ·BAM |
| ·变异判读 | ·变异判读是指依据测序数据与参考基因组之间差异来检出和描述变异(包括单碱基改变和短的碱基序列插入或缺失) | ·GATK, SAMtools和Platypus | ·VCF |
| ·注释 | ·对变异的解读依托于详细的注释。最基础的注释有基因名、区域(外显子、拼接区域、内含子、基因间区域等)和译码改变信息。此外,可利用已知基因多态性等位基因频率、致病性及其他数据库信息进行注释 | ·Annovar,SNPeff,Cartagenia Bench Lab NGS,dbSNP,1 000 Genomes,ESP 6500,SIFT,PhyloP,MutationTaster,COSMIC,OMIM,ClinVar,HGMD | ·CSV,TSV,TXT,Excel |
| ·筛选 | ·从大量变异列表里寻找与鉴别疾病相关变异须经过严格筛选。典型变异筛选要排除低质量变异、非编码区(如内含子或基因间区域的变异)、同义SNVs及已知健康人群中低频的基因多态性。实验室应建立内部数据库,分析在自己平台上经常出现的假阳性变异,针对其进行严格筛选去除 | ·Cartagenia Bench Lab NGS,SnpSift | ·CSV,TSV,TXT,Excel |
注:NGS:二代测序;FASTQ:一种存储了生物序列(通常是核酸序列)以及相应质量评价的文本格式;CutAdapt:剪辑序列软件;BWA:比对序列软件;BAM:序列比对之后常用的输出格式;Trimmomatic:数据读取过滤工具;SeqPrep:合并双端序列的工具;Novalign:序列对比工具;Stampy:序列对比工具;SOAP2:用于短寡核苷酸比对的软件;LifeScope:数据分析工具;Bowtie:超快速、高效读取器;PCR:聚合酶链反应;Picard Mark Duplicates:处理高通量测序数据和格式并标记重复的命令行工具;GATK:基于多重序列(重新)对齐改进读取的工具;SRMA:短读取并进行调整的工具;ReQON:校正工具;SAMtools:读/写/编辑/索引/查看SAM / BAM / CRAM格式的工具;Platypus:高通量测序数据中高效准确的变异检测工具;VCF:通常为GATK和SAMtools软件处理所得到的文件格式;Annovar:利用更新的最新信息对从不同基因组中检测到的基因变异进行功能注释的工具;SNPeff:基因组变异注释和功能效果预测工具;Cartagenia Bench Lab NGS:用于NGS自动变体评估方案和实验室报告生成;dbSNP:单核苷酸多态性数据库;1 000 Genomes:迄今为止最详细的人类遗传变异目录;ESP 6500:包含6 500个外显子组的外显子组测序项目;SIFT:变异预测工具;Phylop:测量个别校准点的进化保存;MutationTaster:评估DNA序列变体的致病潜力的工具;COSMIC:收集和显示癌症体细胞突变的信息数据库;OMIM:关于人类基因和遗传紊乱的数据库;ClinVar:由国立生物技术信息中心(NCBI)主办和国立卫生研究院(NIH)资助的人类变异与表型关系报告的公共档案;HGMD:人类基因变异数据库;CSV:逗号分隔值,其文件以纯文本形式存储表格数据(数字和文本);TSV:制表符分隔值,是一种用于储存数据的文本格式文件,其数据以表格结构(例如数据库或电子表格数据)储存;TXT:是微软在操作系统上附带的一种文本格式,是最常见的一种文件格式;SNVs:单碱基位点变异;SnpSift:使用数据库、过滤器和操纵基因组注释的变量以注释基因组变量的工具
声明25:体细胞与胚系来源的变异应加以区分
体细胞突变在肿瘤诊疗中的临床意义较大。特定的胚系突变的生物学意义需说明,如乳腺癌中BRCA1/2的相关胚系来源突变。
声明26:应对各个瘤种中具有明确或重要临床意义的基因变异进行关注分析和说明
比如肺癌中的EGFR exons 18-21基因突变、ALK基因重排、ROS基因重排、MET E14基因拼接变异、HER2基因插入突变或扩增等突变的说明。
近年来,关于肿瘤细胞分子异质性的研究进展发现,肿瘤中存在不同分子变异亚型的亚克隆。异质性亚克隆的存在会影响肿瘤疾病的整体进化路径及对靶向治疗的应答和耐药。因此NGS用于肿瘤诊断或耐药监测时需达到较深的测序覆盖度,这样才能充分解释肿瘤转移进化过程中的异质性及其与临床表型的关系。
声明27:CAGC建议,临床肿瘤组织样本的NGS检测数据中测序有效深度应达到500倍以上,测序深度是指测序得到的碱基总量与基因组大小的比值,是评价测序量的指标之一;血浆游离DNA标本的NGS有效测序深度应达到1 000倍以上
说明:(1)有效深度定义为去除PCR重复读取(复制)之后的深度;(2)应在80%以上的目标捕获区域达到这个深度,而非是所有区域的平均,否则在区域间覆盖波动较大的情况下会有大量区域出现覆盖不够的情况。
声明28:CAGC项目数据的管理应在标准流程和各类数据管理方面实现同一化
在后续将开展每个瘤种约100例的分析,从而对本次共识的可操作性进行验证。
NGS是多步骤的复杂的检测和分析方法,产生报告的内容较多。对临床而言需遵循首页简明、结果明确、解释清楚、信息充分的原则。
在开展NGS临床测试之前,应明确定义所检测的驱动基因在基因组上的哪些靶区域范围,并在报告中告知临床医师。
NGS报告结果应简明清晰。具有临床靶向意义的检测结果及其参数应优先报告,如EGFR L858R突变,突变位点占比(MAF)=15.6%。在报告首页中应包括患者身份标识号、临床诊断、测试的简单描述、技术类型等信息。根据情况,报告可以适当添加附件成多页。各个瘤种的核心基因检测结果必须明确报告,其他基因根据结果概括性报告,对于特别的突变(如在其他肿瘤中发现是核心变异)可特别进行报告。
对于生物学未明确分类的变异结果应收集整理,并提倡在同行间交换,为最终确认这些未明确分类的变异是功能性或非功能性的提供帮助。
对于不同瘤种的NGS数据,可以整合到同一个数据库进行分析,这将有助于不同瘤种间变异信息在临床应用中的借鉴。
声明29:报告格式和内容需标准化,应能适应短期未来的发展趋势(靶点知识和检测内容的变更)
报告内容应包括患者身份标识号、疾病信息、样本信息、分析技术信息、主要结果、结果解释、操作员和实验室专家(肿瘤分子生物学或遗传学专家)签字等。
声明30:报告范围应根据测试目的及测序能够达到质量要求的覆盖范围来决定
例如在对外显子组序列的检测中,在肺癌中不一定报告所有覆盖区域中检出的变异。但需知情临床医师本次报告不能排除潜在的非关注区域的变异。
声明31:NGS报告应简明扼要、明确易懂
能充分提供必要信息以供医师参考用于当前临床的靶向治疗方案决策。
声明32:结果解释需客观中肯,平实地描述肿瘤突变结果
在疾病相关性方面只描述既往研究中的疗效或预测相关性,不应出现建议使用何种治疗手段或策略的文字。检测结果在临床决策中的应用应由临床医师或多学科专家讨论决定。
声明33:遗传咨询需多学科支持
NGS数据用于临床决策的制定过程中需与实验室、癌症遗传学、分子生物学、生物信息学、病理学等多方面专家进行必要的信息沟通。
声明34:NGS检测项目开展前应向患者提供知情同意或相关遗传咨询
应充分向患者解释疾病条件下进行特定NGS检测分析的目的和利弊。同时需向医师及患者告知测试的局限性,包括驱动基因的信息、报告范围、技术分析的敏感度和特异度等。
知情同意书的内容至少必须包含:(1)进行基因检测的依据,其中包括医学指南、专家共识、治疗规范等内容,需使患者知悉检测的意义和目的;(2)基因检测过程中可能有的局限性,如提取样本量是否足够,样本质量控制指标是否适合基因检测,如发生类似情况,需及时向患者以及家属说明情况,再决定下一步;(3)进行基因测序可辅助个体化治疗,但是最终决定须由临床医师决定;(4)基因检测结果要只针对该次送检样本;(5)检测机构必须对患者的身份信息、病例信息以及检测结果等保密。患者进行基因检测之前需知悉以上事项,并签署知情同意书。
由于技术的快速发展和应用单位的能力提升,NGS用于研究或是诊断的界限已经模糊,但在实际应用中,应明确特定的NGS测试是用于研究还是诊断。诊断目的的NGS检测是为了回答临床患者肿瘤中是否具有特定的基因变异状态(可作用靶点的变异)。诊断检测通常是在一个得到认证的实验室开展。而研究目的的NGS检测是研究假设驱动的检测,检测结果对入组患者通常只具有有限或不确定的临床意义。全外显子或基因组测序的诊断结果由于数据量大,除了满足特定的基因诊断,其他数据也可能产生新的研究假设。
声明35:研究项目获得有临床意义的分子变异结果需进一步在诊断条件下确证后才可纳入患者的医疗记录
通常研究活动中发现的某种与临床治疗决策相关的靶点变异,需进一步采用已有的规范化或标准化的测试分析验证后,才可以用于临床实践和纳入医疗记录。
声明36:研究和诊断的检测结果都应建立数据库管理
NGS数据是当代疾病大数据的主要成分之一,数据库建立也便于与本地、区域性的、国家或国际的数据库进行比对分析,这有助于对变异进行临床分类。
对于应用于CAGC-POI临床实践、试验或研究的NGS检测项目,为保证质量的同一性和延续性,将由CSCO CAGC专家组对所测试的NGS项目进行监督管理,管理方式包括现场考察、管理文件和流程监督、测试样本的室间评比等。目的是使得参与CAGC-POI项目的各个NGS实验室能够达到统一的质量标准。
共识制定专家组
组长:广东省人民医院(广东省医学科学院)肿瘤治疗中心(吴一龙)
成员(排名不分先后):解放军南京总医院呼吸内科(宋勇),病理科(周晓军);吉林省肿瘤医院肿瘤内科(程颖),研究中心(李慧);中国医学科学院肿瘤医院病理科(应建明),肿瘤内科(王洁、徐兵河),内科实验室(白桦);四川大学华西医院病理科(刘卫平),肿瘤科(卢铀);复旦大学附属中山医院病理科(侯英勇),肿瘤内科(刘天舒);暨南大学医学院血液病研究室血液科(李杨秋),血液病研究所(田红霞);解放军第三〇七医院肿瘤学研究室(刘毅),病理科(邰艳红),肿瘤科(刘晓晴);华南理工大学第一临床学院(候珺);解放军第八一医院肿瘤内四科(刘秀峰);中山大学附属肿瘤医院病理科(邵建永),结直肠科(陈功),肿瘤内科(徐瑞华);北京医院病理科(王征);天津医科大学肿瘤医院乳腺肿瘤科(付丽);北京协和医院病理科(梁智勇),呼吸内科(王孟昭);广东省人民医院(广东省医学科学院)肿瘤治疗中心肺三科(周清),肿瘤内科(杨衿记),肿瘤外科(钟文昭、杨学宁),乳腺科(廖宁),广东省肺癌研究所(张绪超、陈宇);福建省肿瘤医院胸部肿瘤内科(黄诚);复旦大学附属肿瘤医院病理科(周晓燕),乳腺外科(邵志敏);杭州市第一人民医院转化医学中心(张仕蓉);上海交通大学医学院附属瑞金医院血液研究所(任瑞宝),国家转化医学中心(上海)(王升跃);上海市肺科医院病理科(武春燕);解放军第一一三医院肿瘤科肿瘤科(朱忠政);浙江大学医学院附属邵逸夫医院肿瘤中心(潘宏铭);江苏省人民医院肿瘤内科(顾艳宏);上海交通大学医学院附属仁济医院肿瘤科(王理伟);中国医科大学附属第一医院肿瘤内科(刘云鹏);浙江大学医学院附属第二医院肿瘤外科(张苏展);哈尔滨医科大学附属肿瘤医院肿瘤内科(张清媛);北京大学肿瘤医院消化肿瘤内科(沈琳);上海交通大学附属胸科医院肺部医学肿瘤中心(陆舜)
主要撰稿专家:广东省人民医院(广东省医学科学院)广东省肺癌研究所(张绪超);北京协和医院病理科(梁智勇);上海交通大学瑞金医院国家转化医学中心(王升跃);上海交通大学附属胸科医院肺部医学肿瘤中心(陆舜);广东省人民医院(广东省医学科学院)肿瘤治疗中心(吴一龙)
厦门艾德生物医药科技股份有限公司(朱冠山);广州燃石医学检验所有限公司(揣少坤);北京雅康博生物科技有限公司(徐军武);罗氏诊断产品(上海)有限公司(杜雷);北京基因加科技有限公司(杨玲);广州精科生物技术有限公司(蒋馥蔓);上海思路迪精准医疗集团(熊磊);华大基因生物科技(深圳)有限公司(赵美茹);上海至本生物科技有限公司(王凯);北京诺禾致源生物信息科技有限公司(蒋智)

























