
采用生物信息分析技术探讨胶质瘤(Glioma)差异表达基因筛选、功能富集和相关信号通路。
在基因表达谱数据库(GEO)中选取胶质瘤中相关基因表达谱芯片数据,采用R软件lima包筛选胶质瘤患者肿瘤组织和正常脑组织中差异表达基因。对筛选出的差异表达基因进行功能富集(GO和KEGG),应用采用蛋白-蛋白相互作用数据库(STRING)分析筛选出的差异表达基因编码蛋白间的相互作用关系,分析相关信号通路。
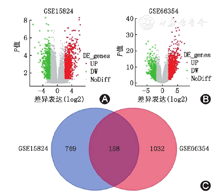
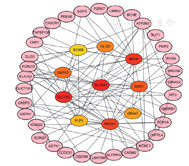
选取GSE15824和GSE66354基因表达谱数据集为分析对象,筛选出差异表达超过2倍,且P<0.05的基因158个。158个差异基因主要分子功能(MF)为整合素结合、细胞黏附分子结合、钙离子结合及AMPA谷氨酸受体活性等;细胞组分定位(CC)于细胞膜、神经元细胞体及神经细胞轴突等,而其生物学过程(BP)主要为细胞黏附、神经系统发育、细胞增殖、GTP酶活性、细胞凋亡和血管生成等;KEGG信号通路主要为cAMP信号通路、嘌呤代谢通路、MAPK信号通路及cGMP-PKG信号通路,158个差异表达基因蛋白相互作用网络中相互作用连接共177个,平均每个节点间相互作用为2.39个,聚集系数为0.37。Cytohubb筛选信号通路中的关键基因(hub基因),结果提示,SLC6A1、SLC1A2、BDNF、CAP43、NRXN1、GAD1、OLIG2、PLP1、S100B和GRIA3为相互作用蛋白网络信号通路中的关键基因。10个关键基因均与患者预后有关(P<0.05)。
胶质瘤患者肿瘤组织和正常组织存在差异表达基因谱,SLC6A1、SLC1A2、BDNF、GAP43、NRXN1、GAD1、OLIG2、PLP1、S100B和GRIA3为胶质瘤发生的关键基因并与患者的预后有关。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
胶质瘤在普通人群年发病率为3~8例/10万[1,2]。2016年世界卫生组织(WHO)提出了分子和病理学的5种胶质瘤分型[3,4]。多数研究认为,胶质瘤是遗传因素为背景,后天环境因素为诱发因素的疾病[4,5,6]。相关遗传因素包括神经纤维瘤病(Ⅰ型)以及结核性硬化疾病等为胶质瘤的遗传因素。有研究显示,手机等电离辐射与脑胶质瘤的发生无相关性[7],胶质瘤更倾向于为一种多基因,多步骤的遗传相关的疾病[6]。近年来随着基因表达谱芯片及二代高通量测序技术的进展,联合分析胶质瘤差异表达基因及相关信号通路成为可能。在本研究筛选了胶质瘤相关基因表达谱芯片数据集为研究对象,探讨差异表达基因、相关功能、信号通路及其作为胶质瘤患者预后分子标志物的可行性。
选取基因表达谱数据库(GEO)(https://www.ncbi.nlm.nih.gov/geo/)中收录的关于胶质瘤患者肿瘤组织与正常脑组织差异表达的芯片数据为研究对象。生物学信息注释数据库DAVID(https://david.ncifcrf.gov/),用于GEO数据库中筛选出的差异表达基因进行功能注释与富集。其中功能组注释和富集主要包括:基因本体论(GO)和KEGG信号通路分析。GO分析包括3个层次,分别为生物学过程(BP)、细胞组分(CC)和主要分子功能(MF)。选择蛋白-蛋白相互作用网络构建数据库STRING(http://string-db.org/cgi/input.pl),对筛选出的差异表达基因编码蛋白进行相互作用网络构建,采用cytoscape中cytohubba筛选网络中的关键基因。采用TCGA数据库中胶质瘤患者给予表达水平与预后关系数据,绘制生存曲线。
首先在GEO数据库中检索胶质瘤患者相关基因表达谱芯片数据集,检索词为"Glioma",物种为"human"。对检索到的可能相关数据集进行人工复核,最终选取了GSE15824[8]和GSE66354[9]2个基因表达谱数据集为分析对象。采用R软件lima包筛胶质瘤患者肿瘤组织和正常脑组织中差异表达基因进行筛选。筛选条件为,筛选出差异表达超过2倍,且P<0.05。分别对2个数据集中上述筛选条件获得的差异表达基因进行重叠分析并绘制venn图,进一步筛选出差异表达基因。在生物学信息注释数据库DAVID(https://david.ncifcrf.gov/)对筛选出的差异表达基因进行GO和京都基因与基因组百科全书(KEEG)生物功能富集分析,预测其可能功能及信号通路。在STRING数据库中对上述筛选出的差异表达基因编码蛋白进行相互作用网络构建,构建条件为:(1)蛋白相互作用数据来源选择Textmining,Experiment,database,co-expression,gene function和co-occurrence。(2)最小相互作用评分为0.4。将STRING构建的蛋白网路数据导入cytoscape中筛选网路信号通路中的关键基因。采用TCGA数据在线分析软件,将cytoscape中筛选出的关键基因根据表达水平分为高表达组和低表达组,比较高低表达组患者生存期是否存在差异,并绘制生存曲线,计算风险比(HR)。
应用Stata 11.0软件进行统计分析(TX),计量资料应用 ±s表示,应用t检验;计数采用率表示,应用Chi-Square检验,生存分析采用风险比例模型,并绘制Kaplan-Meier曲线进行分析。以P<0.05为差异有统计学意义。
±s表示,应用t检验;计数采用率表示,应用Chi-Square检验,生存分析采用风险比例模型,并绘制Kaplan-Meier曲线进行分析。以P<0.05为差异有统计学意义。
选取了GSE15824和GSE66354基因表达谱数据集为分析对象。GSE15824数据集中共分析了30例胶质瘤标本及5例正常脑组织标本,对比了上述组织标本中基因的表达情况。GSE66354数据集对比了149例胶质瘤患者及对应的正常脑组织中基因的表达情况。筛选出差异表达超过2倍,且P<0.05的基因158个,图1。

对筛选出的158个差异表达基因进行了相关功能分析,结果显示,相关基因MF为整合素结合、细胞黏附分子结合、钙离子结合及AMPA谷氨酸受体活性等;CC于细胞膜、神经元细胞体、神经细胞轴突等,而其BP主要为细胞黏附、神经系统发育、细胞增殖、GTP酶活性、细胞凋亡和血管生成等;KEGG信号通路主要为cAMP信号通路、嘌呤代谢通路、MAPK信号通路及cGMP-PKG信号通路。
在STRING数据库中分析上述158个基因编码蛋白间相互作用关系,结果,蛋白相互作用网络中相互作用连接宫177个,平均每个节点间相互作用为2.39个,聚集系数为0.37。Cytohubb筛选信号通路中的关键基因(hub基因),结果,SLC6A1、SLC1A2、BDNF、GAP43、NRXN1、GAD1、OLIG2、PLP1、S100B和GRIA3为相互作用蛋白网络信号通路中的关键基因,图2。

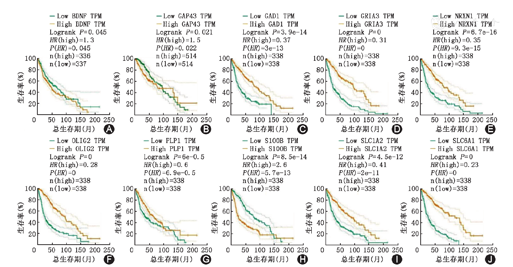
根据hub基因表达水平分为高表达组和低表达组,比较高低表达组患者预后是否存在差异。TCGA数据库生存分析显示,上述10个关键基因均与患者预后有关(P<0.05),图3。

神经胶质瘤占所有脑肿瘤的30%左右,而80%的恶性中枢神经系统肿瘤为胶质瘤[10]。脑胶质瘤中最常见的类型是胶质母细胞瘤,其预后较差,中位数为10~15个月。胶质瘤的发病机制十分复杂,被认为是多基因和蛋白质参其发生发展过程,但目前胶质瘤的确切发病机制仍不十分清楚[11]。既往大多数研究从单个或几个基因DAN,mRNA和转录后修饰、翻译等水平探讨脑胶质瘤的发病机制及其与细胞表型的关系[12,13]。此类研究无法实现高通量,且各个研究间由于实验室条件、研究方法等原因,研究结果并不完全一致,甚至会出现相反的结果。因此,从分子水平进行高通量多基因检测,并探讨其与胶质瘤发生、发展及侵袭转移等生物学表型的关系成为了近年来的研究热点。
近年来随着基因表达谱芯片及二代高通量测序技术的不断进展,联合分析胶质瘤差异表达基因及相关信号通路成为可能。基因表达谱分析是一种广泛应用于临床的有效方法。从分子水平鉴定不同类型癌症中肿瘤相关基因的表达水平是否存在差异,从而为相关肿瘤的诊断、治疗、预后评价提供了可行性方案。
在本研究选取GSE15824和GSE66354基因表达谱数据集为分析对象,筛选出了158个差异表达基因。158个差异表达基因MF为整合素结合、细胞黏附分子结合、钙离子结合及AMPA谷氨酸受体活性等;CC于细胞膜、神经元细胞体及神经细胞轴突等,而其BP主要为细胞黏附、神经系统发育、细胞增殖、GTP酶活性、细胞凋亡和血管生成等;KEGG信号通路主要为cAMP信号通路、嘌呤代谢通路、MAPK信号通路及cGMP-PKG信号通路。本研究进一步筛选信号通路中的关键基因,发现SLC6A1、SLC1A2、BDNF、CAP43、NRXN1、GAD1、OLIG2、PLP1、S100B和GRIA3为相互作用蛋白网络信号通路中的关键基因,上述10个基因与整个网络中其他蛋白相互作用最为紧密,可能在胶质瘤发生发展等信号通路中起到关键作用。
同时,本研究在TCGA数据库中分析了上述10个基因高低表达与患者预后直接的关系。结果显示,上述10个关键基因均与患者预后有关(P<0.05)。其中BDNF、CAP43(NDRG1)及S100B基因高表达患者预后差于低表达患者;而GAD1、GRIA3、NRXN1、OLIG2、PLP1、SLC6A1及SLC1A2高表达患者预后好于低表达者。结果提示,上述基因表达水平可作为患者预后的分子标志物。虽然本研究通过生物信息学分析筛选出了脑胶质瘤信号通路中的10个关键基因,且这10个基因与患者的预后存在相关性。但上述研究结果缺乏实验室分子生物学实验的进一步证实,是研究的局限性所在。因此,下一步应重点对上述10个基因在脑胶质瘤中的分子生物学功能进行验证,为脑胶质瘤发病机制及治疗靶点提供更为充分的依据。
所有作者均声明不存在利益冲突



























