
采用生物信息学方法探索与非酒精性脂肪肝炎(NASH)病理进程相关的核心基因及分子机制。
由基因表达数据库(GEO)下载基因表达数据集GSE89632,包含单纯非酒精性脂肪肝患者、NASH患者和健康对照分别为20、19和24例,筛选单纯非酒精性脂肪肝患者和NASH患者相对于健康对照的差异表达基因(DEGs),对两组DEGs取交集。采用DAVID 6.8数据库对DEGs进行GO功能富集分析,采用KOBAS 3.0数据库对DEGs进行京都基因与基因组百科全书(KEGG)信号通路分析。利用STRING数据库构建DEGs蛋白相互作用网络(PPI),采用Cytoscape软件筛选核心基因。利用Attie Lab 糖尿病数据库验证核心基因在4组C57BL/6小鼠(分别为4周龄正常组、4周龄肥胖组、10周龄正常组和10周龄肥胖组,每组各5只)肝脏中mRNA相对表达量。分析核心基因和预后临床指标的相关性。
由GSE89632数据集筛选出单纯非酒精性脂肪肝患者和NASH患者相对于健康对照的365个共同DEGs,其中上调和下调基因分别为115和250个。GO分析显示DEGs主要富集于炎症反应和免疫应答等生物过程。KEGG信号通路分析显示:上调基因主要富集于胆固醇代谢、胆汁分泌和脂肪的消化吸收等信号通路;下调基因主要富集于白细胞介素-17信号通路、肿瘤坏死因子信号通路和糖尿病并发症的晚期糖基化终末产物及其受体等信号通路。PPI分析筛选出7个关键核心基因,分别为FOS、EGR1、FOSB、JUNB、FOSL1、MYC和NR4A1。10周龄肥胖小鼠肝脏中EGR1和JUNB的mRNA相对表达量均低于10周龄正常小鼠,均P<0.05;4和10周龄肥胖小鼠肝脏中NR4A1相对表达量均低于同周龄正常组小鼠,均P<0.05。EGR1基因表达水平与肝脏脂肪变性程度呈负相关(r=-0.785,P<0.001)。FOSB、MYC和NR4A1基因表达水平与血液谷丙转氨酶水平呈负相关(r=-0.649、-0.597和-0.580,均P<0.001)。
EGR1、FOSB、MYC、JUNB和NR4A1等基因可能为NASH病理进程中的核心基因,肝细胞内炎症反应和免疫应答、白细胞介素-17信号通路和肿瘤坏死因子信号通路可能是NASH病理进程的关键分子机制。

非酒精性脂肪肝病(NAFLD)是指除外过量饮酒和其他明确的损肝因素所致的以肝细胞内脂肪过度沉积为主要病变特征的临床病理综合征[1],2013年中国大陆人群患病率为20.09%,2015年全球患病率为25.24%,是世界范围内最常见的慢性肝脏疾病[2, 3]。NAFLD包括单纯非酒精性脂肪肝和非酒精性脂肪肝炎(NASH),预期NASH患者占比将由2015年的20%升高至2030年的27%[4, 5]。NASH患者中约有20%进展为晚期肝纤维化,最终导致终末期肝癌、肝细胞癌(HCC)、肝移植和死亡[5, 6, 7]。目前尚无预测NAFLD病理进程的适宜方法[8, 9],本研究基于美国国家生物技术信息中心的基因表达数据库(GEO)(https://www.ncbi.nlm.nih.gov/geo/)探索NASH发病的核心基因及分子机制,为该病的早期诊断和临床治疗提供依据。
筛选NAFLD差异表达基因(DEGs)所需资料源于美国国家生物技术信息中心GEO芯片平台(GPL14951)的基因表达谱芯片(GSE89632)数据集,该数据来自63例人源肝脏活检组织样本,其中单纯非酒精性脂肪肝、NASH和健康对照分别为20、19和24例。核心基因表达验证所需资料源于Attie Lab 糖尿病数据库(http://diabetes.wisc.edu)。
1.DEGs筛选:采用GEO2R在线分析程序(http://www.ncbi.nlm.nih.gov/geo/geo2r/)从GSE89632数据集分别筛选出单纯非酒精性脂肪肝患者和NASH患者相对于健康对照的DEGs(基因上调或下调差异倍数>1且调整后P值<0.05),对两组DEGs取交集。
2.基因本体(GO)和京都基因与基因组百科全书(KEGG)富集分析:利用DAVID 6.8数据库(https://david.ncifcrf.gov/),以人源基因为背景,对筛选出的DEGs进行GO功能注释。采用KOBAS 3.0数据库(http://kobas.cbi.pku.edu.cn/kobas3)对DEGs进行KEGG信号通路富集分析。
3.蛋白相互作用网络(PPI)构建和核心基因筛选:采用STRING 11.0数据库(https://string-db.org/)对DEGs进行PPI网络构建,导入Cytoscape软件后进行可视化分析。采用分子复合物检测分析插件(MCODE)筛选出2个关键聚类功能模块并计算出各模块的5个关键基因。采用Cytohubba插件的最大集团中心性(MCC)算法,依照基因在PPI网络的重要性筛选评分前10位的基因,与上述关键聚类功能模块中获得的10个关键基因取交集,获得核心基因。
4.核心基因的表达验证:从Attie Lab糖尿病数据库获取4组C57BL/6小鼠(分别为4周龄正常组、4周龄肥胖组、10周龄正常组和10周龄肥胖组,每组各5只)肝脏中核心基因mRNA相对表达量[10]。
5.核心基因表达与预后临床指标相关性分析:预后临床指标包括年龄、体质指数(BMI)、腰围、肝脏脂肪变性程度、高密度脂蛋白胆固醇、低密度脂蛋白胆固醇、总胆固醇、甘油三酯、谷草转氨酶和谷丙转氨酶水平等10项。以原始芯片数据集(GSE89632)中上述指标完整的41名对象,分析核心基因与预后临床指标的相关性。
采用GraphPad Prism 8分别绘制单纯非酒精性脂肪肝患者和NASH患者相对于健康对照的DEGs火山图。对DEGs进行GO及KEGG分析,采用独立样本t检验分别比较4周龄正常组和肥胖组小鼠、10周龄正常组和肥胖组小鼠肝脏中核心基因mRNA相对表达水平的差异,并采用GraphPad Prism 8绘制散点图,采用Spearman相关分析,分析核心基因和预后临床指标的相关性,绘制热图。双侧检验,以P<0.05为差异有统计学意义。
1. NAFLD患者的人口学特征及DEGs情况:单纯非酒精性脂肪肝患者年龄为(44.35±9.65)岁,男性占70.0%(14/20);NASH患者年龄为(43.47±12.73)岁,男性占47.3%(9/19);健康对照年龄为(30.59±21.08)岁,男性占45.8%(11/24)。由GSE89632数据集分别得到单纯非酒精性脂肪肝患者和NASH患者相对于健康对照的DEGs火山图(图1),其中共同DEGs365个,包括115个上调基因和250个下调基因。
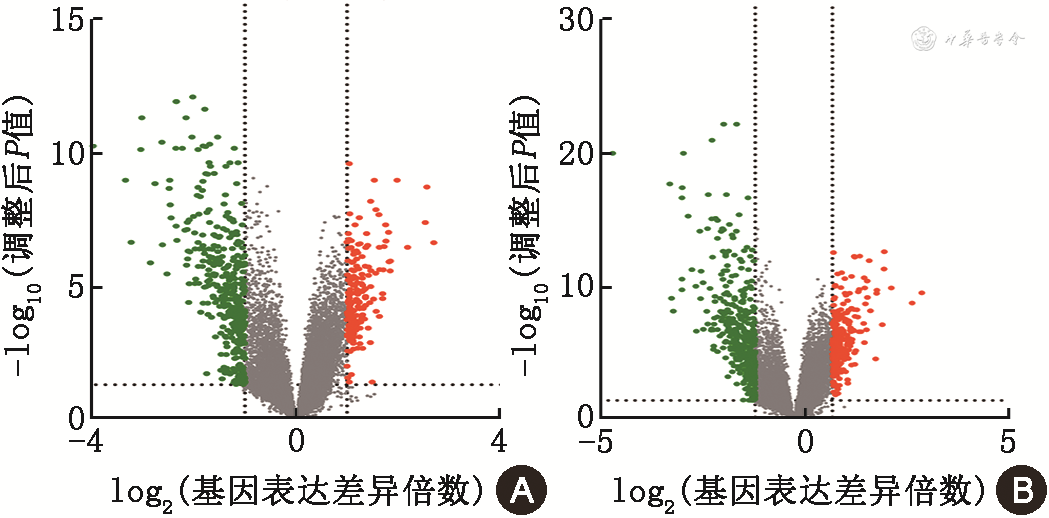

注:红点为上调基因;绿点为下调基因
2.DEGs的GO和KEGG分析结果:GO分析显示,上调基因主要富集于转运、跨膜转运和烟酰胺腺嘌呤二核苷酸磷酸代谢过程等生物过程,细胞组成与顶端质膜有关;下调基因主要富集于炎症反应、RNA聚合酶Ⅱ启动子转录的正调控和免疫应答等生物过程,细胞组成与细胞外间隙有关(表1)。KEGG通路分析显示,上调基因主要富集于胆固醇代谢、胆汁分泌和脂肪的消化吸收等信号通路;下调基因主要富集于白细胞介素-17信号通路、肿瘤坏死因子信号通路和糖尿病并发症的晚期糖基化终末产物及其受体等信号通路。

非酒精性脂肪肝病患者差异表达基因的基因本体(GO)功能富集分析
非酒精性脂肪肝病患者差异表达基因的基因本体(GO)功能富集分析
| GO序号 | GO分类 | GO注释 | 调节类型 | 富集基因数 | P值 |
|---|---|---|---|---|---|
| GO:0006810 | 生物过程 | 转运 | 上调 | 8 | 0.001 |
| GO:0016324 | 细胞组成 | 顶端质膜 | 上调 | 6 | 0.012 |
| GO:0055085 | 生物过程 | 跨膜转运 | 上调 | 5 | 0.025 |
| GO:0006739 | 生物过程 | 烟酰胺腺嘌呤二核苷酸磷酸代谢过程 | 上调 | 2 | 0.027 |
| GO:0031849 | 分子功能 | 嗅觉受体结合 | 上调 | 2 | 0.028 |
| GO:0006954 | 生物过程 | 炎症反应 | 下调 | 29 | <0.001 |
| GO:0045944 | 生物过程 | RNA聚合酶Ⅱ启动子转录的正调控 | 下调 | 40 | <0.001 |
| GO:0005615 | 细胞组成 | 细胞外间隙 | 下调 | 42 | <0.001 |
| GO:0006955 | 生物过程 | 免疫应答 | 下调 | 22 | <0.001 |
| GO:0001077 | 分子功能 | 转录激活因子活性,RNA聚合酶Ⅱ核心启动子近端区序列特异性结合 | 下调 | 16 | <0.001 |
3.DEGs的PPI构建和核心基因筛选:两个关键聚类功能模块共得到10个关键基因;采用Cytohubba插件筛选出365个共同DEGs蛋白互作网络的10个关键基因,并与上述10个关键基因取交集,得到7个核心基因,分别为FOS、EGR1、FOSB、JUNB、FOSL1、MYC和NR4A1(图2)。
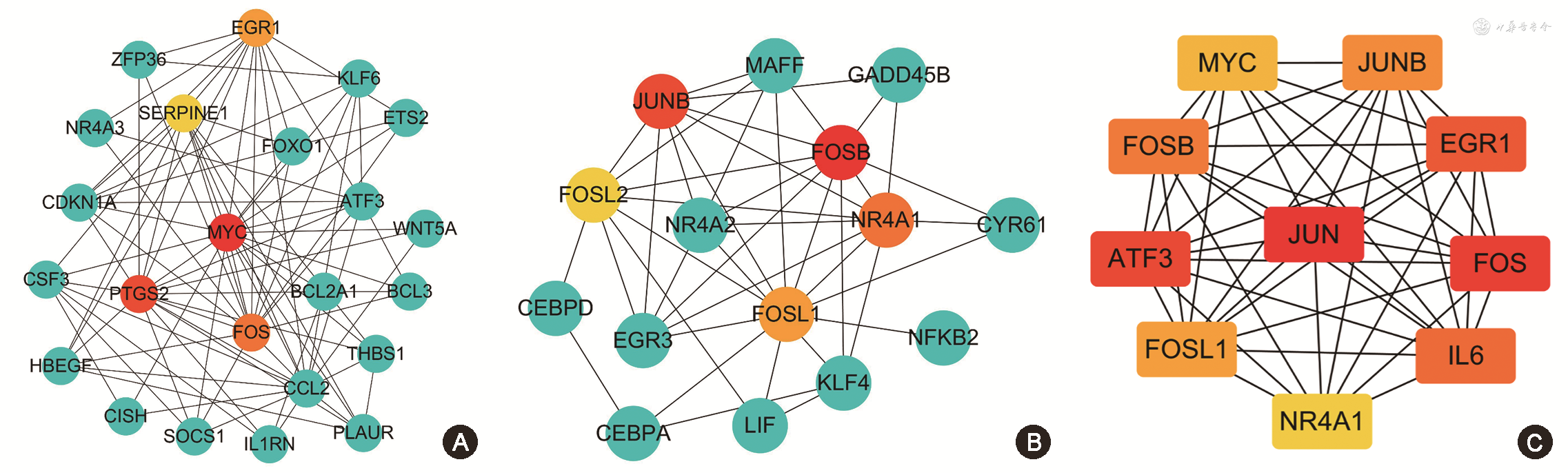

注:节点颜色按照MCC算法评分由高(红色)至低(黄色)
4.核心基因表达验证:对Attie Lab糖尿病数据库小鼠实验数据的分析显示:10周龄肥胖小鼠肝脏EGR1相对表达量低于10周龄正常小鼠(t=3.42,P=0.001);4周龄正常和肥胖小鼠肝脏中EGR1的相对表达量差异无统计学意义(t=2.20,P=0.059)。JUNB在10周龄肥胖小鼠肝脏中的相对表达量低于10周龄正常小鼠(t=3.76,P=0.001);4周龄正常和肥胖小鼠肝脏中JUNB的相对表达量差异无统计学意义(t=1.73,P=0.121)。4和10周龄肥胖小鼠肝脏中NR4A1相对表达量均低于同周龄正常组小鼠(t=2.57、3.59,P=0.033、0.007)(图3)。
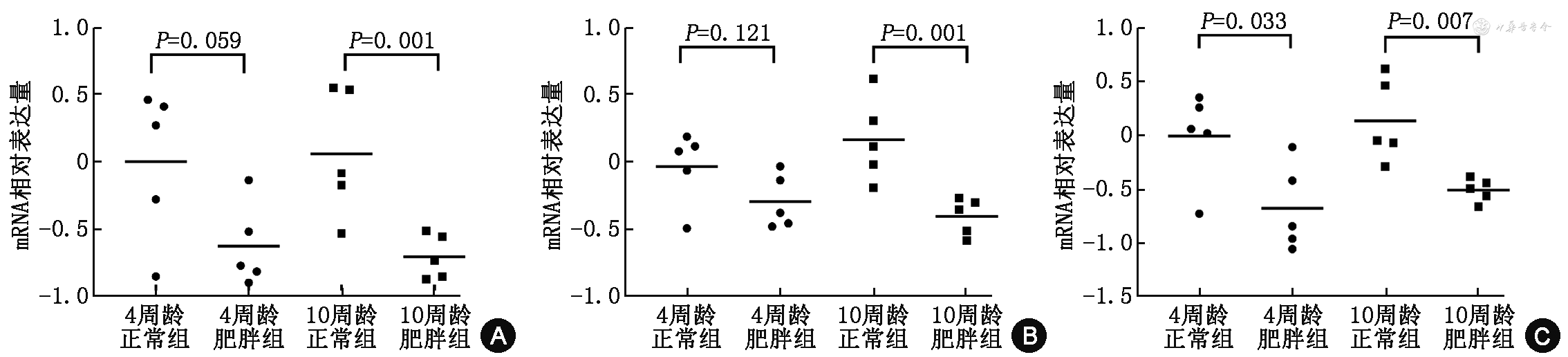

注:mRNA相对表达量=log10(某小鼠特定基因的相对表达量/全部小鼠特定基因参照池表达水平)
5.核心基因表达与预后临床指标的相关性:EGR1基因表达与肝脏脂肪变性程度呈负相关(r=-0.785,P<0.001)。FOSB、MYC和NR4A1基因表达与血液谷丙转氨酶水平呈负相关(r分别为-0.649、-0.597和-0.580,P值均<0.001)(图4)。
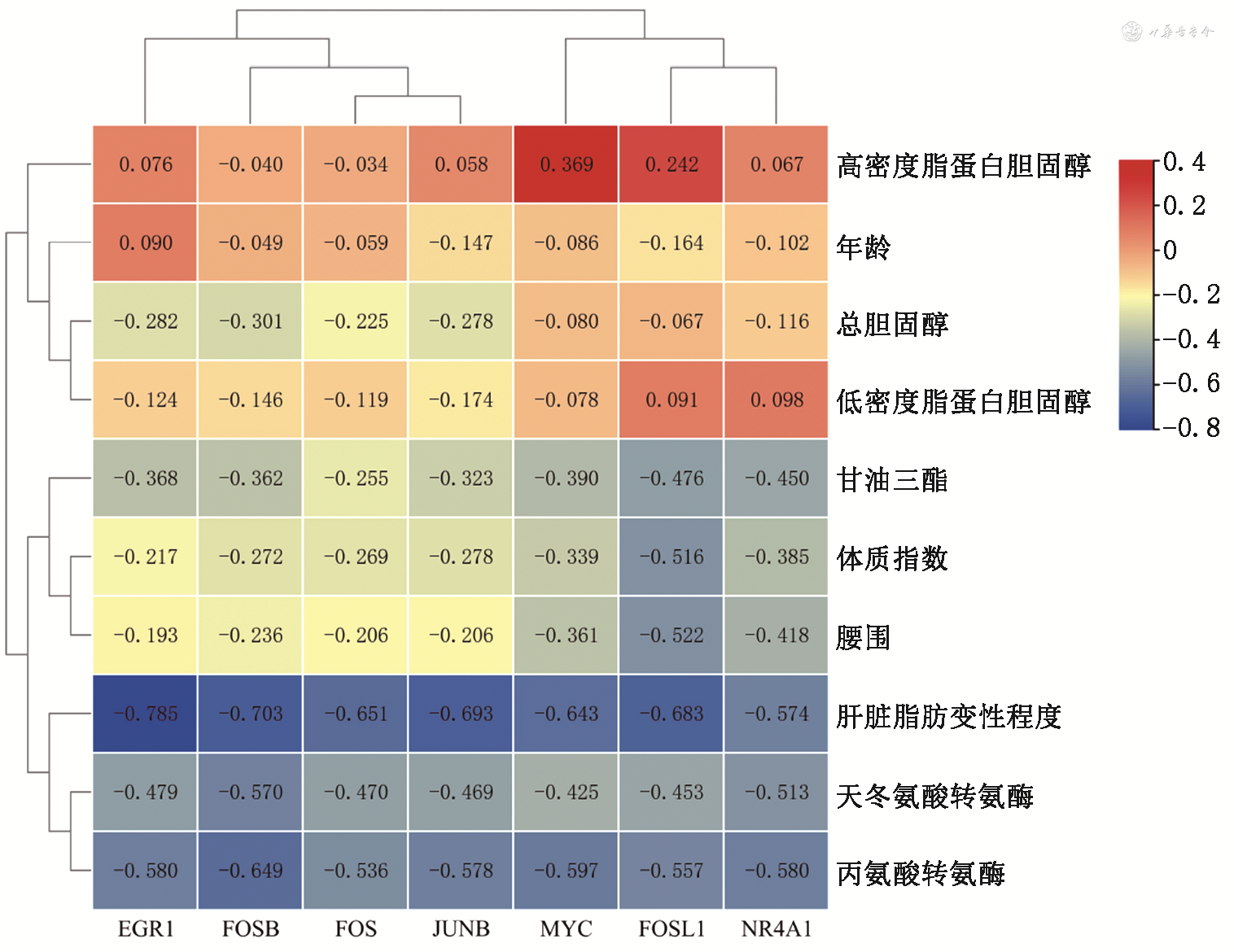

注:图中数值为Spearman相关系数;研究对象包括单纯非酒精性脂肪肝患者(n=16)、非酒精性脂肪肝炎患者(n=12)和健康对照(n=13)
NASH发病与肥胖、高脂血症和2型糖尿病等疾病密切相关,约9%~20%的NASH患者于发病15年内进展为肝硬化甚至肝癌[11]。超声和CT作为诊断NAFLD的主要临床方法,其灵敏度随病理进展和患者肥胖程度的增加明显下降,如:对于BMI<25 kg/m2和BMI>30 kg/m2的患者,超声诊断晚期肝纤维化的灵敏度分别为92.3%和45.5%[12]。因而,探究NASH发生发展过程中的核心基因及分子机制对于建立NAFLD早期筛查和诊断方法具有重要的临床意义。
本研究从GSE89632数据集筛选出与NASH疾病进展相关的7个核心基因,分别为FOS、EGR1、FOSB、JUNB、FOSL1、MYC和NR4A1。GO富集分析显示其功能主要富集于炎症反应、免疫应答等生物过程。静态肝星状细胞激活及其向肌成纤维细胞的分化是由单纯非酒精性脂肪肝向NASH进展的关键事件[13],该分化过程主要依赖于肝脏免疫细胞(主要是库普弗细胞)的炎症活性[14]。肝细胞中大量脂质的持续聚集导致肝细胞损伤和凋亡,肝星状细胞和库普弗细胞吞噬肝细胞释放的凋亡小体,导致纤维化因子分泌或炎性递质释放[15, 16, 17]。库普弗细胞分泌的细胞因子和趋化因子促进其他免疫细胞招募,进一步维持肝脏脂肪变性诱发的炎症反应和肝星状细胞的激活状态[18]。
核心基因NR4A1在细胞炎症反应和糖脂代谢中起重要作用,可调节脂肪形成相关基因的表达,进而抑制脂肪细胞的分化和脂肪生成[19, 20]。对于高脂饲料喂养的小鼠,敲除NR4A1基因可加重胰岛素抵抗和肝组织脂肪变性,而NR4A1基因过表达则降低肝脏甘油三酯聚积[21, 22]。EGR1可识别富含GC核苷酸序列的锌指结构域,在炎症和组织修复过程中起重要作用[23, 24],其在胰岛素抵抗个体的肝脏表达水平明显低于无胰岛素抵抗者。EGR1在FOS基因家族(包括FOS,FOSB,FOSL1和FOSL2)转录起始点附近有结合信号,提示EGR1可能通过调控FOS基因家族转录对胰岛素抵抗和NASH的病理进展发挥关键作用[25]。FOS基因家族和JUN基因家族可与活化蛋白-1结合形成蛋白质二聚体,以亮氨酸拉链形式参与肝脏急性应激反应和脂肪代谢活动[26]。本研究KEGG富集分析显示:核心基因FOS、FOSB、FOSL1和JUN富集于白细胞介素-17信号通路,FOS和JUN富集于肿瘤坏死因子信号通路。本研究还发现EGR1基因表达水平与肝脏脂肪变性程度负相关,FOSB、MYC和NR4A1基因表达与血液谷丙转氨酶水平呈负相关,可结合其他非酒精性脂肪肝定量诊断指标[27],作为预测NASH严重程度的分子标志物。
本研究的局限性在于缺乏分子生物学实验证实,需在NASH细胞及动物模型中对上述7个核心基因进行深入生物功能验证,为NASH的早期筛查和临床诊断提供可靠依据。
所有作者均声明不存在利益冲突



























