
探讨奥沙利铂诱导化疗引起的周围神经病理性疼痛(CIPNP)的分子机制。
SPF级SD雄性大鼠16只采用随机数字表法分为两组:奥沙利铂实验组(5.0%葡萄糖溶液中溶解2.4 mg/kg奥沙利铂,n=8)和对照组(等体积5%的葡萄糖溶液,n=8)。通过奥沙利铂连续给药建立大鼠CIPNP模型,测定并比较两组大鼠机械性痛觉、热痛觉过敏、冷痛觉过敏等疼痛行为学指标。采用RNA测序技术对大鼠背根神经节(DRG)基因转录组水平进行定量,分析奥沙利铂诱导CIPNP的分子机制。
实验组大鼠在第7天开始出现机械痛觉异常和冷热痛觉过敏,在第14天达到最强。通过转录组测序,共定量到20 152个基因,以组间差异倍数绝对值≥2和P<0.05的标准筛选到379个差异表达基因(DEG);其中Npy、Car3、Cdkn1a、Nts、Prc1、Ms4a7和Ecel1 7个基因为外周神经损伤疼痛相关基因。通过基因本体论功能分析,奥沙利铂诱导的差异表达基因主要参与氧运输、细胞分裂、中间体、着丝粒、氧转运蛋白活性、氧结合等功能。通过京都基因与基因组百科全书信号通路(KEGG)分析,奥沙利铂诱导的差异表达基因主要参与疟疾、非洲锥虫病、原发性免疫缺陷、过氧化物酶体增殖物激活受体信号通路(PPAR)等。
奥沙利铂可能通过诱导疼痛相关基因以及相关信号通路引起外周神经性疼痛。

化疗引起的周围神经病理性疼痛(CIPNP)是一种临床上常见且难以治疗的化疗相关并发症,是由化疗药物或其代谢产物对周围神经功能造成的损伤及产生的一系列神经功能紊乱症状和体征[1]。CIPNP可引起患者感觉、运动和自主神经系统功能障碍,常伴有神经病理性疼痛,对患者的治疗效果和生活质量造成不良影响,严重者使患者减轻用药量甚至不得不终止化疗。铂衍生物奥沙利铂(Oxaliplatin)是广泛用于结直肠癌的一线治疗药物[2]。奥沙利铂可引起两种类型的人类神经病性症状:以急性触发性肢端感觉异常为特征的早期急性症状,以及累积剂量的奥沙利铂形成的慢性远端感觉神经病[3]。多达40%的接受铂类药物治疗的癌症患者会出现疼痛和感觉改变[4]。接受奥沙利铂治疗后,奥沙利铂将在背根神经节(DRG)的外周感觉神经元中积累[5],表明DRG中奥沙利铂的积聚是奥沙利铂诱导CIPNP发生的关键触发因素[6]。脊髓星形胶质细胞和小胶质细胞也参与了奥沙利铂诱导的CIPNP的启动和维持[7]。奥沙利铂诱导的大鼠机械性超敏反应与星形胶质细胞的过度活化以及促炎性和神经兴奋性细胞因子的产生增加有关[8, 9]。近期的研究表明,星形胶质细胞的有机阳离子转运体2(Organic cation transporter 2,OCT2)在奥沙利铂诱导的神经毒性中具有重要作用[6]。目前,对奥沙利铂引起的的周围神经性疼痛的分子机制尚不清楚。
随着RNA测序(RNA-Seq)技术的成熟和成本的降低,RNA-Seq已经成为转录组学研究的主要手段[10, 11]。目前,尚无关于奥沙利铂与周围神经性疼痛在转录水平上的研究。本研究旨在通过RNA-seq技术,高通量分析奥沙利铂诱导外周病理性痛大鼠模型DRG组织的基因转录组。
1. 动物:无特定病原体级(SPF)SD大鼠16只,体质量200~250 g,雄性,购自广东省医学实验动物中心,合格证号码SCXK(粤)2013-0002。饲养条件:自由摄食和饮水,室温保持在20~25 ℃,湿度为45%,光照与黑暗时间均为12 h,适应7 d后用于实验。
2. 奥沙利铂致神经痛实验设计:将16只大鼠采用随机数字表法分为两组:奥沙利铂实验组(5.0%葡萄糖溶液中溶解2.4 mg/kg奥沙利铂,n=8)和对照组(等体积的5%的葡萄糖溶液,n=8),连续5 d腹腔注射,诱导CIPNP。在第1次注射奥沙利铂前、注射后第7天和第14天测定行为学,第15天处死大鼠并收集DRG组织(图1A)[12]。
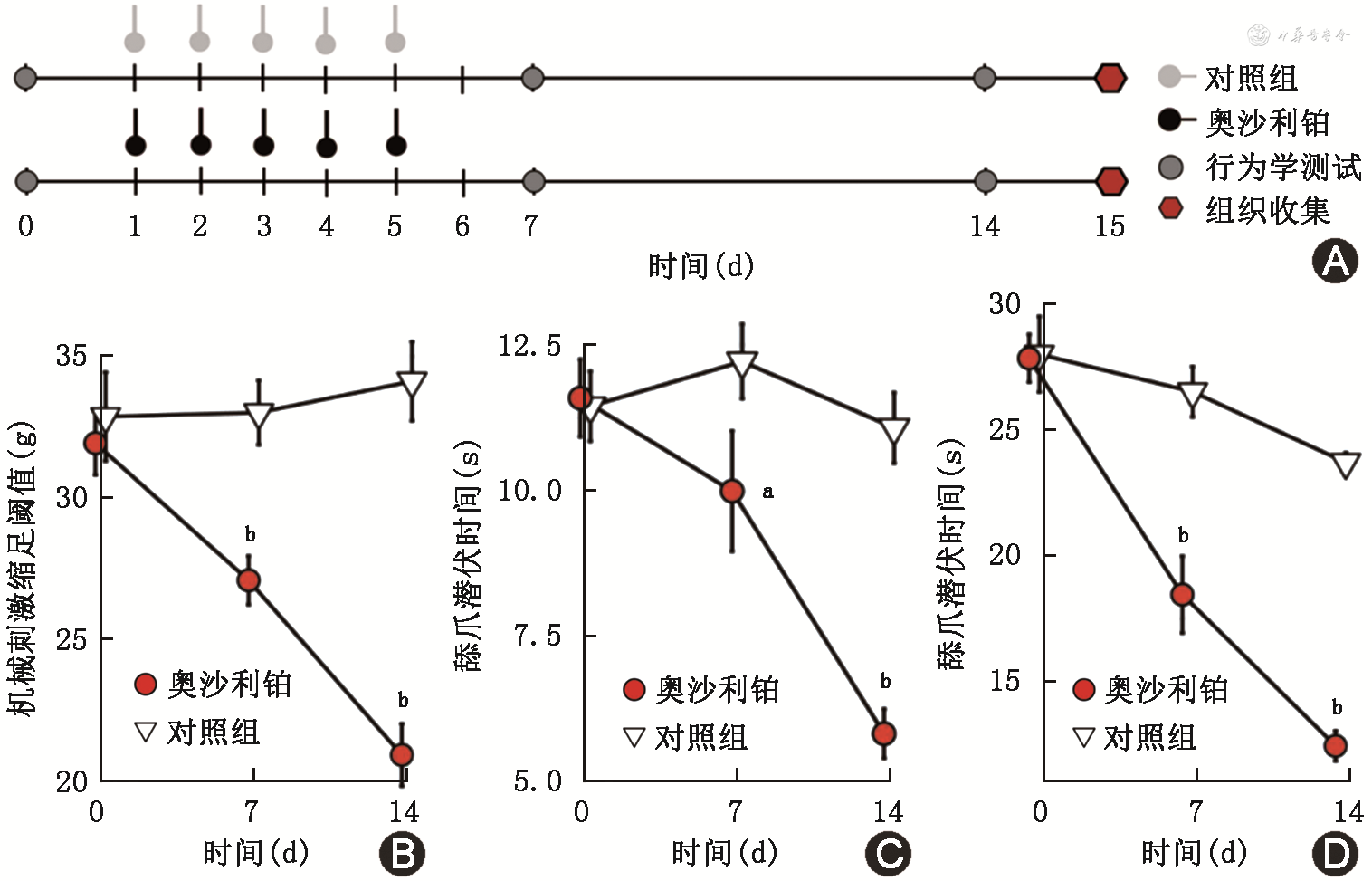

注:与对照组在相同时间点比较,aP<0.05,bP<0.01
3. 机械性痛觉测定:先将动物置于底部为铁丝网的透明箱,适应30 min后开始测定,用Von Frey针垂直顶至大鼠后足爪心固定位置直至大鼠发生撤足反应,记录撤足时测痛仪所显示最大力度,时隔5 min测定1次,共3次,取平均值,记录数据(刺激后出现明显缩足、舔足、抬足均视为阳性反应)。
4. 热痛觉过敏(热板试验)测定:定热板温度为(52.0±0.5)℃。将大鼠放在热板上,大鼠从放在热板上开始计时,到大鼠出现舔后足或者跳跃停止计时,记录大鼠的痛阈值(s)。凡舔足时间<5 s或>30 s或跳跃严重者弃之不用。将合格大鼠重复测3次正常痛阈值,每次间隔10 min,取平均值作为该大鼠给药前的痛阈值。
5. 冷痛觉(丙酮滴试验)测定:大鼠被单独放置在聚乙烯笼中的金属网地板上。在注射器的平头针上形成的一滴丙酮被轻轻地涂抹在大鼠后爪的足底表面。在40 s的时间内记录退出/舔爪反应的时间,每只大鼠测量3次,计算平均值。
6. RNA提取:第15天,选取3项痛阈值(机械性痛觉、热痛觉、冷痛觉)均出现显著的变化大鼠,处死并收集腰椎(L)4、5、6 DRG混合为1份样品,并用Trizol试剂溶解,每组4个生物重复。用氯仿萃取和异丙醇沉淀法提取RNA,储存于-80 ℃深冰箱中,直至使用。
7. 转录组测序分析:测序cDNA文库的制备和RNA测序均由华大基因使用Illumina HiSeq.3000的高通量测序平台完成,平均每份样本获得6 G读段(reads)。原始数据经过去除含有低质量、高含量未知碱基(N)的序列读取过滤之后,使用HISAT4/Bowtie25比对到大鼠参考基因组。使用RSEM6软件包对基因表达水平进行定量。基于基因表达水平,使用NOIseq7和PossionDis8算法来识别样本之间的差异表达基因。使用R语言clusterProfiler包对奥沙利铂组和对照组对比的差异表达基因进行基因本体论(GO)功能分析和京都基因与基因组百科全书信号通路(KEGG)富集分析。
8. 转录组测序原始数据:原始数据已提交至基因表达综合数据库(GEO),ID号为SE16054(https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE160543)。
9. 实时荧光定量PCR(qPCR):qPCR以β-actin作为内参基因,SYBR Green I染料作为荧光检测信号,在96孔反应板中进行实验,每份样品设置4个重复反应孔。引物序列见表1。

实时荧光定量PCR引物序列
实时荧光定量PCR引物序列
| 基因名称 | 上游引物(5′-3′) | 下游引物(5′-3′) |
|---|---|---|
| Npy | TGCTCGTGTGTTTGGGCATT | GATGTAGTGTCGCAGAGCGG |
| Ecel1 | CCTGGAGGAGCTGGACTGGATG | TGGATGCTGAATCGGATGCTGTTC |
| H3f3c | CCACTGAACTTCTGATTCGCAAGC | GCACTCTGGAAGCGCAGGTC |
| Alkal2 | CGCTGCTGAGGTTGCTAGTTGAG | TCGGACTCGCCAAGGAGCTG |
10. 统计学分析:实验数据分析与图可视化采用R语言(3.4.5)统计分析软件。正态分布计量资料采用表示,采用重复测量单因素方差分析及LSD检验;双侧检验,检验水准α=0.05。
1. 奥沙利铂诱导外周病理性痛大鼠模型的建立:实验设计如图1A所示,奥沙利铂连续5 d腹腔注射给药,给药前、给药后第7天和第14天测定行为学阈值[12]。与对照组相比,实验组在给药第7天和第14天的von-Frey试验中出现阈值降低(P<0.001,图1B);热板试验(P<0.05,图1C)以及冷丙酮试验中的舔爪潜伏时间缩短(P<0.01,图1D),表明奥沙利铂诱导外周病理性痛大鼠模型建立成功。
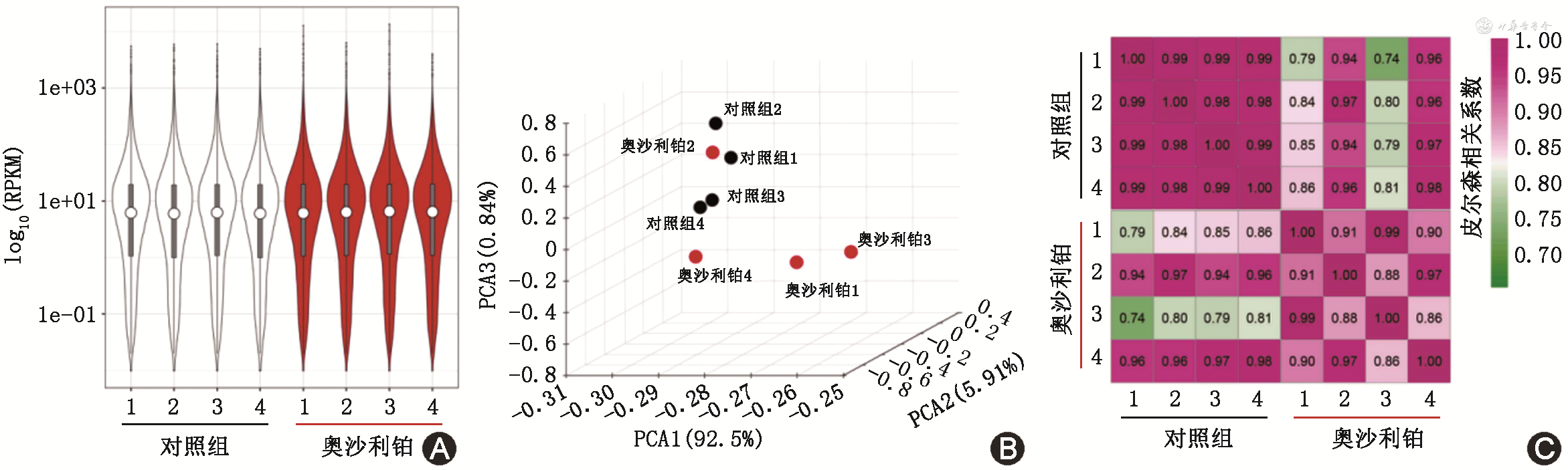
2. 转录组测序对奥沙利铂诱导外周病理性痛大鼠模型的基因表达定量:通过转录组测序共定量20 152个基因,各组基因表达水平的分布情况如图2A所示,各样品基因表达水平均一致(图2B),通过主成分分析(PCA)并结合Pearson检验的Pearson相关系数(图2C),表明对照组和实验组的各重复样品之间相关性较高,转录组测序结果可靠。

3. 奥沙利铂诱导外周病理性痛大鼠模型的差异表达基因分析:以组间差异倍数绝对值≥2、P<0.05的标准筛选奥沙利铂诱导的差异表达基因(图3A),共筛选到379个差异表达基因(其中304个为上调表达基因,75个表达下调基因),前60个显著性差异表达基因如图3B所示。为了研究奥沙利铂的致痛分子机制,分析已经公开发表的选择性外周神经损伤疼痛(SNI)小鼠模型DRG组织基因芯片表达数据(GEO ID为:GSE89224),并将该模型的差异基因与奥沙利铂诱导的差异表达基因进行比较(图3C),发现Npy、Car3、Cdkn1a、Nts、Prc1、Ms4a7


和Ecel1 共7个可被奥沙利铂诱导表达的疼痛相关基因。
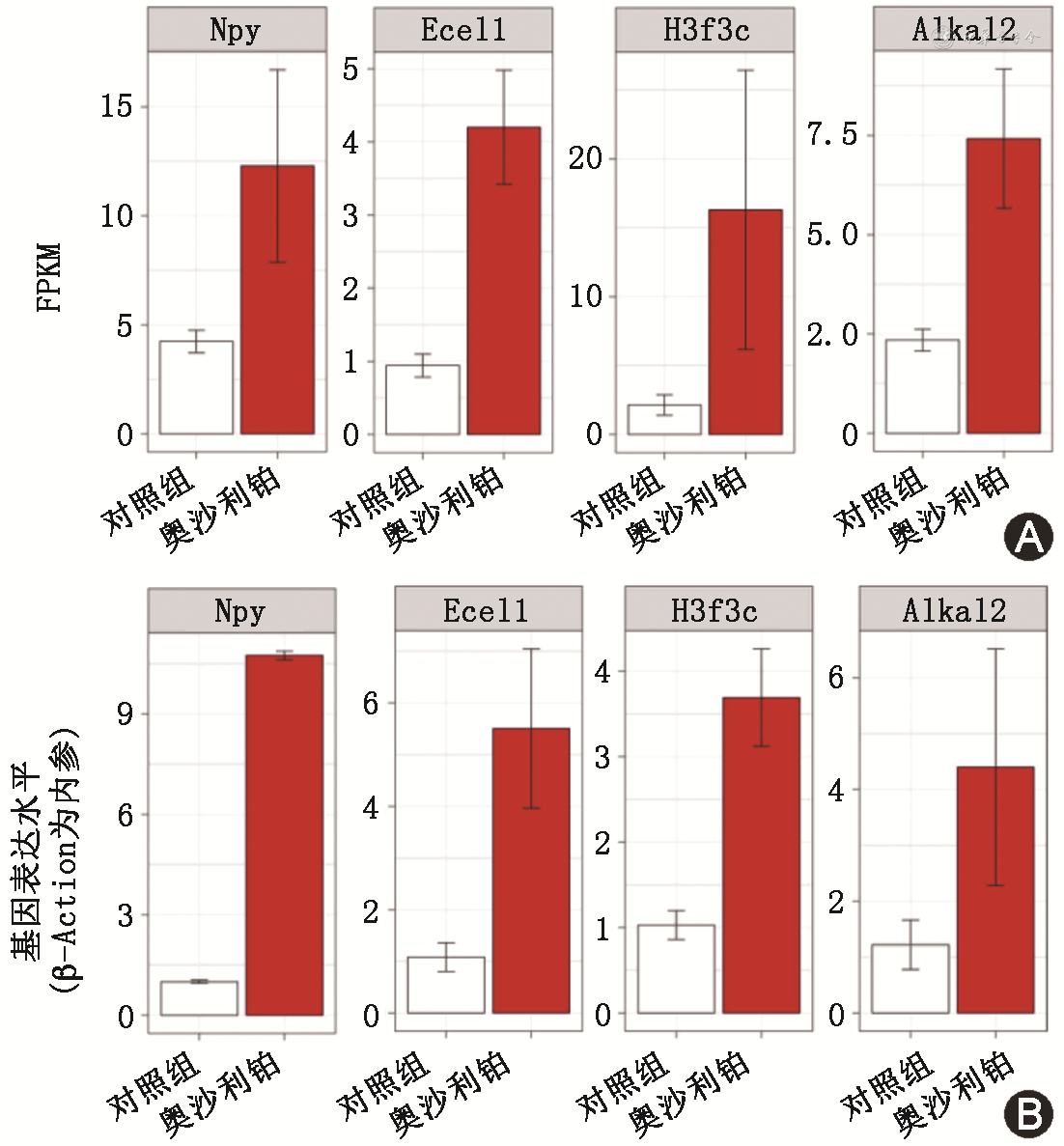
4. qPCR验证奥沙利铂诱导的差异表达基因:为了验证转录组测序定量的准确性,本研究选取显著受奥沙利铂影响的差异表达基因进行qPCR验证(图4)。qPCR技术与转录组测序技术对各组中Npy、Ecel1、H3f3c、Alkal2的定量结果基本一致,表明转录组测序结果可靠。

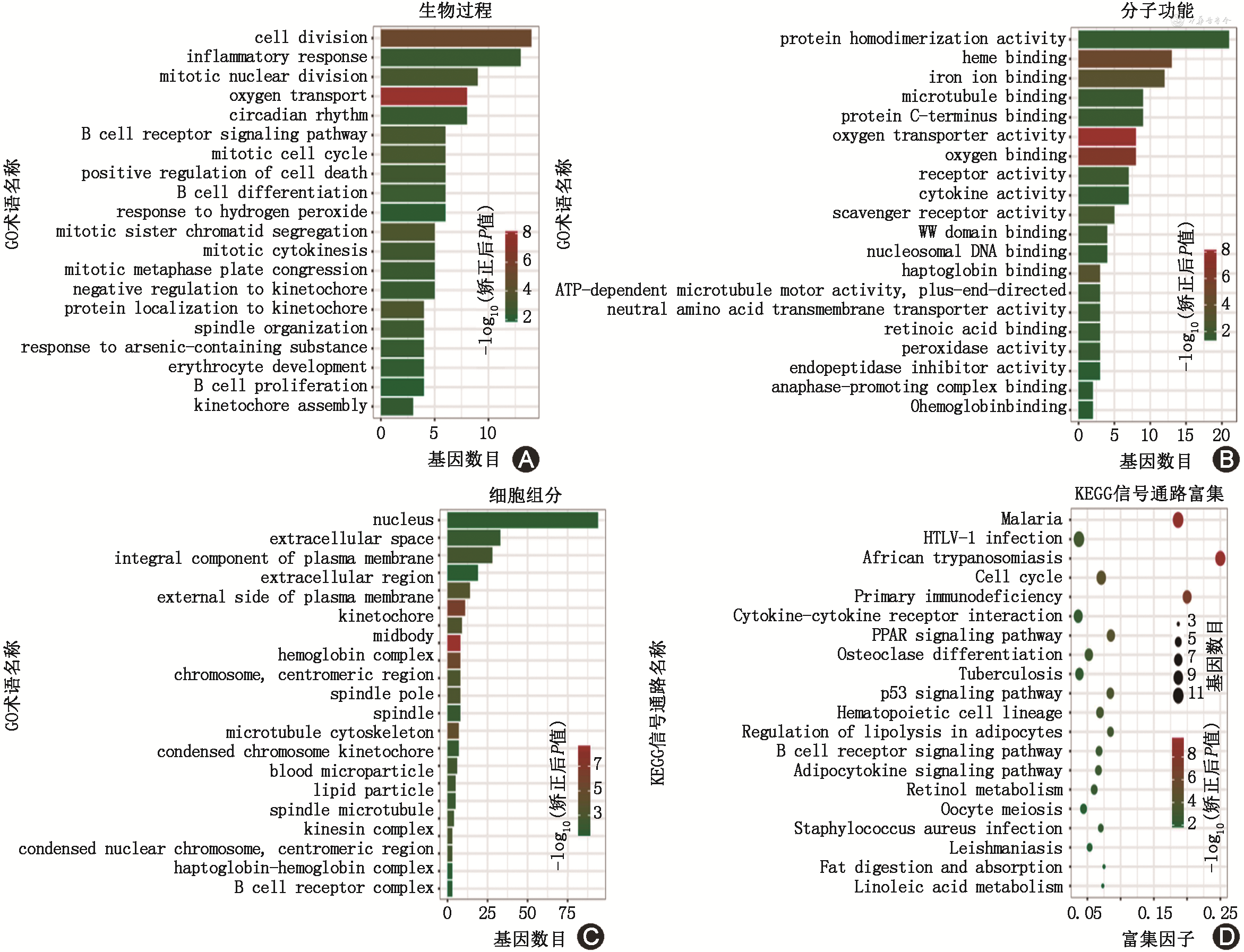
5. 奥沙利铂诱导外周病理性痛大鼠模型的差异表达基因功能分析:为了进一步了解奥沙利铂诱导的差异表达基因相关功能,对差异表达基因进行GO功能富集分析。图5展示了GO分析富集到20个最显著的GO 功能以及差异基因个数分布情况。受奥沙利铂影响的差异表达基因参与的生物学过程主要有:氧运输、细胞分裂(图5A);细胞组成主要有中间体、着丝粒(图5B);分子功能主要有:氧转运蛋白活性、氧结合(图5C)。与此同时,对差异表达基因进行KEGG信号通路富集分析(图5D),受奥沙利铂影响的差异表达基因参与的KEGG信号通路主要有:疟疾、非洲锥虫病、原发性免疫缺陷、过氧化物酶体增殖物激活受体信号通路。通过对受奥沙利铂影响差异表达基因进行GO功能与KEGG信号通路富集分析,提示奥沙利铂给药15 d后,大鼠DRG组织氧运输、氧转运蛋白活性相关基因的表达受到奥沙利铂干扰,并且奥沙利铂可引起类似疟疾、非洲锥虫病等疾病的信号通路反应。

化疗引起的周围神经性疼痛是化疗过程中化疗药最常见的严重不良反应。奥沙利铂广泛应用于胃肠道肿瘤和其他恶性肿瘤的治疗,但奥沙利铂诱导的CIPNP严重影响患者的生活质量限制了奥沙利铂在恶性肿瘤治疗中的应用。然而目前,对奥沙利铂引起的的周围神经性疼痛的分子机制尚不清楚,并且尚无关于奥沙利铂与周围神经性疼痛在转录水平上的研究。
为了弥补该空缺,本研究通过连续5 d奥沙利铂给药,大鼠在第7天开始出现机械痛觉异常和冷热痛觉过敏,在14天达到最强,表明奥沙利铂诱导的周围神经性疼痛模型建立成功。在第15天收集大鼠DRG,通过转录组测序,总共定量到20 152个基因。通过差异基因筛选,获得379个奥沙利铂诱导的差异表达基因。为了分析奥沙利铂诱导周围神经性疼痛的可能分子机制,结合已经公开发表的SNI小鼠模型DRG基因芯片表达数据进行分析发现,奥沙利铂可诱导Npy,Car3,Cdkn1a,Nts,Prc1,Ms4a7和Ecel1 7个的外周神经损伤疼痛相关基因的表达。Npy是一种由36个氨基酸组成的神经肽,Npy系统是疼痛调节的关键元件。大鼠DRG中Npy的上调参与了神经病理性疼痛的发病机制[13];另外,Cdkn1a[14]、Nts[15]也在周围神经损伤中发挥重要作用。
在生物体内,不同基因相互协调行使其生物学功能,GO数据库可分为三个模块:生物过程、细胞组成和分子功能,收了大约45 000个基因功能注释,常被研究者用于差异基因的功能分析[16]。对铂诱导的差异表达基因进行GO富集分析发现铂诱导的差异表达基因显著参与氧运输、细胞分裂、中间体、着丝粒、氧转运蛋白活性和氧结合等GO功能。KEGG是基因参与信号通路的知识库,不仅被广泛用于分析基因组学数据,还被用于转录组学、蛋白质组学、糖组学、代谢组学、宏基因组学和其他高通量数据的分析[17]。通过KEGG信号通路显著性富集分析发现铂诱导的差异表达基因显著参与疟疾、非洲锥虫病、原发性免疫缺陷、过氧化物酶体增殖物激活受体信号通路等KEGG信号通路。过氧化物酶体增殖物激活受体(PPARs)是配体激活的转录因子,属于核激素受体超家族,包含三种亚型(α、β/δ和γ)[18]。PPAR配体通过一种称为配体依赖性直接反式抑制的分子机制抑制参与两种疼痛的炎症介质(如促炎细胞因子)的基因表达[19]。有研究发现,给予PPAR配体可以减轻炎症性疼痛和神经性疼痛[20]。PPAR配体在动物模型中对神经炎症的影响提示其可能用于治疗人类炎症性疼痛和神经性疼痛[18]。PPARs广泛分布在哺乳动物组织中,包括周围组织和中枢神经系统[21],最近的临床前研究表明,过氧化物酶体增殖物激活受体γ(PPARγ)的激活剂(格列酮)在神经性疼痛模型中具有较好的功效[22],这提示格列酮在奥沙利铂引起的外周神经损伤可能具有缓解作用。
总之,本研究采用连续给药奥沙利铂成功建立了大鼠CIPNP模型,通过RNA-seq技术对大鼠DRG进行基因转录组水平分析,发现奥沙利铂诱导CIPNP的分子机制可能与诱导Npy、Car3等相关基因及PPAR等信号通路有关。
所有作者均声明不存在利益冲突



























